1.所需要软件下载:
(1)libsvm(http://www.csie.ntu.edu.tw/~cjlin/libsvm/)
(2)python
(3)gnuplot 画图软件(ftp://ftp.gnuplot.info/pub/gnuplot/)
这里只考虑windows的环境:
1、 下载libsvm的zip包,只要解压到某个文件夹就好就好(随便D:\gjs\libsvm)

2、安装python(我的是2.7.3)
3、下载好gnuplot ,直接解压就好,无需安装(C:\gnuplot)
2.数据格式说明
0 1:5.1 2:3.5 3:1.4 4:0.2
2 1:4.9 2:3.0 3:1.4 4:0.2
1 1:4.7 2:3.2 3:1.3 4:0.2
[label] [Index1]:[value1] [index2]:[value2] [index3]:[value3]
[label]:类别(通常是整数)[index n]: 有顺序的索引 [value n]
可能需要自己转换训练以及测试数据的格式。
3.使用方法
1. windows cmd命令窗口
下载的libsvm包里面已经为我们编译好了(windows)。
进入libsvm\windows,可以看到这几个exe文件:
1.svm-predict: svmpredict test_file mode_file output_file 依照已经train好的model ,输入新的数据,并输出预测新数据的类别。
2.svm-scale: 有时候特征值的波动范围比较大需要对特征数据进行缩放,可以缩放到0--1之间(自己定义)。
3.svm-toy:似乎是图形界面,可以自己画点,产生数据等。
4.svm-train: svmtrain [option] train_file [model_file] train 会接受特定格式的输入,产生一个model 文件。
第一步:可以自己生成数据,使用svm-toy:
双击svm-toy,点击change可以在画布上画点:

点击run,其实就是train的过程,划分的区域:

点击save可以保存数据(假设保存的数据在D://libsvm.txt)。
第二步:使用训练数据libsvm.txt进行建模,使用svm-train:
使用cmd命令进入到我们解压的libsvm目录中的windows目录,使用svm-train,如下:

其中,
#iter为迭代次数,
nu 是你选择的核函数类型的参数,
obj为SVM文件转换为的二次规划求解得到的最小值,
rho为判决函数的偏置项b,
nSV 为标准支持向量个数(0<a[i]<c),
nBSV为边界上的支持向量个数(a[i]=c),
Total nSV为支持向量总个数(对于两类来说,因为只有一个分类模型Total nSV = nSV,但是对于多类,这个是各个分类模型的nSV之和
同时在该目录下会生成一个训练好的model(libsvm.txt.model)可以打开文件查看里面的内容,主要包括一些参数和支持向量等
第三步:使用建好的model进行预测,使用svm-predict
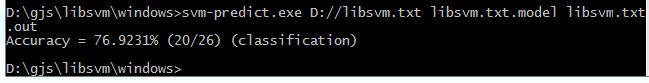
同时会生成一个输出文件(libsvm.txt.out),每行代表该行的预测值类别。
参数优化:
svm的参数优化很重要,libsvm包里面包含了参数的优化函数,主要是暴力求解参数。一般来说我们会使用高斯核函数,包含两个参数(c 和 g)
使用gird.py文件进行参数优化选择:
grid.py在libsvm/tools里面,首先需要修改gird.py中的gnuplot文件路径问题,把文件里的路径改成gnuplot 存放的目录:
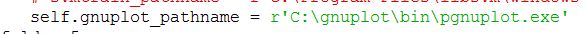
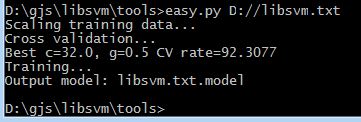
进入grid.py的相应目录,执行grid.py D://libsvm.txt
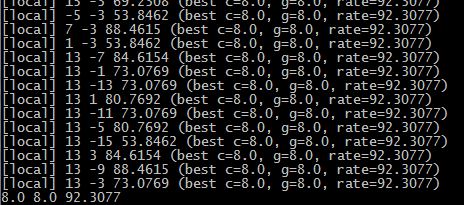
前面两个分别是c 跟g的值,这时候我们重新训练模型(加上参数c g)
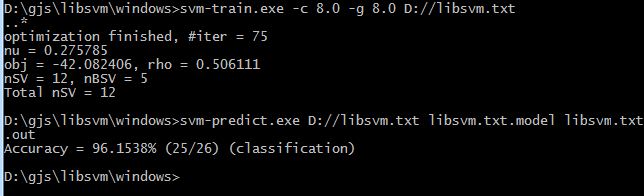
可以看到,准确率有了显著的提升, 其实这些步骤完全可以使用easy.py进行实现,同理也需要修改eays.py里面的gnuplot文件路径问题,把文件里的路径改成gnuplot 存放的目录:
步骤总结如下:
1.转换训练数据为相应的格式。
2.有时候可能需要使用 svm-scale对数据进行相应的缩放,有利于训练建模。

3.使用grid.py或者easy.py进行参数优化。
4.使用svm-train建模和svm-predict进行预测。
2.python版本 使用:
>>> import os >>> os.chdir('D://gjs//libsvm//python') >>> from svmutil import * >>> y,x=svm_read_problem("D://libsvm.txt") >>> m=svm_train(y,x,'-c 8.0 -g 8.0') >>> p_lable,p_acc,p_val=svm_predict(y,x,m) Accuracy = 96.1538% (25/26) (classification) >>>
>>> import os >>> os.chdir('D://gjs//libsvm//python') >>> from svmutil import* >>> data=svm_problem([1,-1],[[1,0,1],[-1,0,-1]]) #元组一表示分类类别 >>> param=svm_parameter('-c 8.0 -g 8.0') >>> model=svm_train(data,param) >>> svm_predict([1],[1,1,1],model) >>>svm_predict([1,-1],[[1,-1,-1],[1,1,1]],model) Accuracy = 0% (0/2) (classification) ([-1.0, 1.0], (0.0, 4.0, 1.0), [[0.0], [0.00033546262790251185]])
3.weka中使用libSVM:
可以参照: http://datamining.xmu.edu.cn/~gjs/project/LibD3C.html
4.eclipse中调用libsvm:
http://datamining.xmu.edu.cn/~gjs/download/LibSVM.jar
http://datamining.xmu.edu.cn/~gjs/download/libsvm.jar
下载以上两个包libsvm的包,然后在eclipse工程目录里面添加相应的jar包:

DataSource source = new DataSource("D://iris.arff"); Classifier clas=new LibSVM(); String[] optSVM = weka.core.Utils.splitOptions("-c 8.0 -g 8.0"); ((LibSVM) clas).setOptions(optSVM); Instances data=source.getDataSet(); data.setClassIndex(data.numAttributes()-1); Evaluation eval=new Evaluation(data); eval.crossValidateModel(clas, data, 10, new Random(1)); System.out.println(eval.toClassDetailsString()); System.out.println(eval.toSummaryString()); System.out.println(eval.toMatrixString());
输出结果为:

5. linux下使用libsvm:
确认已经安装好python
1. wget http://www.csie.ntu.edu.tw/~cjlin/cgi-bin/libsvm.cgi?+http://www.csie.ntu.edu.tw/~cjlin/libsvm+tar.gz。
2. tar -zxvf /home/gjs/libsvm.tar.gz。
3. 进入目录执行 make 编译。
4. ./svm-train /home/gjs/libsvm.txt 其他也类似。
5. python grid.py /home/gjs/libsvm.txt 优化参数。


 浙公网安备 33010602011771号
浙公网安备 33010602011771号