实验室最近用到nmi( Normalized Mutual information )评价聚类效果,在网上找了一下这个算法的实现,发现满意的不多.
浙江大学蔡登教授有一个,http://www.zjucadcg.cn/dengcai/Data/code/MutualInfo.m ,他在数据挖掘届地位很高,他实现这个算法的那篇论文引用率高达三位数。但这个实现,恕个人能力有限,我实在是没有看懂:变量命名极为个性,看的如坠云雾;代码倒数第二行作者自己添加注释why complex,我就更不懂了;最要命的是使用他的函数MutualInfo(L1,L2)得到的结果不等于MutualInfo(L2,L1),没有对称性!
还有个python的版本http://blog.sun.tc/2010/10/mutual-informationmi-and-normalized-mutual-informationnmi-for-numpy.html,这个感觉很靠谱,作者对nmi的理解和我是一样的。
我的理解来自wiki和stanford,其实很简单,先说一下问题:例如stanford中介绍的一个例子:

比如标准结果是图中的叉叉点点圈圈,我的聚类结果是图中标注的三个圈。
或者我的结果: A = [1 1 1 1 1 1 2 2 2 2 2 2 3 3 3 3 3];
标准的结果 : B = [1 2 1 1 1 1 1 2 2 2 2 3 1 1 3 3 3];
问题:衡量我的结果和标准结果有多大的区别,若我的结果和他的差不多,结果应该为1,若我做出来的结果很差,结果应趋近于0。
MI可以按照下面的公式计算。X=unique(A)=[1 2 3],Y=unique(B)=[1 2 3];
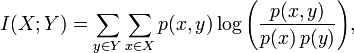
分子p(x,y)为x和y的联合分布概率,
p(1,1)=5/17, p(1,2)=1/17, p(1,3)=0;
p(2,1)=1/17, p(2,2)=4/17, p(2,3)=1/17;
p(3,1)=2/17, p(3,2)=0, p(3,3)=3/17;
分母p(x)为x的概率函数,p(y)为y的概率函数,x和y分别来自于A和B中的分布,所以即使x=y时,p(x)和p(y)也可能是不一样的。
对p(x): p(1)=6/17 p(2)=6/17 p(3)=5/17
对p(y): p(1)=8/17 p(2)=5/17 P(3)=4/17
这样就可以算出MI值了。
标准化互聚类信息也很简单,有几个不同的版本,大体思想都是相同的,都是用熵做分母将MI值调整到0与1之间。一个比较多见的实现是下面所示:
![]()
H(X)和H(Y)分别为X和Y的熵,下面的公式中log的底b=2。
![]()
例如H(X) = -p(1)*log2(p(1)) - -p(2)*log2(p(2)) -p(3)*log2(p(3))。
matlab实现代码如下
%NMI Normalized mutual information
% http://en.wikipedia.org/wiki/Mutual_information
% http://nlp.stanford.edu/IR-book/html/htmledition/evaluation-of-clustering-1.html
% Author: http://www.cnblogs.com/ziqiao/ [2011/12/13]
if length( A ) ~= length( B)
error('length( A ) must == length( B)');
end
total = length(A);
A_ids = unique(A);
B_ids = unique(B);
% Mutual information
MI = 0;
for idA = A_ids
for idB = B_ids
idAOccur = find( A == idA );
idBOccur = find( B == idB );
idABOccur = intersect(idAOccur,idBOccur);
px = length(idAOccur)/total;
py = length(idBOccur)/total;
pxy = length(idABOccur)/total;
MI = MI + pxy*log2(pxy/(px*py)+eps); % eps : the smallest positive number
end
end
% Normalized Mutual information
Hx = 0; % Entropies
for idA = A_ids
idAOccurCount = length( find( A == idA ) );
Hx = Hx - (idAOccurCount/total) * log2(idAOccurCount/total + eps);
end
Hy = 0; % Entropies
for idB = B_ids
idBOccurCount = length( find( B == idB ) );
Hy = Hy - (idBOccurCount/total) * log2(idBOccurCount/total + eps);
end
MIhat = 2 * MI / (Hx+Hy);
end
% Example :
% (http://nlp.stanford.edu/IR-book/html/htmledition/evaluation-of-clustering-1.html)
% A = [1 1 1 1 1 1 2 2 2 2 2 2 3 3 3 3 3];
% B = [1 2 1 1 1 1 1 2 2 2 2 3 1 1 3 3 3];
% nmi(A,B)
% ans = 0.3646
为了节省运行时间,将for循环用矩阵运算代替,1百万的数据量运行 1.795723second,上面的方法运行3.491053 second;
但是这种方法太占内存空间, 五百万时,利用matlab2011版本的内存设置就显示Out of memory了。
%NMI Normalized mutual information
% http://en.wikipedia.org/wiki/Mutual_information
% http://nlp.stanford.edu/IR-book/html/htmledition/evaluation-of-clustering-1.html
% Author: http://www.cnblogs.com/ziqiao/ [2011/12/15]
if length( A ) ~= length( B)
error('length( A ) must == length( B)');
end
total = length(A);
A_ids = unique(A);
A_class = length(A_ids);
B_ids = unique(B);
B_class = length(B_ids);
% Mutual information
idAOccur = double (repmat( A, A_class, 1) == repmat( A_ids', 1, total ));
idBOccur = double (repmat( B, B_class, 1) == repmat( B_ids', 1, total ));
idABOccur = idAOccur * idBOccur';
Px = sum(idAOccur') / total;
Py = sum(idBOccur') / total;
Pxy = idABOccur / total;
MImatrix = Pxy .* log2(Pxy ./(Px' * Py)+eps);
MI = sum(MImatrix(:))
% Entropies
Hx = -sum(Px .* log2(Px + eps),2);
Hy = -sum(Py .* log2(Py + eps),2);
%Normalized Mutual information
MIhat = 2 * MI / (Hx+Hy);
% MIhat = MI / sqrt(Hx*Hy); another version of NMI
end
% Example :
% (http://nlp.stanford.edu/IR-book/html/htmledition/evaluation-of-clustering-1.html)
% A = [1 1 1 1 1 1 2 2 2 2 2 2 3 3 3 3 3];
% B = [1 2 1 1 1 1 1 2 2 2 2 3 1 1 3 3 3];
% nmi(A,B)
% ans = 0.3646
参考: stanford的讲解:http://nlp.stanford.edu/IR-book/html/htmledition/evaluation-of-clustering-1.html
wiki百科的讲解:http://en.wikipedia.org/wiki/Mutual_information
某作者的python的实现:http://blog.sun.tc/2010/10/mutual-informationmi-and-normalized-mutual-informationnmi-for-numpy.html
蔡登的matlab实现:http://www.zjucadcg.cn/dengcai/Data/code/MutualInfo.m




【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】凌霞软件回馈社区,博客园 & 1Panel & Halo 联合会员上线
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】博客园社区专享云产品让利特惠,阿里云新客6.5折上折
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步