【怪毛匠子整理】
一、使用SOFT文件
1. 确定GEO平台,比如GPL25318, 通过NCBI找到这个平台,也可以通过下面的链接,把GPL25318 替换成你想要的平台ID。
1 | https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL25318 |
2. 下载SOFT文件
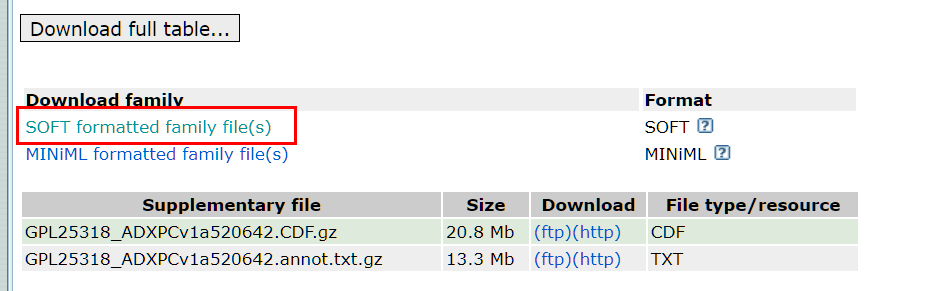
点开红框:
下载文件:

解压文件:
R语言处理# 读入文件
1 | df2=read.table("GPL25318_family.soft",sep = "\t")<br># 使用merge合并<br> |
【怪毛匠子整理】
二、使用GEOquery包
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 | library(GEOquery)GPL=getGEO("GPL25318",destdir = ".")g1=Table(GPL)head(exprSet$ID)[1] "200000_s_at" "200001_at" "200002_at" "200003_s_at" "200004_at" "200005_at"new_datasets=merge(exprSet,g1,by = "ID" ,all.x = T)colnames(new_datasets)[1] "ID" "GSM******" "GSM******".....[247] "GSM******" "GSM******" "GSM******"[250] "Ensembl Version" "Ensembl Genome Version" "Orientation"[253] "Probeset mapping position" "Ensembl Gene ID" "Gene Symbol"[256] "Chromosomal location" "Strand" "Gene Description"[259] "Entrez Gene" "SPOT_ID" |
这样就搞定了!
【怪毛匠子整理】




【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】凌霞软件回馈社区,博客园 & 1Panel & Halo 联合会员上线
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 深入理解 Mybatis 分库分表执行原理
· 如何打造一个高并发系统?
· .NET Core GC压缩(compact_phase)底层原理浅谈
· 现代计算机视觉入门之:什么是图片特征编码
· .NET 9 new features-C#13新的锁类型和语义
· Spring AI + Ollama 实现 deepseek-r1 的API服务和调用
· 《HelloGitHub》第 106 期
· 数据库服务器 SQL Server 版本升级公告
· 深入理解Mybatis分库分表执行原理
· 使用 Dify + LLM 构建精确任务处理应用