遗传算法GA(goat工具箱)
function [pop]=initializega(num,bounds,eevalFN,eevalOps,options)
输出参数:
pop 生成的初始种群
输入参数:
num 种群中个体数目
bounds 代表变量的上下界的矩阵
eevalFN 适应度函数
eevalOps 传递给适应度函数的参数
options 选择编码形式(浮点编码或二进制编码)
precision 变量进行二进制编码时指定的精度
function [x,endPop,bPop,traceInfo] = ga(bounds,evalFN,evalOps,startPop,opts,... termFN,termOps,selectFN,selectOps,xOverFNs,xOverOps,mutFNs,mutOps)
输出参数:
x 求得的最优解
endPop 最终得到的种群
bPop 最优种群的一个搜索轨迹
输入参数:
bounds 代表变量上下界的矩阵
evalFN 适应度函数
evalOps 传递给适应度函数的参数
startPop 初始种群
opts[epsilon prob_ops display] opts(1:2)等同于initializega的options参数,第三个参数控制是否输出,一般为0。
如[1e-6 1 0]
termFN 终止函数的名称
termOps 传递终止函数的参数
selectFN 选择函数的名称
selectOps 传递选择函数的参数
xOverFNs 交叉函数名称表
xOverOps 传递给交叉函数的参数表
mutFNs 变异函数表
1.求f(x)=x+10*sin(5x)+7*cos(4x)的最大值,其中0<=x<=9
选择二进制编码,种群中的个体数目为10,二进制编码长度为20,交叉概率为0.95,变异概率为0.08
function[sol,eval]=fitness(sol,options) x=sol(1); eval=x+10*sin(5*x)+7*cos(4*x); end
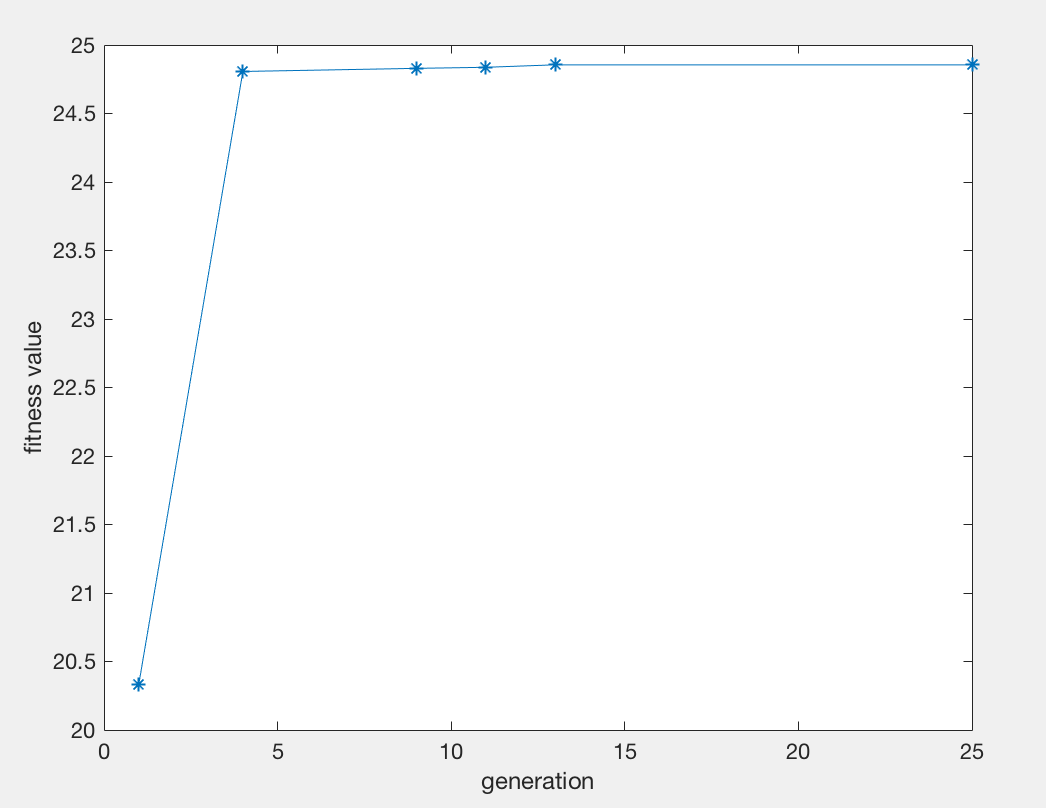
>> initPop=initializega(10,[0 10],'fitness'); [x endPop,bPop,trace]=ga([0 10],'fitness',[],initPop,... [1e-6 1 1],'maxGenTerm',25,'normGeomSelect',... [0.08],['arithXover'],[2],'nonUnifMutation',[2 25 3]);
>> bPop
bPop =
1.0000 9.3320 20.3311
4.0000 7.8404 24.8068
9.0000 7.8448 24.8296
11.0000 7.8467 24.8373
13.0000 7.8565 24.8554
25.0000 7.8565 24.8554
>> x
x =
7.8565 24.8554
当x为7.8565时,f(x)取最大值24.8554
sol它的每行代表种群中的一个个体,假如有n个变量,那么每行的前n列就代表每个变量的值,如x1,x2....,xn,最后一列代表的是这些变量的适应值,也就是eval。由于intializega函数执行时,要调用fitness函数,并且传递给fitness函数一个个体的信息,该个体信息是1行n+1列的向量,所以sol得到的值实际是1行n+1列的向量,但是计算适应值只有用到前n个信息就行了,即用到(x1,x2....,xn)的值,因此fitness调用sol(1:n).
一般情况下x只是单变量,即n=1,所以就会出现调用sol(1)了
假设有多个自变量
function [sol,eval]=fitness(sol,options) numvar=size(sol,2)-1;%求出自变量个数 假设有多个自变量 x=sol(1:numvar); eval=-(sum(x.^2));%适应度函数 end



