总结U-Net网络及他的变体
今天来简单归纳一下,医学图像分割的内容,还有很多代表性的工作在之后的文章中不断更新,由于同一个网络结构可能在不同的数据集上表现出不一样的性能,在具体的任务场景中还是要结合数据集来选择合适的网络。
医学图像特点
1.图像语义较为简单、结构较为固定。我们做脑的,就用脑CT和脑MRI,做胸片的只用胸片CT,做眼底的只用眼底OCT,都是一个固定的器官的成像,而不是全身的。由于器官本身结构固定和语义信息没有特别丰富,所以高级语义信息和低级特征都显得很重要(UNet的skip connection和U型结构就派上了用场)。举两个例子直观感受下。
2.数据量少。医学影像的数据获取相对难一些,很多比赛只提供不到100例数据。所以我们设计的模型不宜多大,参数过多,很容易导致过拟合。
原始UNet的参数量在28M左右(上采样带转置卷积的UNet参数量在31M左右),而如果把channel数成倍缩小,模型可以更小。缩小两倍后,UNet参数量在7.75M。缩小四倍,可以把模型参数量缩小至2M以内,非常轻量。个人尝试过使用Deeplab v3+和DRN等自然图像语义分割的SOTA网络在自己的项目上,发现效果和UNet差不多,但是参数量会大很多。
3.多模态。相比自然影像,医疗影像比较有趣和不同的一点是,医疗影像是具有多种模态的。以ISLES脑梗竞赛为例,其官方提供了CBF,MTT,CBV,TMAX,CTP等多种模态的数据。
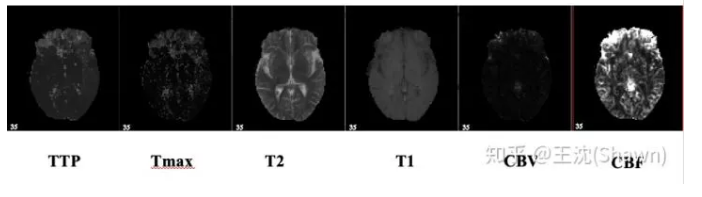
这就需要我们更好的设计网络去提取不同模态的特征feature。这里提供两篇论文供大家参考。
Joint Sequence Learning and Cross-Modality Convolution for 3D Biomedical Segmentation(CVPR 2017) ,
Dense Multi-path U-Net for Ischemic Stroke Lesion Segmentation in Multiple Image Modalities.
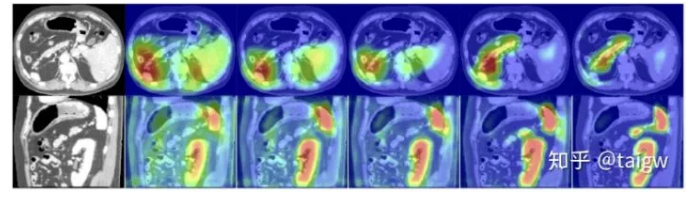
4.可解释性重要。由于医疗影像最终是辅助医生的临床诊断,所以网络告诉医生一个3D的CT有没有病是远远不够的,医生还要进一步的想知道,病灶在哪一层,在哪一层的哪个位置,分割出来了吗,能求体积嘛?同时对于网络给出的分类和分割等结果,医生还想知道为什么,所以一些神经网络可解释性的trick就有用处了,比较常用的就是画activation map。看网络的哪些区域被激活了,如下图。
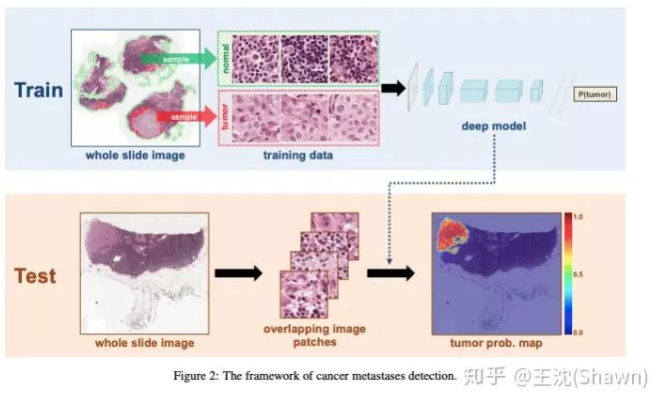
这里推荐两篇工作:周博磊老师的Learning Deep Features for Discriminative Localization(CVPR2016)和其实验室同学的 Deep Learning for Identifying Metastatic Breast Cancer(上图的出处)
(周博磊知乎主页:https://www.zhihu.com/people/zhou-bo-lei/activities)
U-Net
论文地址:https://lmb.informatik.uni-freiburg.de/people/ronneber/u-net/
代码地址:https://github.com/wolny/pytorch-3dunet
自2015年以来,在生物医学图像分割领域,U-Net得到了广泛的应用,该方法在2015年MICCAI会议上提出,目前已达到四千多次引用。至今,U-Net已经有了很多变体,目前已有许多新的卷积神经网络设计方式,但很多仍延续了U-Net的核心思想,加入了新的模块或者融入其他设计理念。U-Net架构在不同的生物医学分割应用中实现了非常好的性能。由于借助具有弹性形变的数据增强功能,它只需要少量的的带标注的图像,并且在NVidia Titan GPU(6 GB)上仅需要10个小时的训练时间。
U-Net如下图所示,是一个encoder-decoder结构,左边一半的encoder包括若干卷积,池化,把图像进行下采样,右边的decoder进行上采样,恢复到原图的形状,给出每个像素的预测。
具体来说,左侧可视为一个编码器,右侧可视为一个解码器。编码器有四个子模块,每个子模块包含两个卷积层,每个子模块之后有一个通过max pool实现的下采样层。输入图像的分辨率是572x572, 第1-5个模块的分辨率分别是572x572, 284x284, 140x140, 68x68和32x32。由于卷积使用的是valid模式,故这里后一个子模块的分辨率等于(前一个子模块的分辨率-4)/2。解码器包含四个子模块,分辨率通过上采样操作依次上升,直到与输入图像的分辨率一致(由于卷积使用的是valid模式,实际输出比输入图像小一些)。该网络还使用了跳跃连接,将上采样结果与编码器中具有相同分辨率的子模块的输出进行连接,作为解码器中下一个子模块的输入。

架构中的一个重要修改部分是在上采样中还有大量的特征通道,这些通道允许网络将上下文信息传播到具有更高分辨率的层。因此,拓展路径或多或少地与收缩路径对称,并产生一个U形结构。
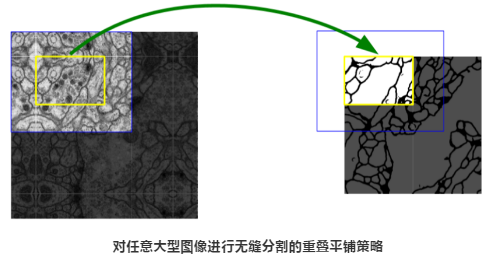
在该网络中没有任何完全连接的层,并且仅使用每个卷积的有效部分,即分割映射仅包含在输入图像中可获得完整上下文的像素。该策略允许通过重叠平铺策略对任意大小的图像进行无缝分割,如图所示。为了预测图像边界区域中的像素,通过镜像输入图像来推断缺失的上下文。这种平铺策略对于将网络应用于大型的图像非常重要,否则分辨率将受到GPU内存的限制。
对于可用训练数据非常少的情况,可以通过对可用的训练图像应用弹性变形来进行数据增强。这使得网络学习这种变形的不变性,而不需要在标注图像语料库中看到这些变形。这在生物医学分割中尤其重要,因为变形曾是组织中最常见的变化,并且可以有效地模拟真实的变形。
许多细胞分割任务中的另一个挑战是分离同一类的接触目标。为此建议使用加权损失,其中在接触单元之间分开的背景标签在损失函数中获得较大的权重。
相比于FCN和Deeplab等,UNet共进行了4次上采样,并在同一个stage使用了skip connection,而不是直接在高级语义特征上进行监督和loss反传,这样就保证了最后恢复出来的特征图融合了更多的low-level的feature,也使得不同scale的feature得到了的融合,从而可以进行多尺度预测和DeepSupervision。4次上采样也使得分割图恢复边缘等信息更加精细。
3D U-Net
论文地址:https://arxiv.org/pdf/1606.06650.pdf
代码地址:https://github.com/wolny/pytorch-3dunet
3D U-Net是U-Net的一个简单扩展,应用于三维图像分割,结构如下图所示。相比于U-Net,该网络仅用了三次下采样操作,在每个卷积层后使用了batch normalization,但3D U-Net和U-Net均没有使用dropout。
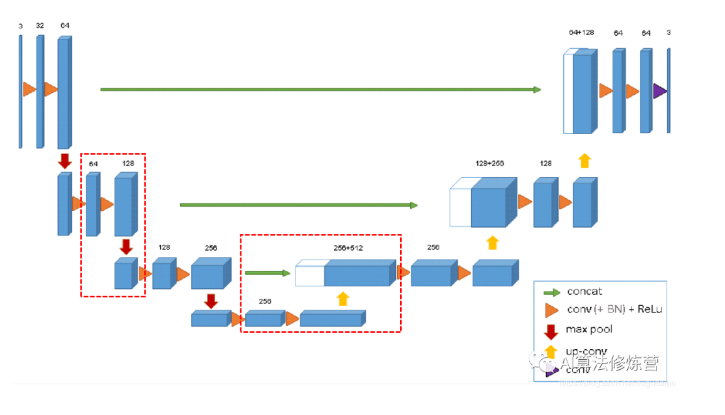
为了避免瓶颈,在上采样和下采样之前都将通道数增加为原来的二倍。左侧的红色虚线方框之内,在进行 maxpooling 之前将通道数从 64 变为了 128,及文中所说的 double;右侧的红色虚线内,在进行转置卷积之前,将通道数从 256 变为了 512。实际上这种思想来源于 inception V3。使用加权的 softmax 损失函数,将没有标签的部分置为 0,让模型只从有有标签的部分学习。
在2018年MICCAI脑肿瘤分割挑战赛(brats)中,德国癌症研究中心的团队使用3D U-Net,仅做了少量的改动,取得了该挑战赛第二名的成绩,发现相比于许多新的网络,3D U-Net仍然十分具有优势。
Res-UNet 和Dense U-Net
Res-UNet和Dense-UNet分别受到残差连接和密集连接的启发,将UNet的每一个子模块分别替换为具有残差连接和密集连接的形式。
将Res-UNet用于视网膜图像的分割,其结构如下图所示,其中灰色实线表示各个模块中添加的残差连接。
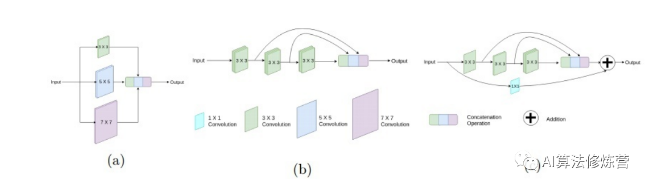
密集连接即将子模块中某一层的输出分别作为后续若干层的输入的一部分,某一层的输入则来自前面若干层的输出的组合。
下图是密集连接的一个例子。该文章中将U-Net的各个子模块替换为这样的密集连接模块,提出Fully Dense UNet 用于去除图像中的伪影。

MultiResUNet
论文地址:https://arxiv.org/abs/1902.04049.pdf
代码地址:https://github.com/nibtehaz/MultiResUNet
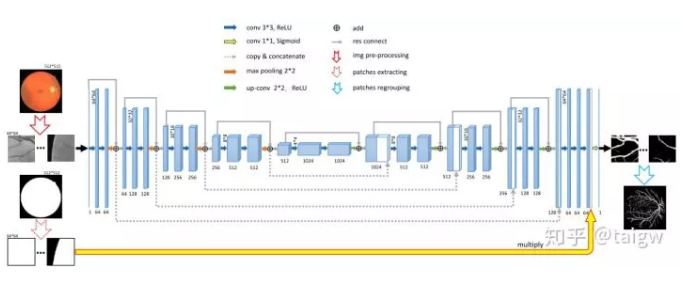
MultiResUNet提出了一个MutiRes模块与UNet结合。MutiRes模块如下图(c)所示,是一个残差连接的扩展,在该模块中三个3x3的卷积结果拼接起来作为一个组合的特征图,再与输入特征图经过1x1卷积得到的结果相加。
该网络的结构图如下图所示,其中各个MultiRes模块的内部即为上图所示。
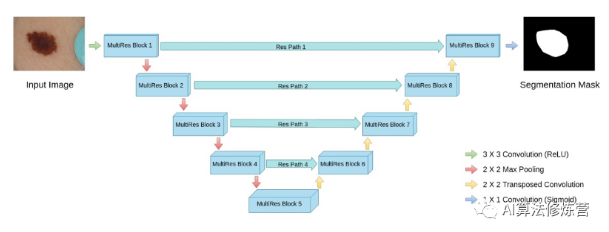
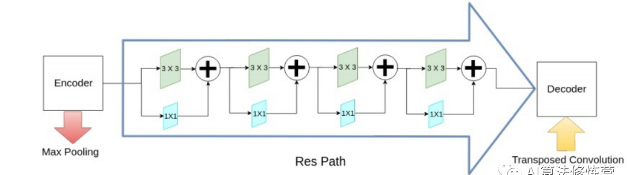
该网络除了MultiRes模块以外,还提出了一个残差路径(ResPath), 使编码器的特征在与解码器中的对应特征拼接之前,先进行了一些额外的卷积操作,如下图所示。作者认为编码器中的特征由于卷积层数较浅,是低层次的特征,而解码器中对应的特征由于卷积层更深,是较高层次的特征,二者在语义上有较大差距,推测不宜直接将二者进行拼接。因此,使用额外的ResPath使二者在拼接前具有一致的深度,在ResPath1, 2, 3, 4中分别使用4,3,2,1个卷积层。
该文章在ISIC、CVC-ClinicDB、Brats等多个数据集上验证了其性能。
Attention UNet
论文地址:https://arxiv.org/pdf/1804.03999.pdf
代码地址:
https://github.com/ozan-oktay/Attention-Gated-Networks
Attention UNet在UNet中引入注意力机制,在对编码器每个分辨率上的特征与解码器中对应特征进行拼接之前,使用了一个注意力模块,重新调整了编码器的输出特征。该模块生成一个门控信号,用来控制不同空间位置处特征的重要性,如下图中红色圆圈所示。
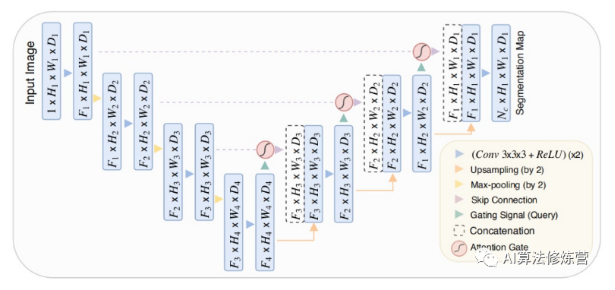
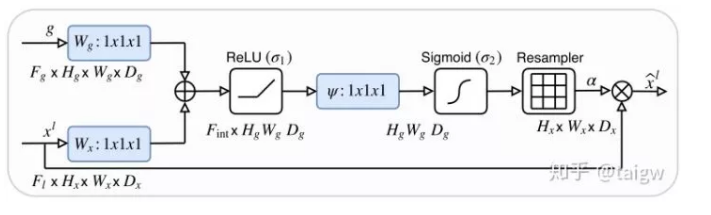
该方法的注意力模块内部如下图所示,该模块通过1x1x1的卷积分别与ReLU和Sigmoid结合,生成一个权重图α, 通过与编码器中的特征相乘来对其进行校正。
下图展示了注意力权重图的可视化效果。从左至右分别是一幅图像和随着训练次数的增加该图像中得到的注意力权重。可见得到的注意力权重倾向于在目标器官区域取得大的值,在背景区域取得较小的值,有助于提高图像分割的精度。
Non-local U-Nets for Biomedical Image Segmentation(AAAI2020)
论文地址:https://arxiv.org/abs/1812.04103
代码地址:https://github.com/Whu-wxy/Non-local-U-Nets-2D-block
global aggregation block
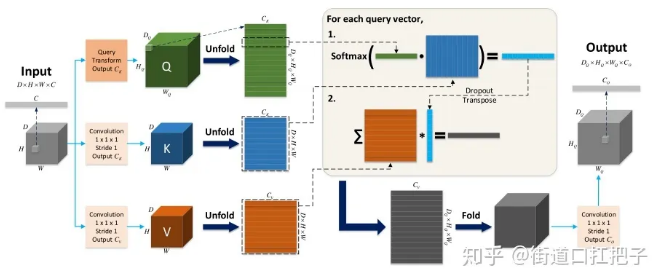
global aggregation block如上图所示,图看起来很复杂,实际上运算过程很简单。
1.该结构与Attention Is All You Need这篇文章的形式很相似。输入图为X(B*H*W*C),经过QueryTransform和1*1卷积,转换为Q(B*Hq*Wq*Ck),K(B*H*W*Ck),V(B*H*W*Cv)。QueryTransform可以为卷积,反卷积,插值等你喜欢的方法,最后的输出结果的H与W将与这个值一致。
2.代码里在Unfold之前有Multi-Head(Attention Is All You Need)的操作,不过在论文中没有说明,实际上是把通道分为N等份。Unfold是把Batch,height,width,N通道合并,Q(B*Hq*Wq*N*ck),K(B*H*W*N*ck),V(B*H*W*N*cv)。

3.接下来是经典的点积attention操作,得到一个权值矩阵A((B*Hq*Wq*N)*(B*H*W*N)),用于self-attention的信息加权,分母Ck是通道数,作用是调节矩阵的数值不要过大,使训练更稳定(这个也是Attention Is All You Need提出的)。最后权值矩阵A和V点乘,得到最终的结果((B*Hq*Wq*N)*cv),可见输出的height和width由Q决定,通道数由V决定。

4.我最近看了两篇上采样的论文,DUpsample和CARAFE,现在很多上采样相关的论文关注于在上采样时用CNN扩大感受野,增加图像局部信息。这篇文章提出的global aggregation block是一个将注意力机制和上/下采样相结合的方法,关注全图信息,感受野更大,可以在其他任务上试用一下,效果如何还是要看实践的结果。
Non-local U-Nets
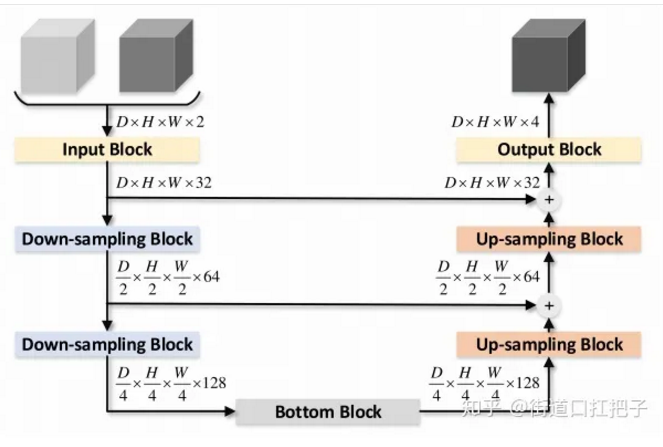
文章提出的Non-local U-Nets如上图所示。
相比U-Net,卷积层数减少,图像下采样倍率从16倍变成4倍,保留了更多空间信息。encoder和decoder之间的skip connections用相加的方式,不是拼接的方式,让模型推理速度更快。
上下采样使用global aggregation block,使分割图更准确。
参考:
[1] Section for Biomedical Image Analysis (SBIA)
( https://www.med.upenn.edu/sbia/brats2018/data.html )
[2] https://zhuanlan.zhihu.com/p/57530767
[3] https://zhuanlan.zhihu.com/p/57237535
[4]https://www.zhihu.com/question/269914775



