矩阵相似性分析
0、简介
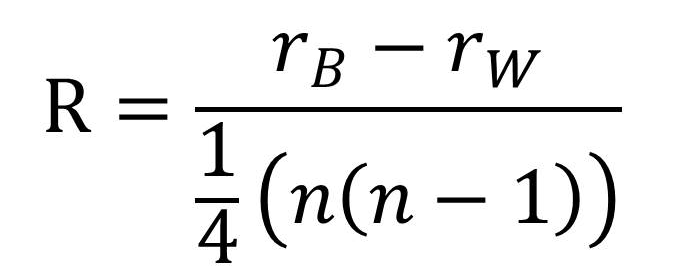
|
1
2
3
4
5
|
data(dune)<br>data(dune.env)dune.dist <- vegdist(dune)dune.ano <- with(dune.env, anosim(dune.dist, Management))summary(dune.ano)plot(dune.ano) |
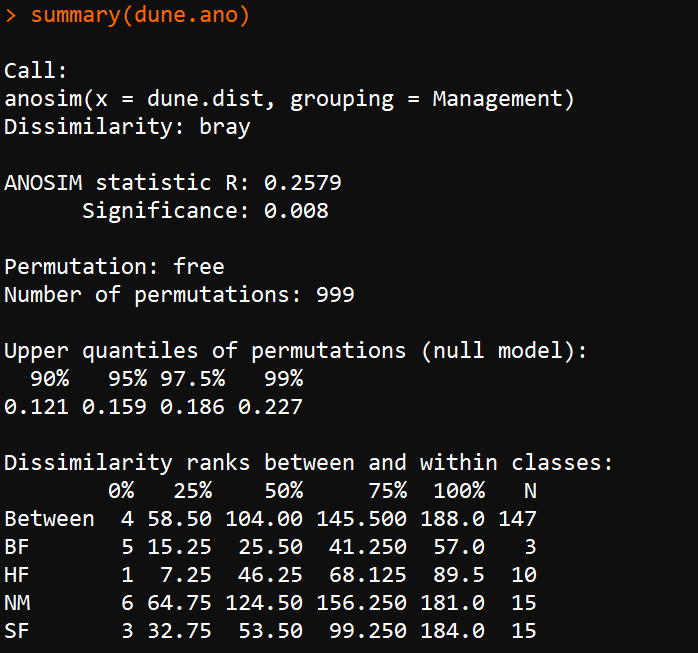
summary(dune.ano)结果为:
plot画出的图形为:
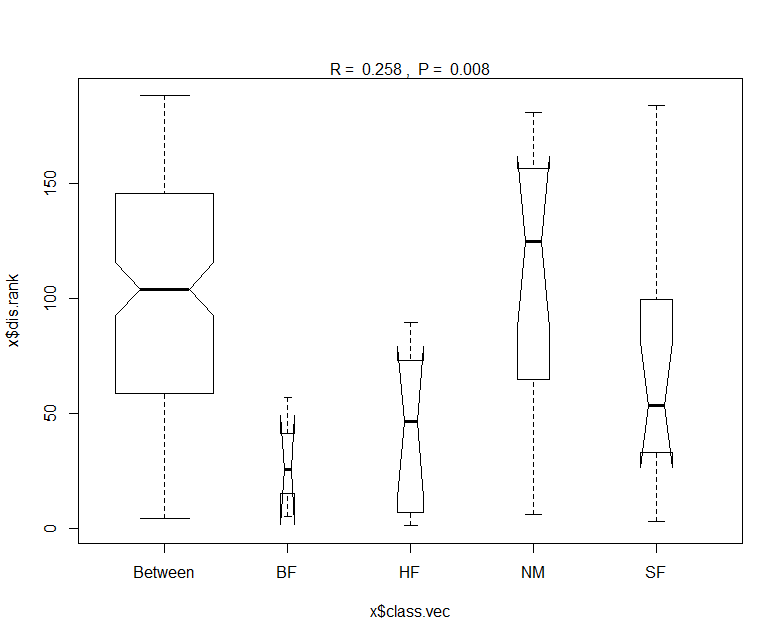
不难看出,图中箱子为summary结果中的Dissimilarity ranks between and within classes的可视化,即组间差异秩分布和组内差异秩分布(下图中的数值分布)。
一、beta多样性简介
Beta多样性作为群落结构研究的根基, 常用来比较不同生态系统之间的差异,反映生物种类因环境所造成异质性。通俗来讲,不同的处理(环境、健康状态等),会导致群落结构产生变化。往小了说,也就是物种分布情况的改变,一般通过指示物种分析,定位具体的差异物种,就能阐明驱动群落变化的主要物种因素;往大了说,还可以研究处理效应对样本乃至分组的影响,这个时候Beta多样性就发挥了其作用!
指示Beta的方法有很多,在文章中常见的有-距离指数(Jaccard、Bray-Curtis、Unifrac);排序分析(PCA、PCoA、NMDS);聚类分析(UPGMA);差异分析(adonis、anosim)等几大类。通过这些分析如PCA、PCoA等,能在实验前期帮助规避很多风险,例如,揭示分组是否符合预期,采样是否合理,还可对离群样本进行剔除等。
无论聚类还是排序分析,从分枝或散点距离,例如图1[1],能明显看出三个分组有差异,这种通过人眼的差异结果带有强烈的主观性,缺乏证据,也就是常说的统计学数据,会受到学术质疑。
因此今天给大家介绍两种组间差异的检验方法--ANOSIM、ADONIS。相较于普通的统计检验,像两组比较的Wilcoxon秩和检验、多组kw秩和检验等等。ANOSIM、ADONIS不但能输出显著性p值,来判定比较组之间是否存在差异,还可以额外给出差异的程度(R值),来揭示不同因素对群落变异的贡献。废话不多说进入正题!
ANOSIM、 ADONIS都是基于相似性(距离矩阵)的非参数检验,由于不要求数据正态分布和方差齐性,非常适用于微生物群落这种样本分布不确定的复杂分析。在β多样性中常被看作一个量化的数值,来反应每个组内各个样本间的群落物种组成差异。接下来带大家感受ANOSIM、PERMANOVA原理的奇妙之处。
二、ANOSIM原理简介
ANOSIM[2](Analysis of similarities)是在1993为解决多物种丰度数据的普遍适用性,而开发的非参检验,至今已被引用14285次,在群落多样性研究中极具权威性。
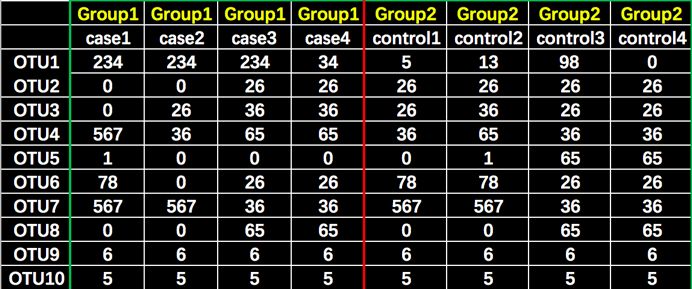
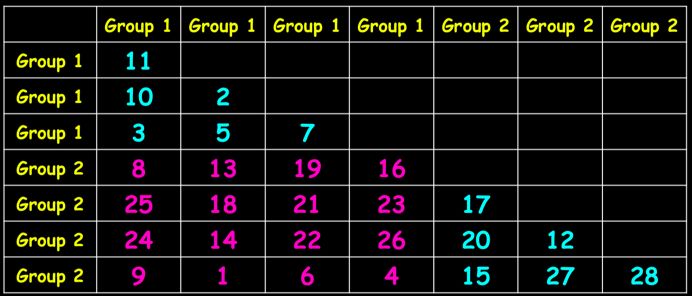
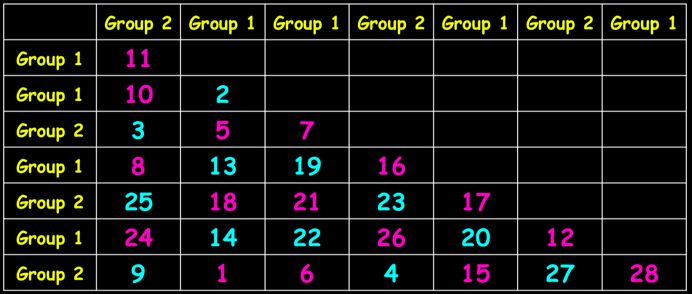
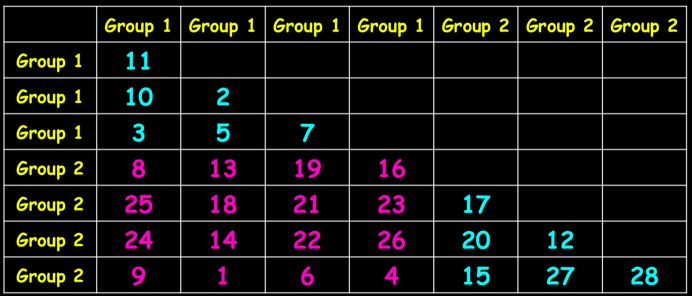
首先,通过变量(物种/OTU等丰度信息)进行样本关系计算,得到距离矩阵(图2 a),距离类型可以是常见的Bray-Curtis,同样Jaccard、Unifrac也适用;
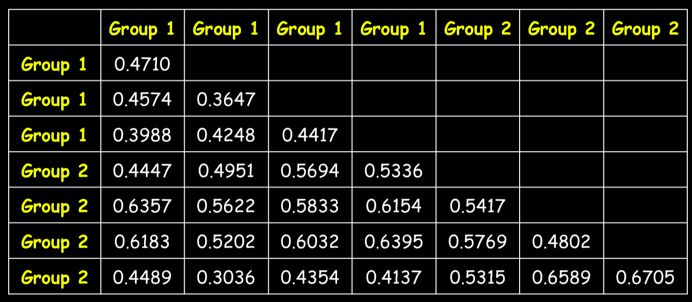
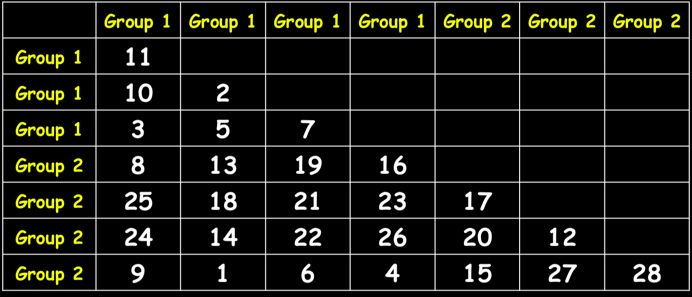
接着,对样本关系从小到大进行排序,最小的距离记为1,依次类推为2,3,4……得到排序后的关系排名(秩)(图2 b);
然后,用上述的秩计算R;
其中:
rB=组间秩的平均值(排序后组间距离的平均值),即图2 b中,橙色区域的平均值,rB=(13+14.5+14.5+7+9+2+4+6+5)/9=8.33;
rW=组内秩的平均值(排序后组内距离的平均值);即图2 b中,蓝色区域的平均值,rW=(3+10+8+1+12+11)/6=7.5;
n为总样本个数。
最后,通过置换检验求p,将样本打乱随机分组,计算置换后的R值(记为Ri),经过N次置换后(至少1000次),Ri大于原始R的概率即为p。
ANOSIM结果输出解读(表1):
其中:
Diffs:差异比较组;
R:差异程度,一般介于(0,1)之间。
R>0,说明组间存在差异(R>0.75:大差异;R>0.5:中等差异,R>0.25:小差异);
R=0或在0附近,表明组间没有差异;
若R出现<0的情况,说明组内差异显著大于组间差异,这个时候表明分组或采样不合理,需要重做实验。
Pvalue:P值,数值小于0.05、0.01或在两者范围内,表明分组有显著性差异,进而反映出目标分组有意义。
三、Adonis原理简介
Adonis[3](non-parametric multivariate analysis of variance),是在ANOSIM 和MANOVA 基础上于2001年开发的另一种分组差异检验方法,在文献中可能大家比较熟悉的是PERMANOVA这个别称。其作用与ANOSIM类似,通过统计检验的p值来判断分群效果是否显著,再辅佐R值判别差异程度!
但两者也有所不同,Adonis的检验模型更多的是借鉴了MANOVA(多元方差分析),本质是基于F统计量的方差分析,利用各种组间距离指数对总方差进行分解,可以分析不同分类因子对群落差异的解释度,并使用置换检验进行统计学检验。
检验原理第一步与ANOSIM一致,计算样本间的距离矩阵,但不再对距离矩阵进行排序;
然后,依据距离矩阵计算F值;
其中:
SST=所有分析对象的平均距离;
SSW=组内平均距离(SSW);
那么组间平均距离为SSA= SST– SSW;
N为总观测数,a为组数。
最后,同样通过随机置换的方式得到p值,先计算多次置换后的伪Fi值(p值精度会随着置换次数的增加而增加,一般来说,精度为0.05至少1000次,精度为0.01至少5000次),当伪Fi值大于原始F观测值的概率即为p值。
以上就是Adonis的原理部分,接下来带大家解读输出的Adonis结果表中(表2),各部分内容所代表的含义。
其中:
Diffs:差异比较组;
Df:自由度;
SumsOfSqs:总方差,又称离差平方和;
MeanSqs :均方(差),即Sums Of Sqs/Df;
Fvalue:F 检验值;
R2:不同分组对样品差异的解释度,即分组方差与总方差的比值,
R2 越大表示分组对差异的解释度越高;
Pvalue:P值,值越小可信度越高。
四、案例应用
Adonis和Anosim在文章中展示的方式有多种,下面就通过几篇应用案例,解锁Adonis和Anosim在高分文献中的花式用法。
案例一:表格展示法[4]
发表期刊:Nature Microbiology(Nature子刊)
影响因子:17.745
发表时间:2021年12月
作者在前期α多样性分析中,得出人体测量指标(治疗和安慰组)之间并没有显著差异,因此继续探索肠道菌群在3个不同年龄组(7-12个月、1-2岁和2岁以上)的beta多样性,以期揭示非洲农村婴儿的肠道菌群受年龄影响的变化规律(表3)。
结果显示年龄组间的PERMANOVA检验和相似性ANOSIM分析的P值为0.0003,表明beta多样性在不同年龄组之间具有显著性差异。
Tips
上述这种直接用表格展示Adonis和Anosim检验结果的方式,仅建议在文章内容极其丰富的情况下,一般推荐用下面这两种方式进行可视化呈现。
案例二:与PCOA、NMDS等搭配法[5]
发表期刊:Genome Medicine
影响因子:11.117
发表时间:2021年9月
作者通过对表现为MCII(minimum clinically important improvement)的患者(MCII+)和没有表现出MCII的患者(MCII-)的多样性研究,探索类风湿关节炎(RA)患者特征的肠道微生物群落差异(图3)。
PERMANOVA结果表明,MCII患者组肠道微生物群落之间具有显著性差异(p=0.002),并且对群落总变异的贡献为3.8% (R2=0.038)。
案例三:盒型图展示法[6]
盒型图是统计检验的经典展示图,例如在下面这篇案例中 。
发表期刊:BioMed Research Internationa
影响因子:2.0
发表时间:2020年6月
作者旨在探讨血液透析对尿毒症患者微生物群落的影响,将患者分为血液透析组(A组)、血液透析+血液透析滤过组(B组)、血液透析+血液透析滤过+血液灌注组(C组)、成人终末期肾脏病ESRD患者(CK组)进行多样性分析(图4)。
Anosim结果表明,CK组与透析组的菌落分布有显著差异;三种不同透析方式患者的菌落分布也有显著差异。
总结:
1)在文章中Adonis和Anosim有单独分开使用,也有同时出现的情况。在实际应用中,如果两种检验结果都与预期一致,那么可以两种检验结果都呈现,在丰富文章内容的同时,还能更加说明分组的合理性。但由于两种检验模型不一致,若出现一种检验是分组有差异,一种没有,择优而选就行。
2)两种检验方法计算原理比较繁琐,并且与不同距离类型(Bray-Curtis、Jaccard、Unifrac)搭配,输出的结果可能不一致,也许一次分析要花费不少时间,才能达到理想的结果,那么有高效率的解决方法吗?有的,给大家推荐基迪奥的在线平台,只要把简单的物种、otu丰度数据导进去,分分钟就能出结果,还能一键切换距离类型,减少试错和时间成本。




【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 阿里最新开源QwQ-32B,效果媲美deepseek-r1满血版,部署成本又又又降低了!
· 开源Multi-agent AI智能体框架aevatar.ai,欢迎大家贡献代码
· Manus重磅发布:全球首款通用AI代理技术深度解析与实战指南
· 被坑几百块钱后,我竟然真的恢复了删除的微信聊天记录!
· 没有Manus邀请码?试试免邀请码的MGX或者开源的OpenManus吧