用TCGA数据库分析癌症和癌旁组织的表达差异
上周收到一条求助信息:“如何用TCGA数据库分析LINC00152在卵巢癌与正常组织的的表达差异?”
所以以这个题目为记录分析过程如下:
一、下载数据
a)进入网站https://cancergenome.nih.gov/ 网页截图如下:
b)进入数据下载 Launch Data Portal ,截图如下:
进入数据下载接口后,有Projects Exploration Analysis Repository 四个栏目,我们数据下载可进入Repository菜单栏,截图如下:
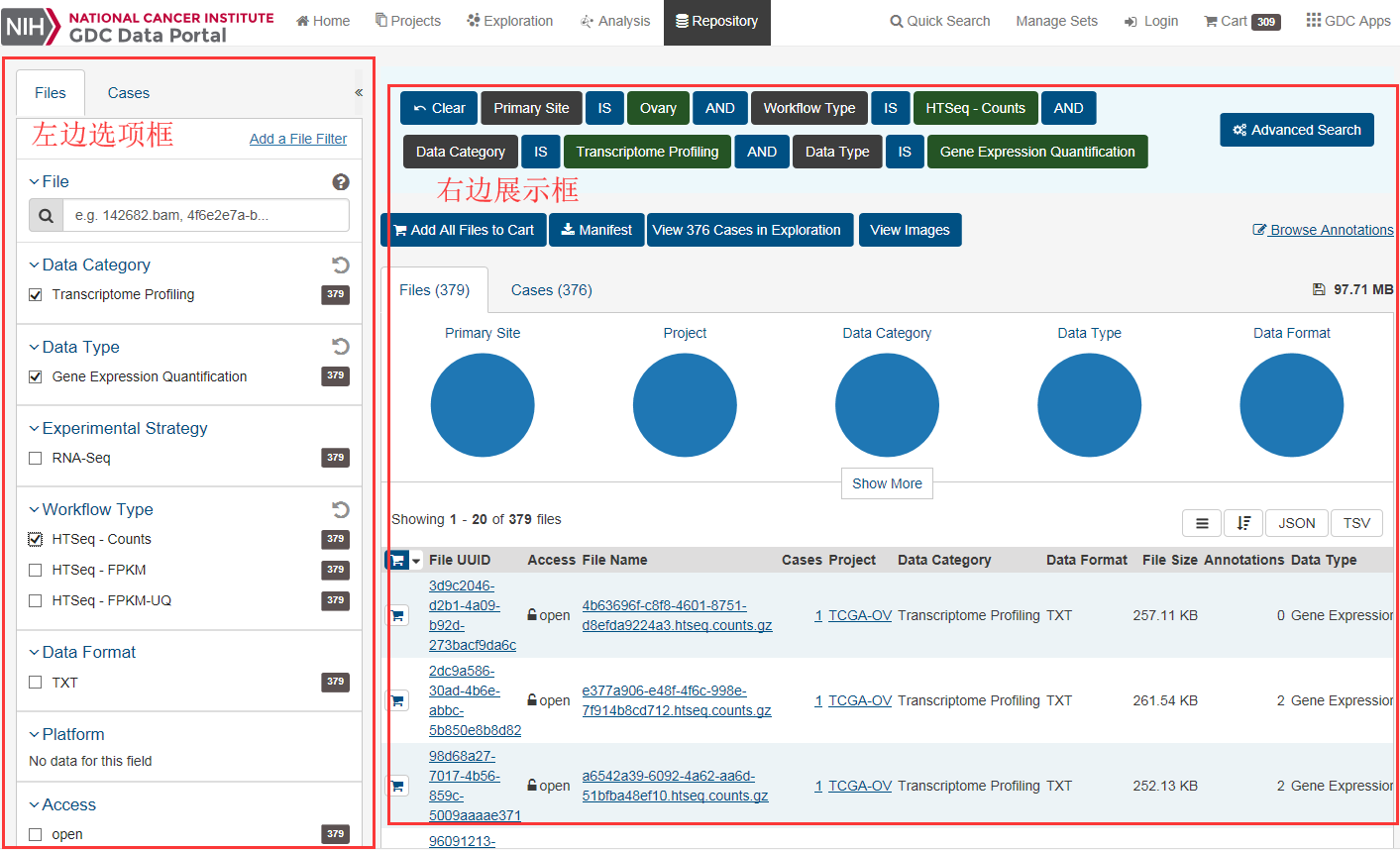
网页分成左右两边,左边主要是提供用户数据选择和过滤的窗口,右边是根据用户的选择后显示及其统计结果。左边选择分为 Cases 和 Files两大类。
根据我们的研究,目的是要看LINC RNA在卵巢癌和正常组织的表达差异,所以我们在左边的栏目的Cases下选择Ovary,在Files 下选择 RNA-seq ,这些选项选择完毕,会出现上面的那张截图
c)下载路径文件
选好文件后,如上图将文件加入购物车,截图如下:
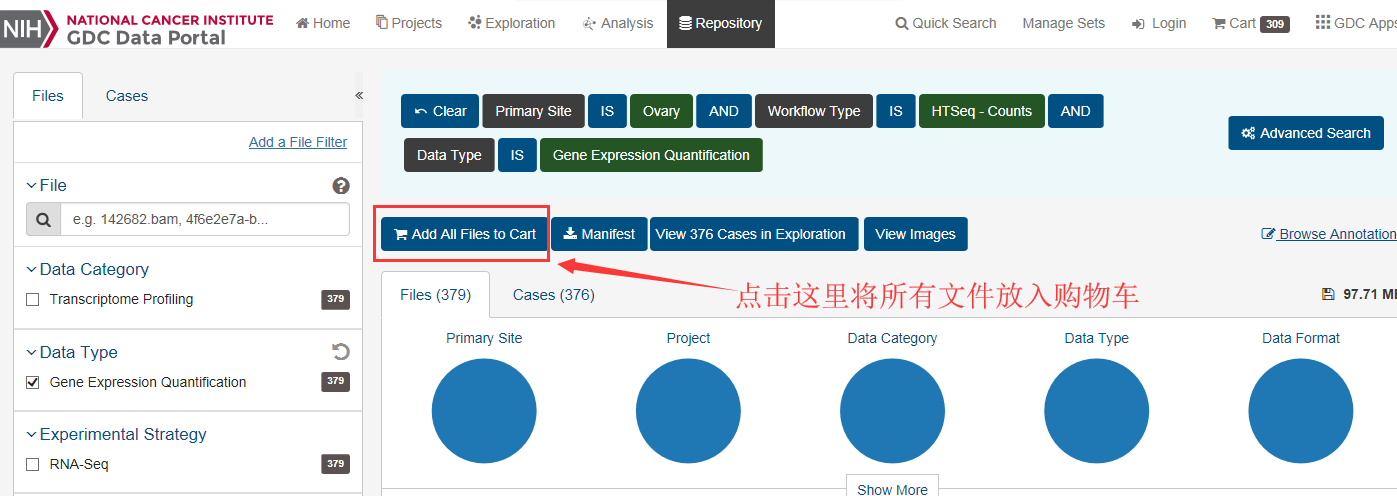
然后点击右上角的Cart,出现如下截图:
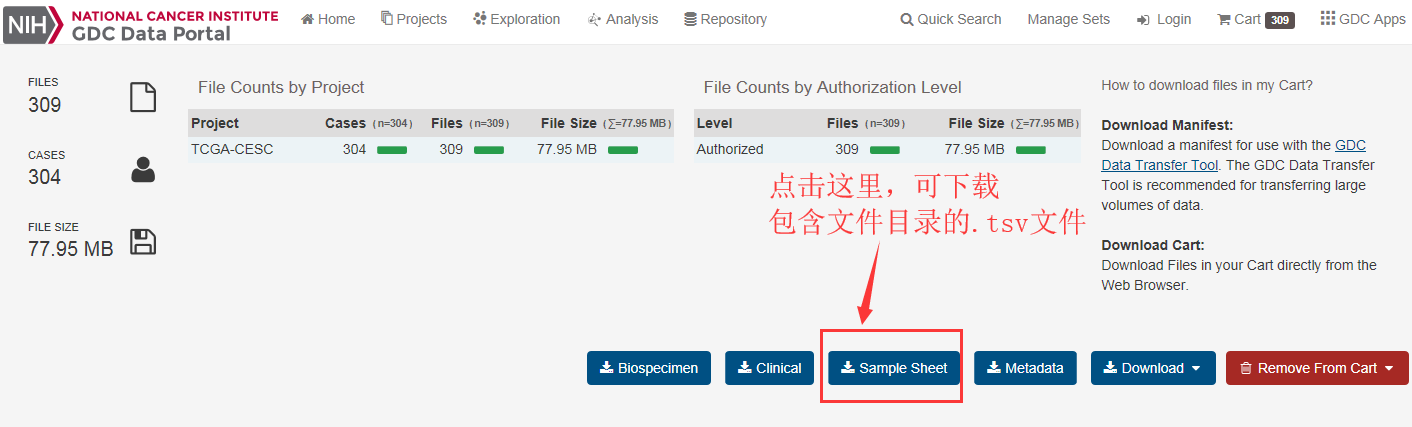
点击Sample sheet之后,包含所需文件目录的.tsv文件gdc_sample_sheet.2018-05-22.tsv就可以下载了,放到对应的目录下。
用NotePad打开文件如下:
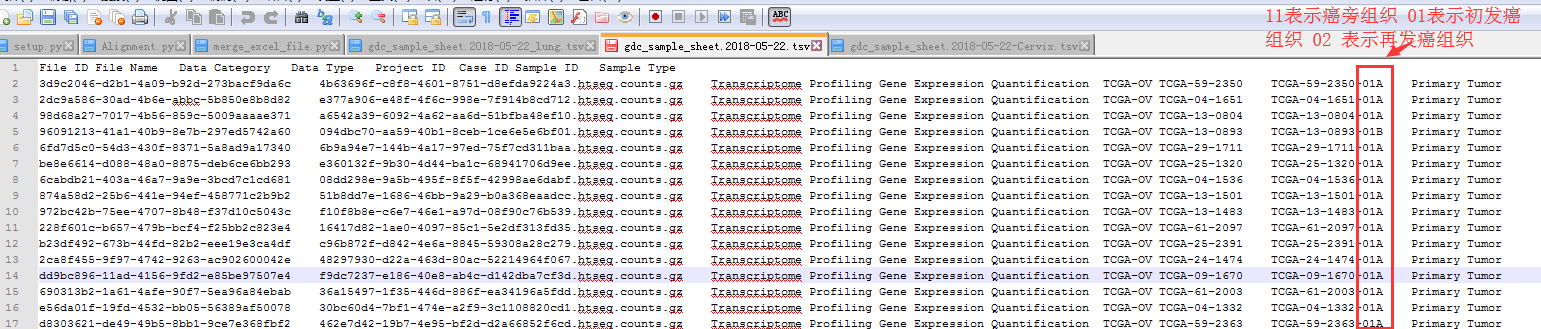
d) 在linux下批量下载文件
将该文件放在linux的 /home/zdwu/rnaseq/11_source_data 目录下,并在该目录下批量下载数据,代码如下:
cat gdc_sample_sheet.2018-05-22.tsv | while read line do echo https://portal.gdc.cancer.gov/files/${line:0:(36-0)} wget -c https://gdc-api.nci.nih.gov/data/${line:0:(36-0)} -O ${line:167:(184-167)}'.htseq.counts.gz' done
下载完毕后查看文件如下:
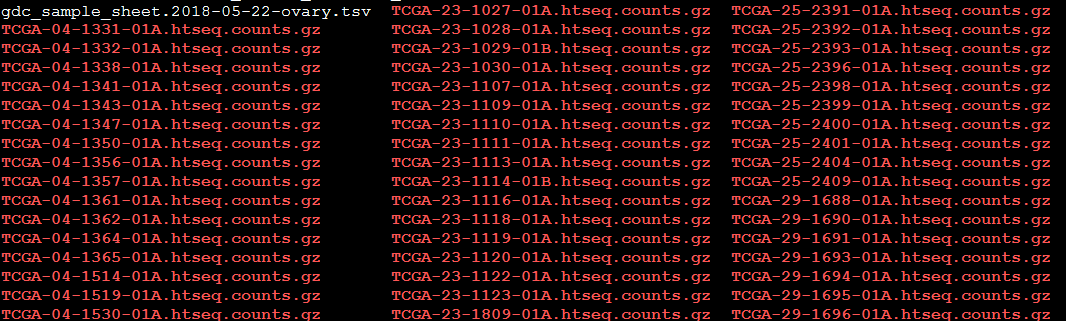
用如下命令,确认文件个数是否完整,完整后数据备用。
ls A-* | wc -l
二、数据分析
a)数据解压
用命令行 解压,解压后得到可读的数据。
zdwu@ubuntu://home/zdwu/rnaseq/11_source_data/ovary$ gunzip *counts
b)找出Linc00152的表达量
由于从TCGA下载的数据里的基因明都是ensemble ID,所以需要 从NCBI 查找Linc00152对应的 ensemble ID,找出的结果是Ensembl:ENSG00000222041

注意:此处只有一个基因,所用手动从NCBI 找出ensemble ID是简单的,但是如果看的是大量的基因,那这将会非常通过,这时就需要通过ID转换文件来编程转换。
基因ID 转换文件的下载地址 :ftp://ftp.ncbi.nlm.nih.gov/gene/DATA/ ,里面有gene2ensemble.gz gene2accession.gz gene2go.gz 等文件可以下载,根据这些文件,写个小脚本就可以批量转换了。
c)整合多样本的LINC00152基因表达counts
zdwu@ubuntu://home/zdwu/rnaseq/11_source_data/ovary$ for file in *counts > do > echo ${file:0:12} >> ovary_linc00152.txt > echo ${file:13:(16-13)} >> ovary_linc00152.csv > cat ${file} | grep "ENSG00000222041" >> ovary_linc00152.csv > done
最后将得到的ovary_linc00152.csv 文件拷贝至于windows电脑,截图如下:
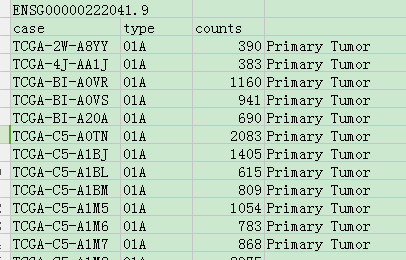
这是没有normalized的数据,如果需要不同样本之间比较的话进行normalized,再简单的统计不同组之间样本的 t-test。分析完毕。



