生物信息学
GU也可以配对,即“wobble” pairing GU。
Hairpin发夹结构,最少不能少于3个碱基。没有配对
Bulge 单侧配对
Loop双侧配对
假结,游离的leading edge与hairpin相结合。大部分软件不考虑此结构。
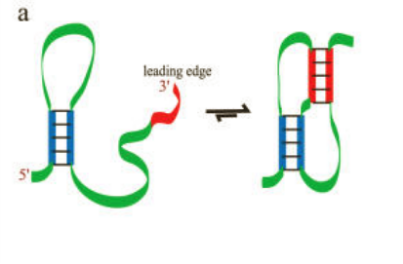
表示RNA5种结构:
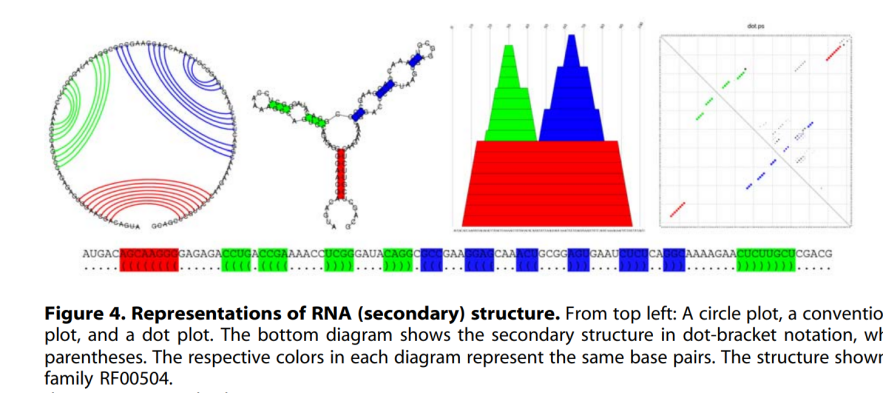
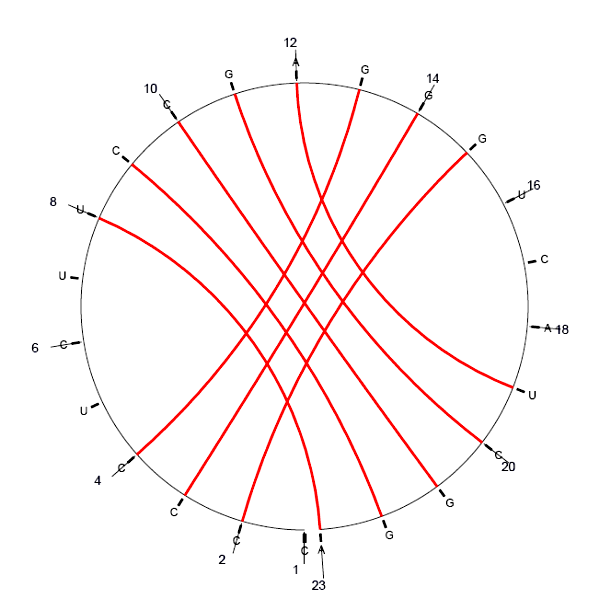
如果有假结,则环形图会有交叉:
颜色代表稳定性:

但是RNA序列较长则得到正确的预测比较困难,因为配对可能性上升。所以采用150bp以内的序列预测比较准确。
常用方法:
1.共变化(进化)方法,基于相似性比对。
2.单序列预测是从头预测方法,基于采用最小自由能,因为自由能越低越稳定。
Eg:查表计算二级结构自由能

计算机使用动态规划计算
==================================================
不同RNA结构:
snRNA参与RNA剪接
snoRNA参与rRNA加工
siRNA就是RNAi,与DNA配对可引导gene editing。
microRNA:早期方法和现有数据相结合或许是一个突破点。需要软件的局限性和偏向性。
piRNA:在低等和节肢动物中出现多。基因组上成簇存在,乒乓模型,调控翻译。
LncRNA:
antisense RNAs:反RNA
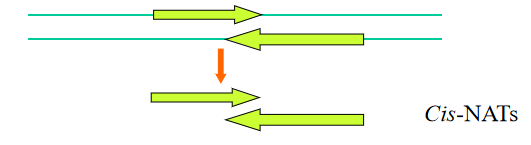
cis-NATs:两个位置在局部
trans-NATs两个位置在远处
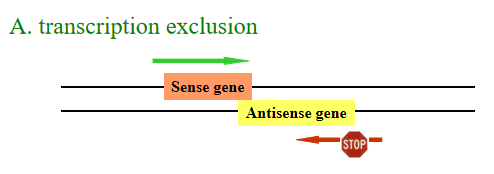
如果需要功能互斥,则基因水平可以是这种结构,只能有一种蛋白质复合结构在序列上:
假基因:假基因一共可以分为三类:unitary pseudogene;duplicated pseudogene以及processed pseudogene。Unitary pseudogene是指蛋白质编码基因在进化过程中自身累积突变而形成,这一类假基因在本物种中找不到亲本基因。Duplicated psuedogene是指蛋白质编码基因在进化过程中发生复制,其中一个副本保留原有的功能,而另一个副本则累积突变形成假基因。这一类假基因保留了亲本基因原有的promoter区和内含子区。Processed pseudogene则是由蛋白质编码基因的转录产物反转录成cDNA以后,反插回基因组中,并在此过程中累积突变而形成的假基因。这一类假基因由于源自亲本基因的转录产物而失去了亲本基因的启动子区和内含子区。
环形RNA:




