(Panda has a high heterozygosity rate)
通过对mtDNA(为了预测SNP的密度)的分析,可知panda的多样性,当前全基因组数据才能完全建立模型。
mitochondrial control region DNA:卫星DNA:微衛星(英语:Microsatellite,亦称为简单重复序列(英语:Simple Sequence Repeats,SSRs
在二倍体基因组上重排可用序列(占85%),占90%的序列用来检查突变(因为他们都有特异性位点)。
我们发现了panda genome的杂合SNP,计算了杂合率(常染色体和编码区),比人高(因为测序取材地的杂交背景)。常染色体比性染色体速度快,转换(transitions)比颠换(transversions)快(因为在构树时,要考虑碱基替换关系)。所以我们认为,熊猫可能虽然种群数目小但是有挽救潜力。
转换:嘌呤和嘌呤之间的替换,或嘧啶和嘧啶之间的替换,比较容易。
颠换:嘌呤和嘧啶之间的替换,比较困难。
杂合SNP密度呈现双峰分布,前一个峰比后一个峰高,TMRCA与有效群体大小成反比(但是MRCA与有效种群大小成正比,同时MRCA正比于SNP数目?????,所以按照理想发展情况,该图应该仅有一个峰,但是此图有两个峰,说明有一定的改变和瓶颈)反映出熊猫种群历史上的有效种群数目改变和瓶颈。
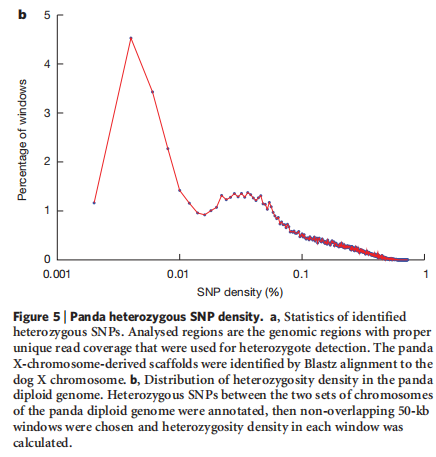
TMRCA:是“最近共同祖先年代”(Time to most recent common ancestor),可以决定SNP
Wright–Fisher population genetic model可以得到方差有效群体大小和近交有效群体大小
单核苷酸多态性(SNP)的密度可以通过微卫星DNA进行预测
鉴定了small indels,发现indel杂合率比SNP杂合率低一个数量级。同时发现了大于100bp的插入、删除、倒位片段,大部分片段与转座原件重叠所以overlap(因为这些片段正来自有功能的转座元件)(因为结构多样性被低估(因为组装过于碎片化不能识别长结构多样性))





