(Panda, dog and human repeat comparison):与其他动物比较重复序列
我们使用Repbase 库(重复序列库)+已知的转录原件序列+识别软件,评估出转录原件占比,并且与狗和人相比。用Repbase数据库(扩张度来自repeat base)分析熊猫基因中的转录原件的扩张度,得到:大部分熊猫转录原件基因有超过10% 的共有序列的扩张度(这是因为repbase数据库数据基于哺乳动物基因,并没有大熊猫基因)。小部分低于10%的扩张度(这可能是哺乳动物最近起源的活性转座子)。
Repbase library:database mask and annotation repetitive DNA
RepeatModeller:de-novo repeat family identification and modeling package
(Panda genome has a low divergence rate):
(investigate the rate of recent segmental duplication):采用self-sequence alignment,先在全基因组识别出重复片段个数及其大小(因为测序reads与组装的全基因组相比较,测序readsmapping上的重复片段多,所以在assembly过程中丢失了一些重复片段,同时因为这些片段的reads深度明显高于其他区域,所以要与depth相乘),所以根据平均depth(重复片段的average depth)和测序reads大小,得到重复片段的大小。
self-sequence alignment:自己的基因和自己的基因相互比对得到
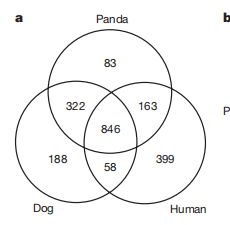
(investigate panda genome conservation and evolution,)比对了狗,人,熊猫的全基因组,三者有相似序列,不相似的部分:包括狗与熊猫的相似度高于(人与狗,人与熊猫),熊猫的特异性序列最少,所以熊猫基因组的扩展性是最低的。
(the panda, dog and human genomes had high genomic synteny)比人,狗,熊猫第二条染色体的35条scaffold,没发现大规模重排。conserved synteny+pairwise syntenic regions
conserved synteny:保守同源区;pairwise syntenic regions:成对同源区
(genomic rearrangement events)重排事件,用几乎全基因组的scaffold序列,比较狗和熊猫的染色体间较小同源片段,片段有大有小(之前打断成不同大小的小序列),使用人类基因作为参照,发现dog中重排是panda的三倍,所以表明panda的扩张度较低。
Cutoff:截取片段





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 10年+ .NET Coder 心语,封装的思维:从隐藏、稳定开始理解其本质意义
· .NET Core 中如何实现缓存的预热?
· 从 HTTP 原因短语缺失研究 HTTP/2 和 HTTP/3 的设计差异
· AI与.NET技术实操系列:向量存储与相似性搜索在 .NET 中的实现
· 基于Microsoft.Extensions.AI核心库实现RAG应用
· 10年+ .NET Coder 心语 ── 封装的思维:从隐藏、稳定开始理解其本质意义
· 地球OL攻略 —— 某应届生求职总结
· 提示词工程——AI应用必不可少的技术
· Open-Sora 2.0 重磅开源!
· 周边上新:园子的第一款马克杯温暖上架