4+ 非常经典的hub基因鉴定路线
原文公众号:一起实验网
对下述分析方法感兴趣或者没有研究思路的小伙伴,欢迎踊跃探讨!
分享一篇2022年9月发表在Front Physiol(IF:4.755)的文章《Identification of hub genes associated with acute kidney injury induced by renal ischemia-reperfusion injury in mice》(PMID: 36246123),作者通过对基因表达数据的综合分析,为缺血再灌注损伤-急性肾损伤的发病机制提供新的潜在生物标志物和见解。该思路同样适用于其他非肿瘤或肿瘤的研究。
背景&方法
背景:
急性肾损伤(AKI)是一种严重的临床综合征,缺血再灌注损伤(RI)是急性肾损伤的重要病因。
方法:
1.对数据集进行归一化处理;
2.分别鉴定两个个数据集中的差异表达基因;
3.DEG的GO功能和KEGG通路富集分析;
4.DEG的基因集富集分析;
5.DEG的PPI网络构建与hub基因的鉴定;
6.转录因-hub基因网络和miRNA-hub基因网络的构建;
7.hub基因相互作用药物的识别;
8.IRI-AKI病hub基因表达水平。
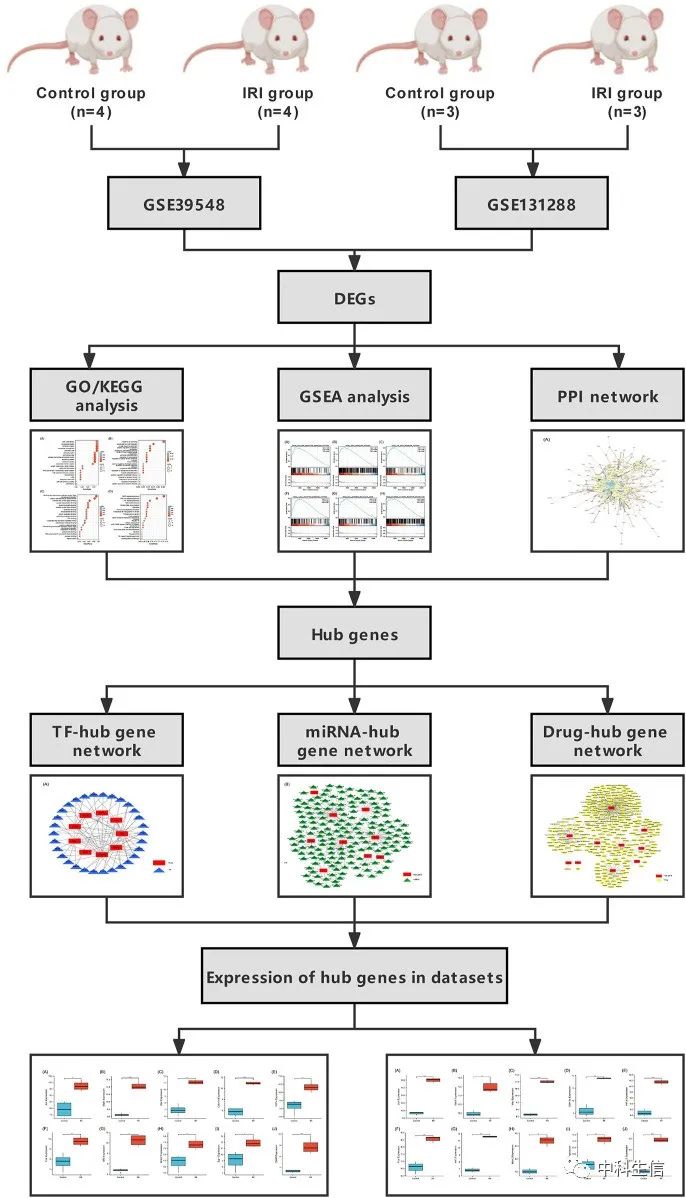
结果
差异表达基因的鉴定
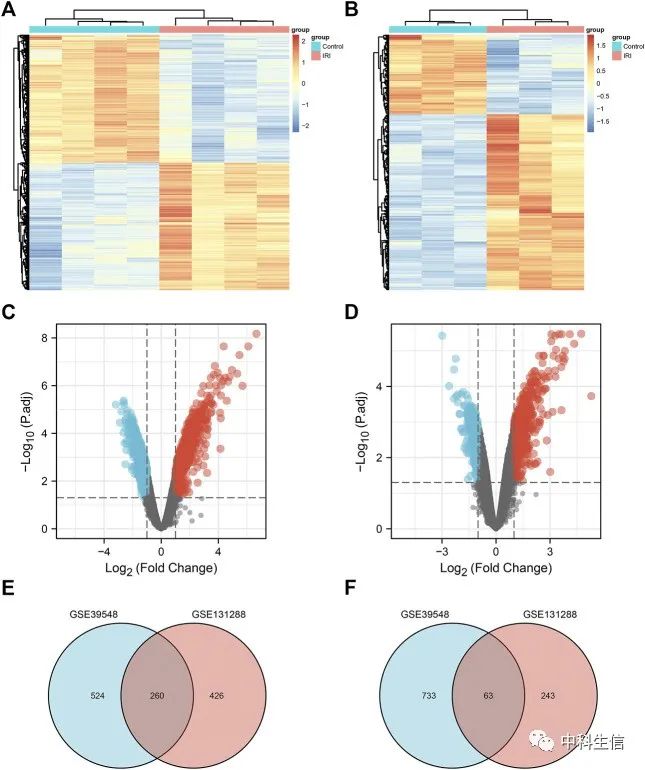
从GSE39548数据集中共筛选出1580个DEG,其中IRI组上调784个,下调796个。在GSE131288数据集中,共筛选出992个DEG,其中在IRI组中表达上调的有686个,下调的有306个。两个数据集的DEG取交集,得到323个共同DEG,其中在IRI组中上调的有260个,下调的有63个。
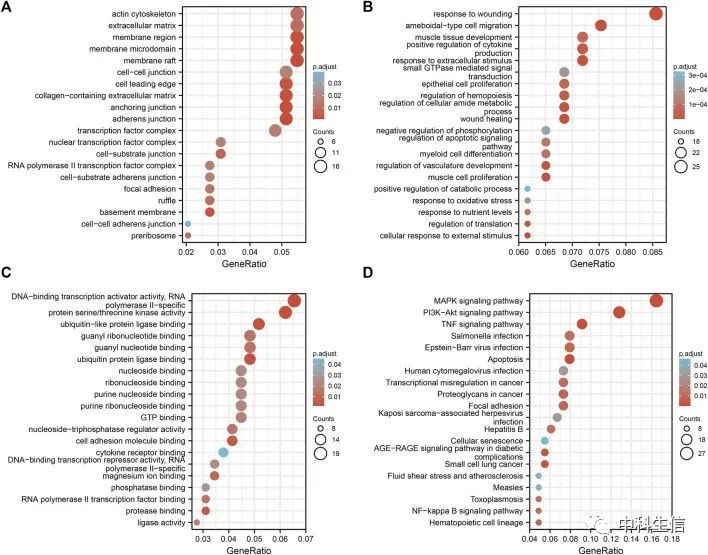
323个DEG的GO和KEGG途径富集分析
利用DAVID数据库,确定了这323个DEG的潜在功能。
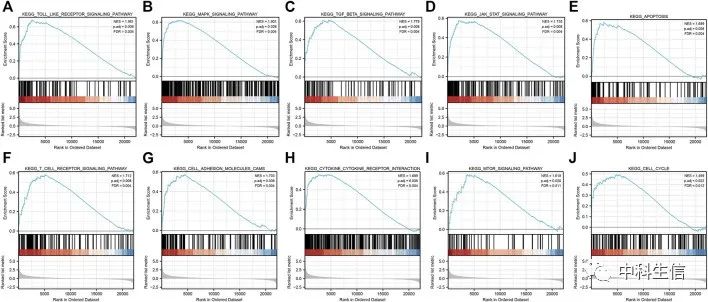
基因集富集分析(GSEA)
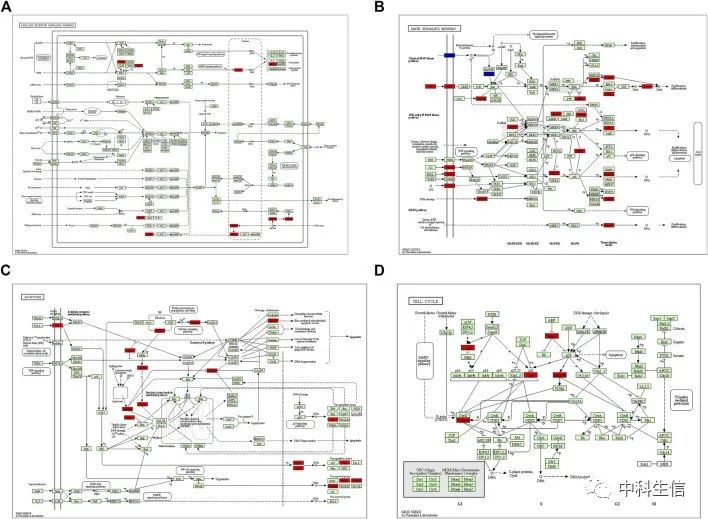
采用C2中的CP基因集作为背景基因集,进行GSEA分析。此外,还利用KEGG-MAP数据库绘制了与缺血-再灌注损伤相关的4条信号通路。
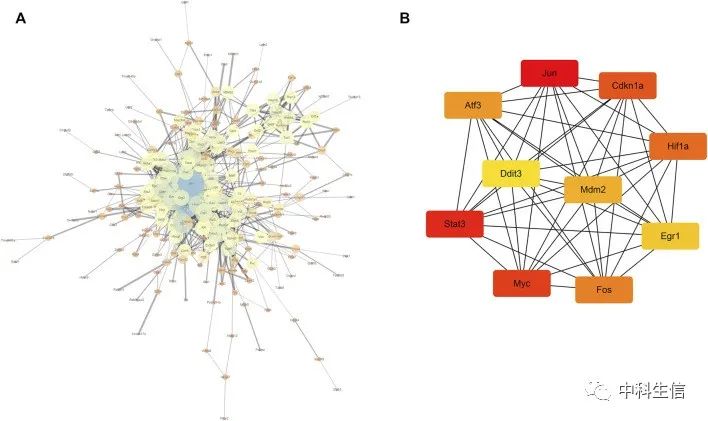
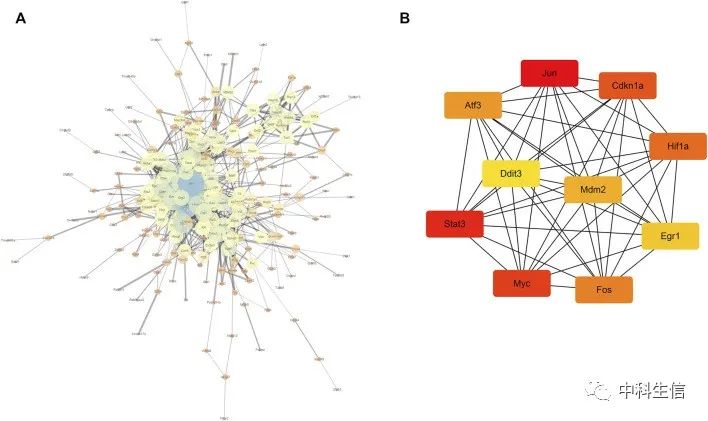
蛋白质-蛋白质相互作用网络的构建与hub基因的鉴定
将上述分析得到的323个共同差异表达基因输入STRING数据库,并将所得结果导入Cytoscape软件构建PPI网络。此外,利用cytoHubba插件计算节点连接的紧密度,确定前10个基因为hub基因,分别为Jun、Stat3、Myc、Cdkn1a、Hif1a、Fos、Atf3、Mdm2、Egr1、dit3。
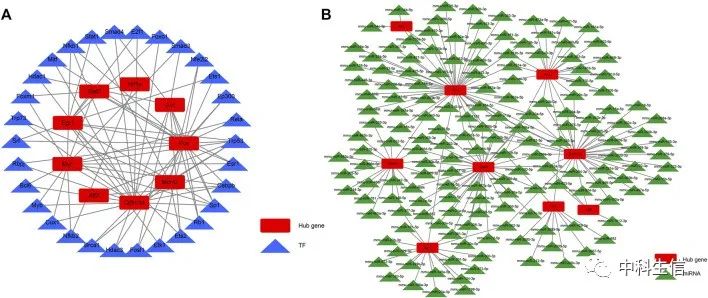
转录因-hub基因网络和miRNA-hub基因网络的构建
我们旨在更好地了解hub基因在AKI发病机制中的调节作用。使用STRUST数据库预测了34个能够与10个枢纽基因相互作用的TF,并构建TF-hub基因调控网络。通过starBase数据库预测了可以与10个hub基因相互作用的miRNA,并构建miRNA-hub基因相互作用网络
识别与hub基因相互作用的潜在药物
使用DGIdb数据库对可能与hub基因相互作用的药物或分子化合物进行了预测,共筛选出299种可能与hub基因存在调控关系的药物或分子化合物,其中与HIF1A相互作用的药物数量最多。
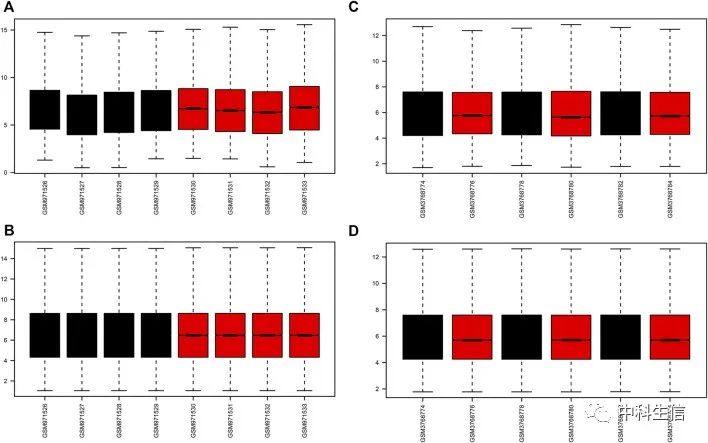
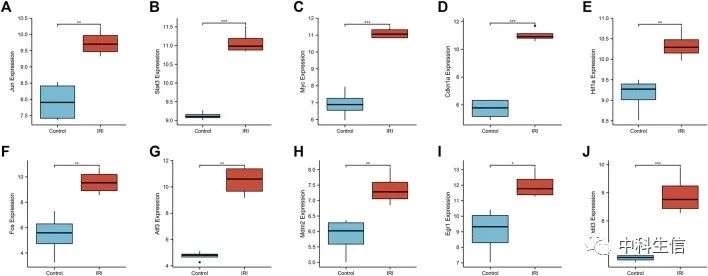
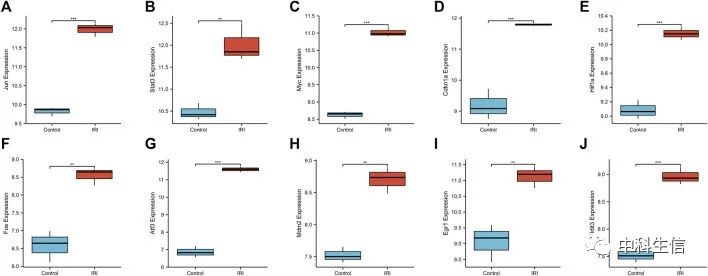
IRI-AKI病hub基因表达水平
在GSE39548数据集中,IRI-AKI组的10个hub基因的表达明显高于对照组。同样,在GSE131288数据集中,IRI-AKI组中有10个hub基因的表达显著增加。
原文公众号:一起实验网
对上述分析方法感兴趣或者没有研究思路的小伙伴,欢迎踊跃探讨!



【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 震惊!C++程序真的从main开始吗?99%的程序员都答错了
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 单元测试从入门到精通
· 上周热点回顾(3.3-3.9)
· winform 绘制太阳,地球,月球 运作规律