机器学习(二)——K-均值聚类(K-means)算法
最近在看《机器学习实战》这本书,因为自己本身很想深入的了解机器学习算法,加之想学python,就在朋友的推荐之下选择了这本书进行学习,在写这篇文章之前对FCM有过一定的了解,所以对K均值算法有一种莫名的亲切感,言归正传,今天我和大家一起来学习K-均值聚类算法。
一 K-均值聚类(K-means)概述
1. 聚类
“类”指的是具有相似性的集合。聚类是指将数据集划分为若干类,使得类内之间的数据最为相似,各类之间的数据相似度差别尽可能大。聚类分析就是以相似性为基础,对数据集进行聚类划分,属于无监督学习。
2. 无监督学习和监督学习
上一篇对KNN进行了验证,和KNN所不同,K-均值聚类属于无监督学习。那么监督学习和无监督学习的区别在哪儿呢?监督学习知道从对象(数据)中学习什么,而无监督学习无需知道所要搜寻的目标,它是根据算法得到数据的共同特征。比如用分类和聚类来说,分类事先就知道所要得到的类别,而聚类则不一样,只是以相似度为基础,将对象分得不同的簇。
3. K-means
k-means算法是一种简单的迭代型聚类算法,采用距离作为相似性指标,从而发现给定数据集中的K个类,且每个类的中心是根据类中所有值的均值得到,每个类用聚类中心来描述。对于给定的一个包含n个d维数据点的数据集X以及要分得的类别K,选取欧式距离作为相似度指标,聚类目标是使得各类的聚类平方和最小,即最小化:
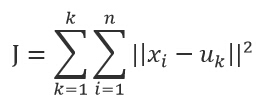
结合最小二乘法和拉格朗日原理,聚类中心为对应类别中各数据点的平均值,同时为了使得算法收敛,在迭代过程中,应使最终的聚类中心尽可能的不变。
4. 算法流程
K-means是一个反复迭代的过程,算法分为四个步骤:
1) 选取数据空间中的K个对象作为初始中心,每个对象代表一个聚类中心;
2) 对于样本中的数据对象,根据它们与这些聚类中心的欧氏距离,按距离最近的准则将它们分到距离它们最近的聚类中心(最相似)所对应的类;
3) 更新聚类中心:将每个类别中所有对象所对应的均值作为该类别的聚类中心,计算目标函数的值;
4) 判断聚类中心和目标函数的值是否发生改变,若不变,则输出结果,若改变,则返回2)。
用以下例子加以说明:
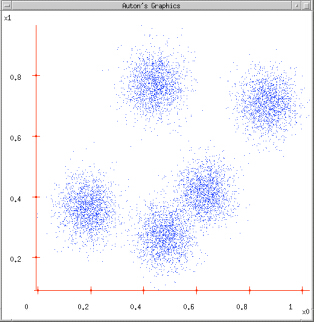

图1 图2

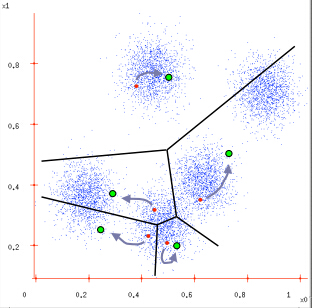
图3 图4
图1:给定一个数据集;
图2:根据K = 5初始化聚类中心,保证 聚类中心处于数据空间内;
图3:根据计算类内对象和聚类中心之间的相似度指标,将数据进行划分;
图4:将类内之间数据的均值作为聚类中心,更新聚类中心。
最后判断算法结束与否即可,目的是为了保证算法的收敛。
二 python实现
首先,需要说明的是,我采用的是python2.7,直接上代码:
#k-means算法的实现
#-*-coding:utf-8 -*-
from numpy import *
from math import sqrt
import sys
sys.path.append("C:/Users/Administrator/Desktop/k-means的python实现")
def loadData(fileName):
data = []
fr = open(fileName)
for line in fr.readlines():
curline = line.strip().split('\t')
frline = map(float,curline)
data.append(frline)
return data
'''
#test
a = mat(loadData("C:/Users/Administrator/Desktop/k-means/testSet.txt"))
print a
'''
#计算欧氏距离
def distElud(vecA,vecB):
return sqrt(sum(power((vecA - vecB),2)))
#初始化聚类中心
def randCent(dataSet,k):
n = shape(dataSet)[1]
center = mat(zeros((k,n)))
for j in range(n):
rangeJ = float(max(dataSet[:,j]) - min(dataSet[:,j]))
center[:,j] = min(dataSet[:,j]) + rangeJ * random.rand(k,1)
return center
'''
#test
a = mat(loadData("C:/Users/Administrator/Desktop/k-means/testSet.txt"))
n = 3
b = randCent(a,3)
print b
'''
def kMeans(dataSet,k,dist = distElud,createCent = randCent):
m = shape(dataSet)[0]
clusterAssment = mat(zeros((m,2)))
center = createCent(dataSet,k)
clusterChanged = True
while clusterChanged:
clusterChanged = False
for i in range(m):
minDist = inf
minIndex = -1
for j in range(k):
distJI = dist(dataSet[i,:],center[j,:])
if distJI < minDist:
minDist = distJI
minIndex = j
if clusterAssment[i,0] != minIndex:#判断是否收敛
clusterChanged = True
clusterAssment[i,:] = minIndex,minDist ** 2
print center
for cent in range(k):#更新聚类中心
dataCent = dataSet[nonzero(clusterAssment[:,0].A == cent)[0]]
center[cent,:] = mean(dataCent,axis = 0)#axis是普通的将每一列相加,而axis=1表示的是将向量的每一行进行相加
return center,clusterAssment
'''
#test
dataSet = mat(loadData("C:/Users/Administrator/Desktop/k-means/testSet.txt"))
k = 4
a = kMeans(dataSet,k)
print a
'''
三 MATLAB实现
之前用MATLAB做过一些聚类算法方面的优化,自然使用它相比python更得心应手一点。根据算法的步骤,编程实现,直接上程序:
%%%K-means
clear all
clc
%% 构造随机数据
mu1=[0 0 0];
S1=[0.23 0 0;0 0.87 0;0 0 0.56];
data1=mvnrnd(mu1,S1,100); %产生高斯分布数据
%%第二类数据
mu2=[1.25 1.25 1.25];
S2=[0.23 0 0;0 0.87 0;0 0 0.56];
data2=mvnrnd(mu2,S2,100);
%第三个类数据
mu3=[-1.25 1.25 -1.25];
S3=[0.23 0 0;0 0.87 0;0 0 0.56];
data3=mvnrnd(mu3,S3,100);
mu4=[1.5 1.5 1.5];
S4=[0.23 0 0;0 0.87 0;0 0 0.56];
data4 =mvnrnd(mu4,S4,100);
%显示数据
figure;
plot3(data1(:,1),data1(:,2),data1(:,3),'+');
title('原始数据');
hold on
plot3(data2(:,1),data2(:,2),data2(:,3),'r+');
plot3(data3(:,1),data3(:,2),data3(:,3),'g+');
plot3(data4(:,1),data4(:,2),data3(:,3),'y+');
grid on;
data=[data1;data2;data3;data4];
[row,col] = size(data);
K = 4;
max_iter = 300;%%迭代次数
min_impro = 0.1;%%%%最小步长
display = 1;%%%判定条件
center = zeros(K,col);
U = zeros(K,col);
%% 初始化聚类中心
mi = zeros(col,1);
ma = zeros(col,1);
for i = 1:col
mi(i,1) = min(data(:,i));
ma(i,1) = max(data(:,i));
center(:,i) = ma(i,1) - (ma(i,1) - mi(i,1)) * rand(K,1);
end
%% 开始迭代
for o = 1:max_iter
%% 计算欧氏距离,用norm函数
for i = 1:K
dist{i} = [];
for j = 1:row
dist{i} = [dist{i};data(j,:) - center(i,:)];
end
end
minDis = zeros(row,K);
for i = 1:row
tem = [];
for j = 1:K
tem = [tem norm(dist{j}(i,:))];
end
[nmin,index] = min(tem);
minDis(i,index) = norm(dist{index}(i,:));
end
%% 更新聚类中心
for i = 1:K
for j = 1:col
U(i,j) = sum(minDis(:,i).*data(:,j)) / sum(minDis(:,i));
end
end
%% 判定
if display
end
if o >1,
if max(abs(U - center)) < min_impro;
break;
else
center = U;
end
end
end
%% 返回所属的类别
class = [];
for i = 1:row
dist = [];
for j = 1:K
dist = [dist norm(data(i,:) - U(j,:))];
end
[nmin,index] = min(dist);
class = [class;data(i,:) index];
end
%% 显示最后结果
[m,n] = size(class);
figure;
title('聚类结果');
hold on;
for i=1:row
if class(i,4)==1
plot3(class(i,1),class(i,2),class(i,3),'ro');
elseif class(i,4)==2
plot3(class(i,1),class(i,2),class(i,3),'go');
elseif class(i,4) == 3
plot3(class(i,1),class(i,2),class(i,3),'bo');
else
plot3(class(i,1),class(i,2),class(i,3),'yo');
end
end
grid on;
最终的结果如下图5和图6:
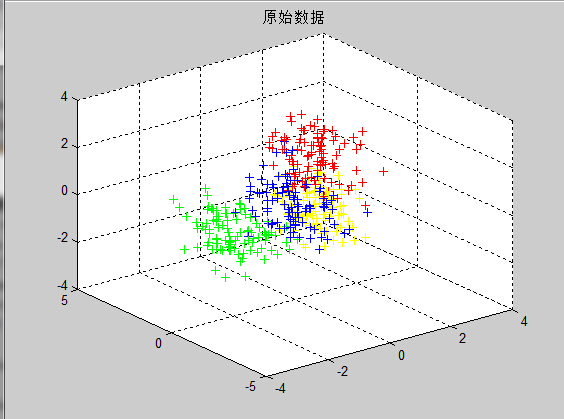
图5 原始数据
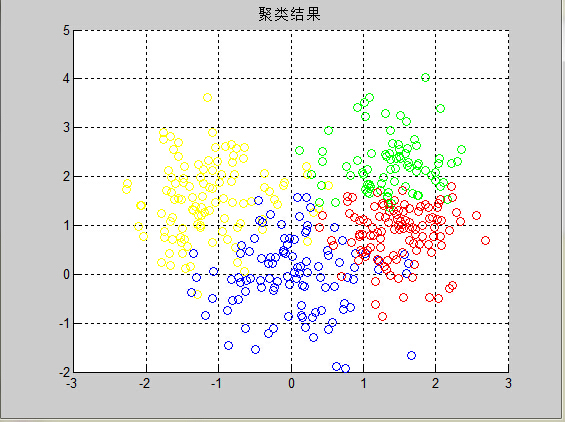 图6 聚类结果
图6 聚类结果
总结:在这次程序的调试中,其实出现的问题还是蛮多的,相似度指标依旧选用的是欧氏距离。在之前,一直是按照公式直接计算的,可欧氏距离其实就是2范数啊,2范数属于酉不变范数,因此矩阵的2范数就是矩阵的最大奇异值,在求解过程中可以直接采用norm函数简化。
上图中的结果可以清晰的看到算法具有一定的聚类效果,要进一步验证的话,可以采取MCR或者NMI和ARI这些常用的准则进行衡量聚类结果的优劣,在此我选取MCR进行验证,代码如下:
%% 采用MCR判定聚类效果
B = class(:,4);
B = reshape(B,1,row);
A = [ones(1,100),2 * ones(1,100),3 *ones(1,100),4 * ones(1,100)];
sum = 0;
for i = 1:row
if ( A(1,i) ~= B(1,i))
sum = sum + 1;
end
end
MCR = sum / row;
fprintf('MCR = %d\n',MCR);
多次计算平均求得的MCR= 0.53,表明误分率还是蛮大的,聚类效果并不是很理想,究其原因:虽然算法收敛,但算法只是收敛到了局部最小值,而并非全局最小值,所以可以引入二分K-均值对算法进行优化。
除此之外,FCM算法在一定程度上也是对算法的一个优化吧。
进而导入UCI数据库中的wine数据进行测试,结果甚是不理想,至于原因吧,算法本身的性能是占一部分的,还有可能是数据的维数相对较多......在此我也不敢妄加猜测,之后慢慢验证吧......



