ggplot2绘制KEGG富集散点图
散点图是 KEGG 富集分析结果的图形化展示方式。在此图中,KEGG 富集程度通过 rich factor, qvalue 和富集到此通路上的基因个数来衡量。其中 rich factor 指差异表达的基因中位于该 pathway 条目的基因数目与所有有注释基因中位于该 pathway 条目的基因总数的比值。Rich factor越大,表示富集的程度越大。qvalue 是做过多重假设检验校正之后的 pvalue, qvalue 的取值范围为[0,1],越接近于零,表示富集越显著。
1.数据的读取与处理
这里读取的是用KOBAS网页工具分析的KEGG结果
pathway<-read.table("SFTSV_24vscontrol_DE_mRNA_kegg.txt",header = T,sep = "\t",stringsAsFactors = F) View(pathway)
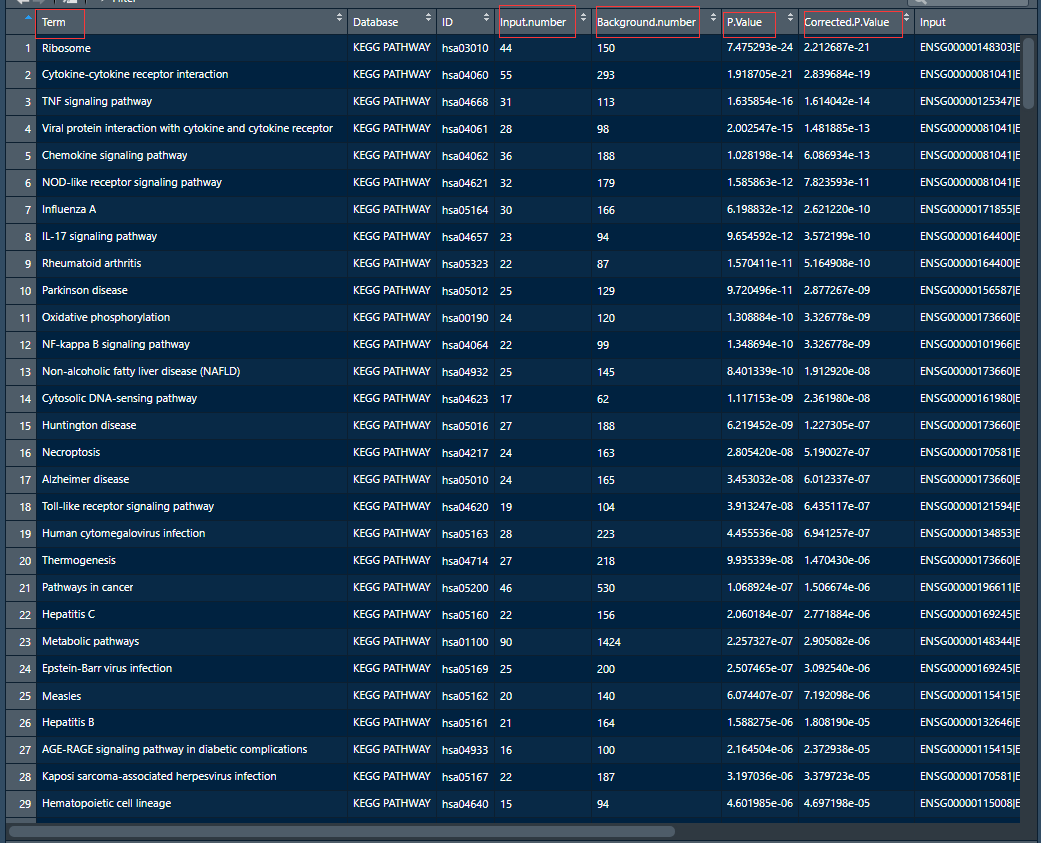
KOBAS网页工具分析的条目里没有Rich factor这一项,所以我们需要在数据框里面加入Rich factor这一列,Rich Factor的计算方式为差异表达的基因中位于该 pathway 条目的基因数目与所有有注释基因中位于该 pathway 条目的基因总数的比值。
pathway$richFactor<-pathway$Input.number/pathway$Background.number #添加richFactor列
kegg<-pathway[1:20,] #这里选择top20的KEGG terms来作图,由于数据框已经是按照Corrected.P.Value项从小到大排序过了,所以这里直接选择了前20项就行
2.ggplot2作图
1.将richFactor和Term映射到x轴和y轴
library(ggplot2)
colnames(kegg)
p<-ggplot(kegg,aes(x=richFactor,y=Term))
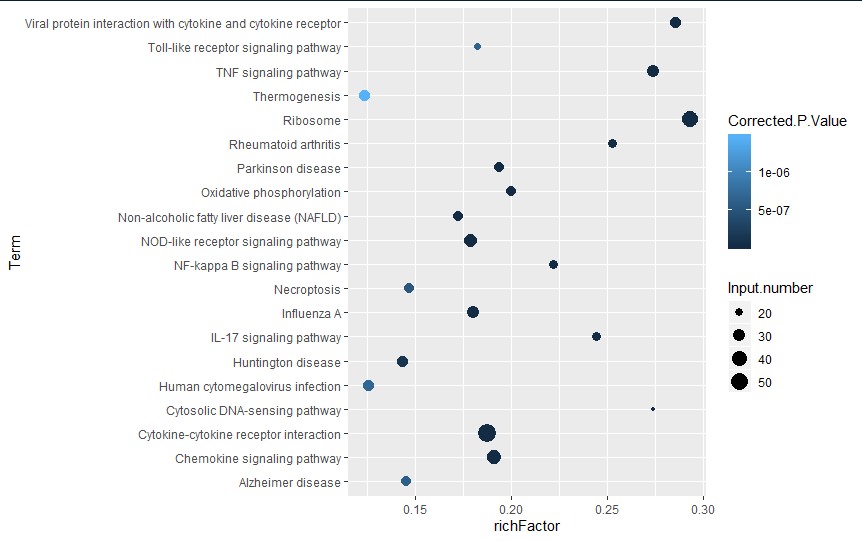
2.绘制散点图
p+geom_point() #绘制散点图
p+geom_point(aes(size=Input.number,color=Corrected.P.Value)) #将点的大小映射到Input.number,将颜色映射到Corrected.P.Value
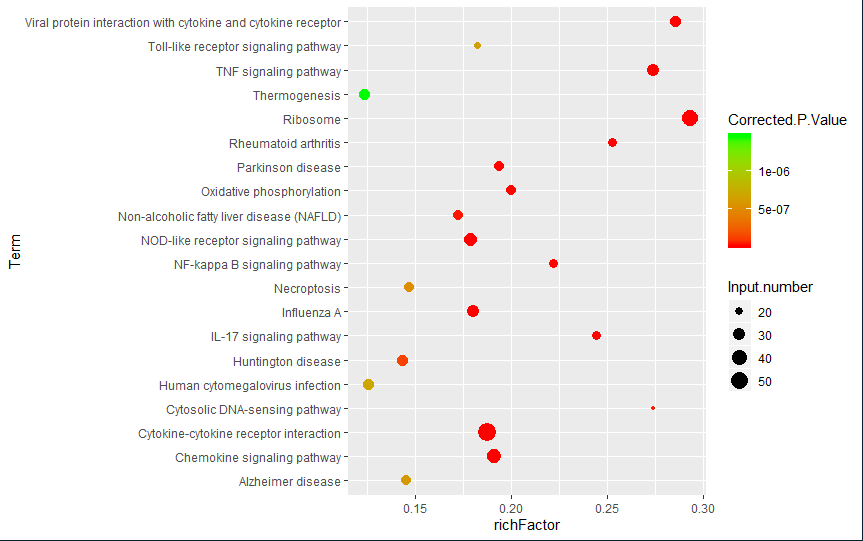
3.修改Rstudio的默认颜色
p+geom_point(aes(size=Input.number,color=Corrected.P.Value))+scale_color_gradient(low="red",high="green")
如果想修改x轴和y轴的label,比如将x轴的richFactor改成Rich factor,将y轴的Term去掉,再加上标题“Statistics of Pathway Enrichment",将Input.number改成Gene_number,将Corrected.P.Value改成qvalue,主题改成theme_bw,我们可以这样操作
p+geom_point(aes(size=Input.number,color=Corrected.P.Value))+scale_color_gradient(low="red",high="green")+labs(title="Statistics of Pathway Enrichment",x="Rich factor",y="",color="qvalue",size="Gene_number")+theme_bw()




