Novel LncRNA的实时定量PCR引物设计教程
Novel LncRNA的选择
novel LncRNA选择的原则:
1.不能选与其他基因exon重叠的lncRNA,否则,RT-qPCR结果就混合了两个基因的表达量。
2.最好选择基因间的lncRNA,即lincRNA,跟其他基因没有重叠。
3.其次选择位于其他基因intron区域的lncRNA。
4.再次,如果lncRNA的一部分与其他基因的exon重叠,另一部分不重叠,就选不重叠的那部分。
Novel LncRNA的引物设计
这里我们选取的novel LncRNA是LNC_000332,LNC_000332的fasta序列如下:
>LNC_000332:
TGGCAGCTTCAAGAACGAGTGATTTAAGAACAGCCCTCCCATCTTAGCAATGTCCCGGGGTGGCTGGAGCCACGGTCACTTCTTGGTCCTGGTCCAGAACTGTCGGTAGCGCTCCACATGCAAGTCATCACTGAGCTCCTGCTCGTACTCCTTCCTCGACTGGAGCTGAGCCTCACGCTTCCGCAGCCTGGCCCGGCGCTCCTCAGGAGACAGGGTGTCCAGATCTATGGGCATCGCCAGCATGCGCCGTGGCTCCCTGTAGCGGATGTGGATGGTGGAGCCATCCTGCTTCACCAGCAGCACGGGGTAGAGTCGTGCATAAGCCTGGCGGTGCACACGAGTGAGTGAGGCCCTGCTGCTGTCAGCTCGCCAGGAGGATGTGTGCAGGCGGCGGAGTGCAGGTCCGGTGGCCTTCACGGTGCTCTGCCTCAGCCGGCCAAGCAGGCTGCCCACGGCCGCCATTCCTCCTTACAGCCCCAGTGGCACGTGGAGGTGGTCAGTACAGGCCCTGGCGTGCAACTGCGTTGGGTGCTGCTGCGCTGCAGCCCACGACGTCACTGGCAGGCGCTGCGTCGCGCGGTATGACGTGTTCCATGGC
根据LncRNA的染色体位置相应位置的DNA序列
LNC_000332的染色体位置为:chr1:228106685-228109338,在染色体的负链上,我们通过UCSC来获取相应位置的DNA序列。UCSC的官网http://genome.ucsc.edu
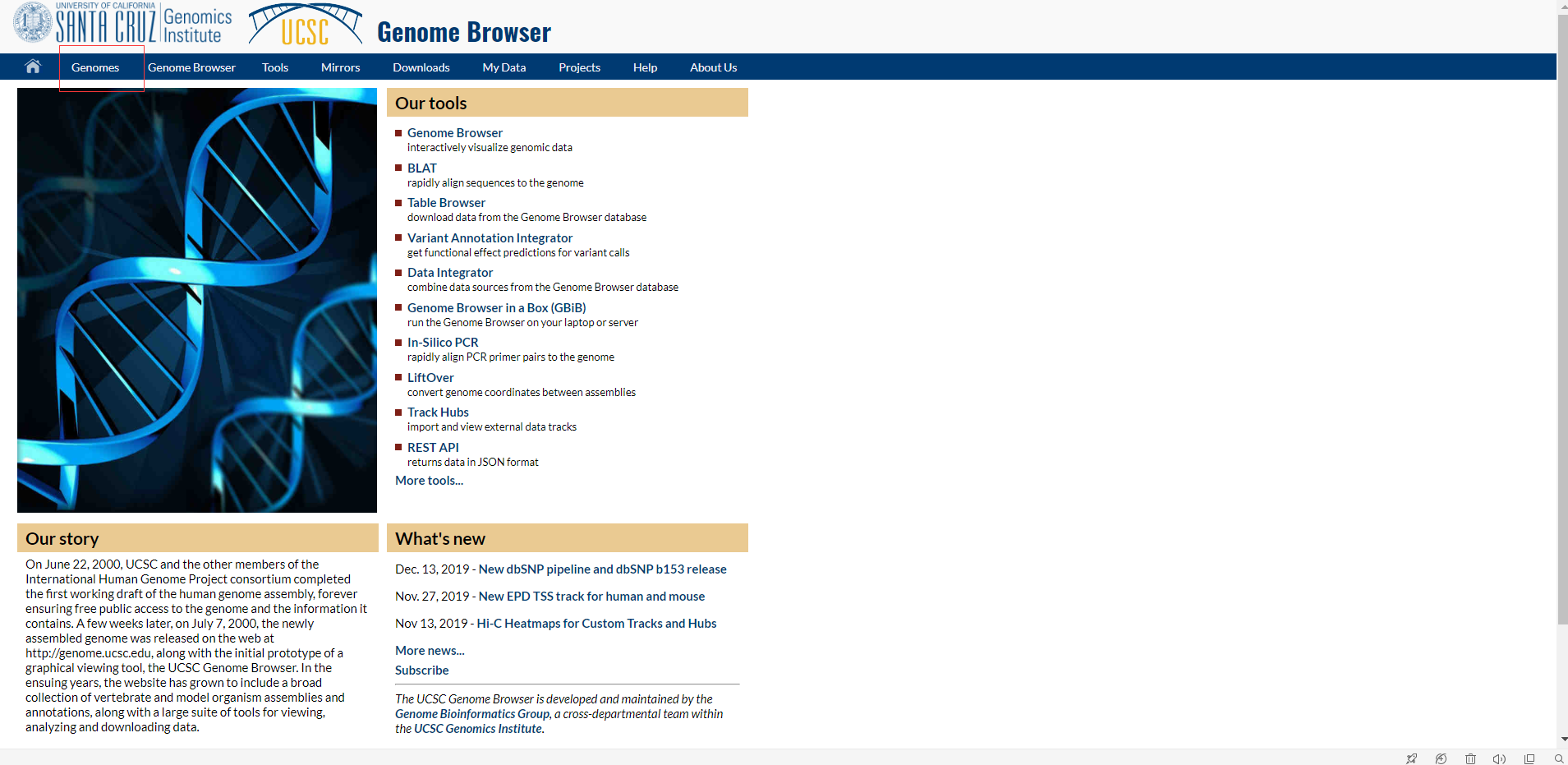
然后选择Genomes,进入后点击View,然后选择DNA
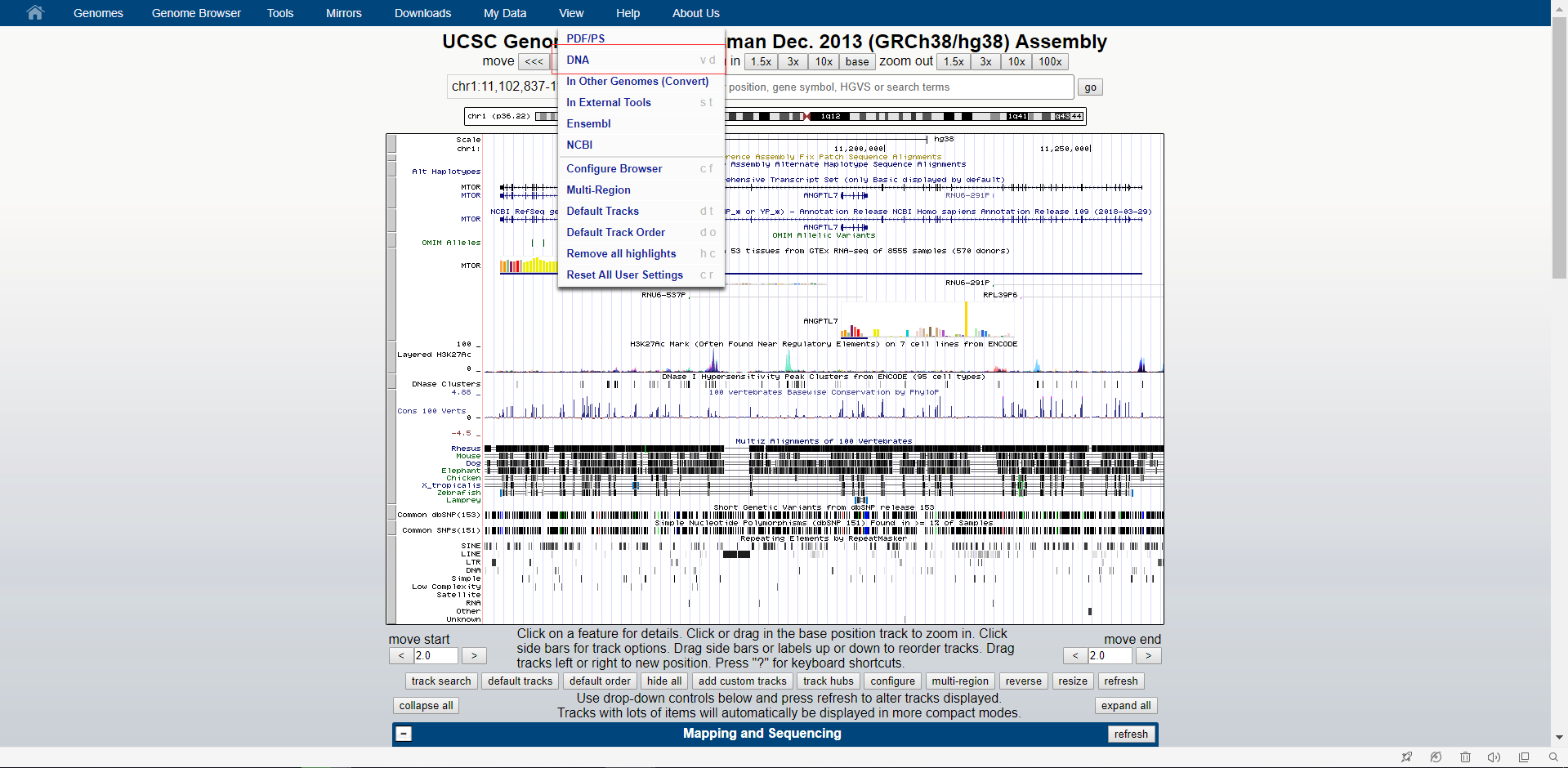

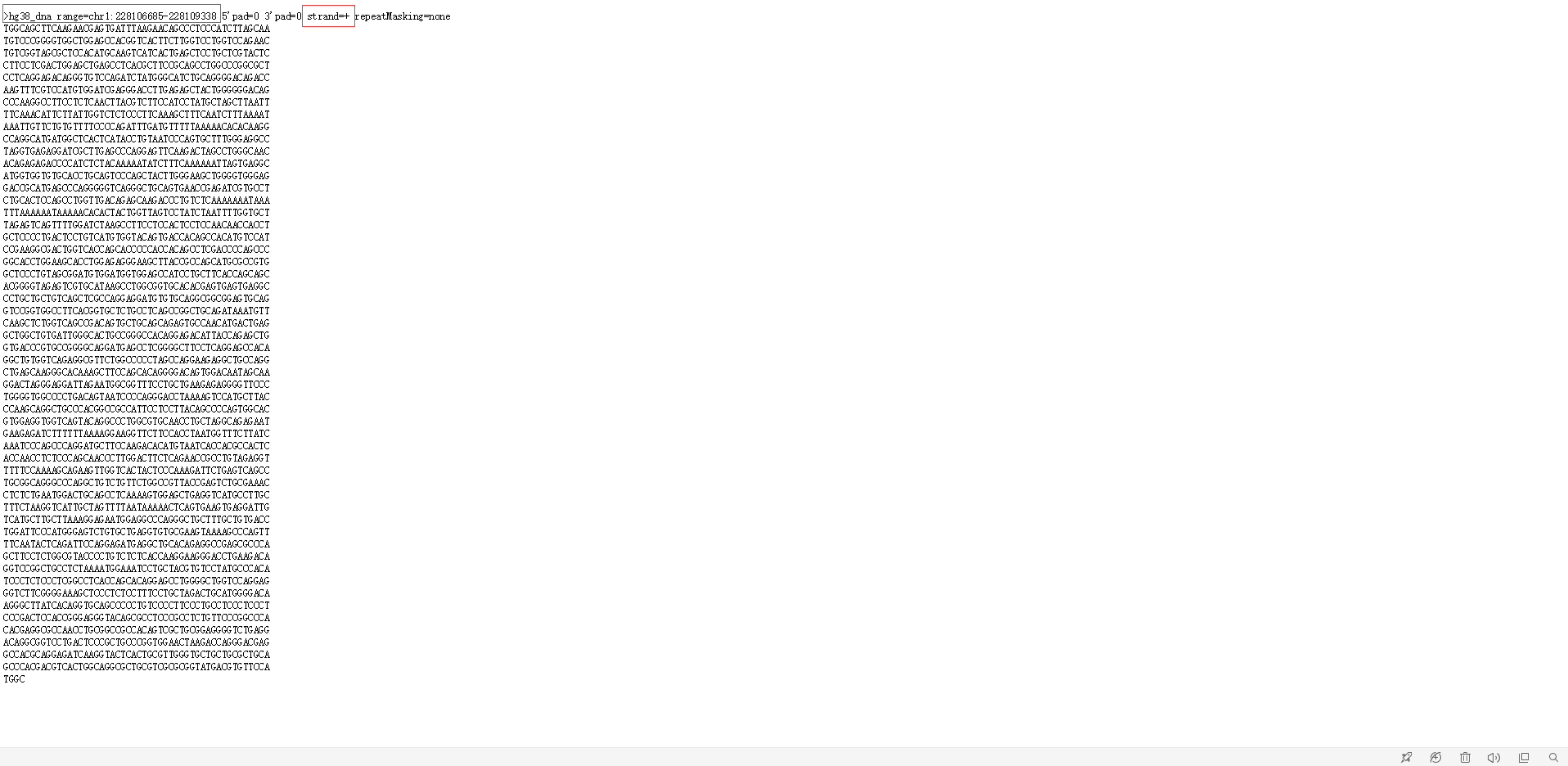
然后在上面的position输入基因的坐标,由于我们的坐标在DNA的负链,所以这里也要勾选Reverse complement,最后点击get DNA,就得到我们想要的DNA序列,下图为得到的DNA序列
通过AnnoLnc获取novel LncRNA的外显子坐标
AnnoLnc是北大生物信息中心开发出的一款分析novel LncRNA的网页工具,网址:http://annolnc.cbi.pku.edu.cn/index.jsp

我们输入LNC_000332的fasta序列,然后点击GO

信息很多,这里只截取一部分,可以看到LNC_000332包含4个外显子区域,不过这是hg19的染色体位置,我们需要用网页版的LiftOver来进行hg19和hg38的坐标转换,网址:http://genome.ucsc.edu/cgi-bin/hgLiftOver
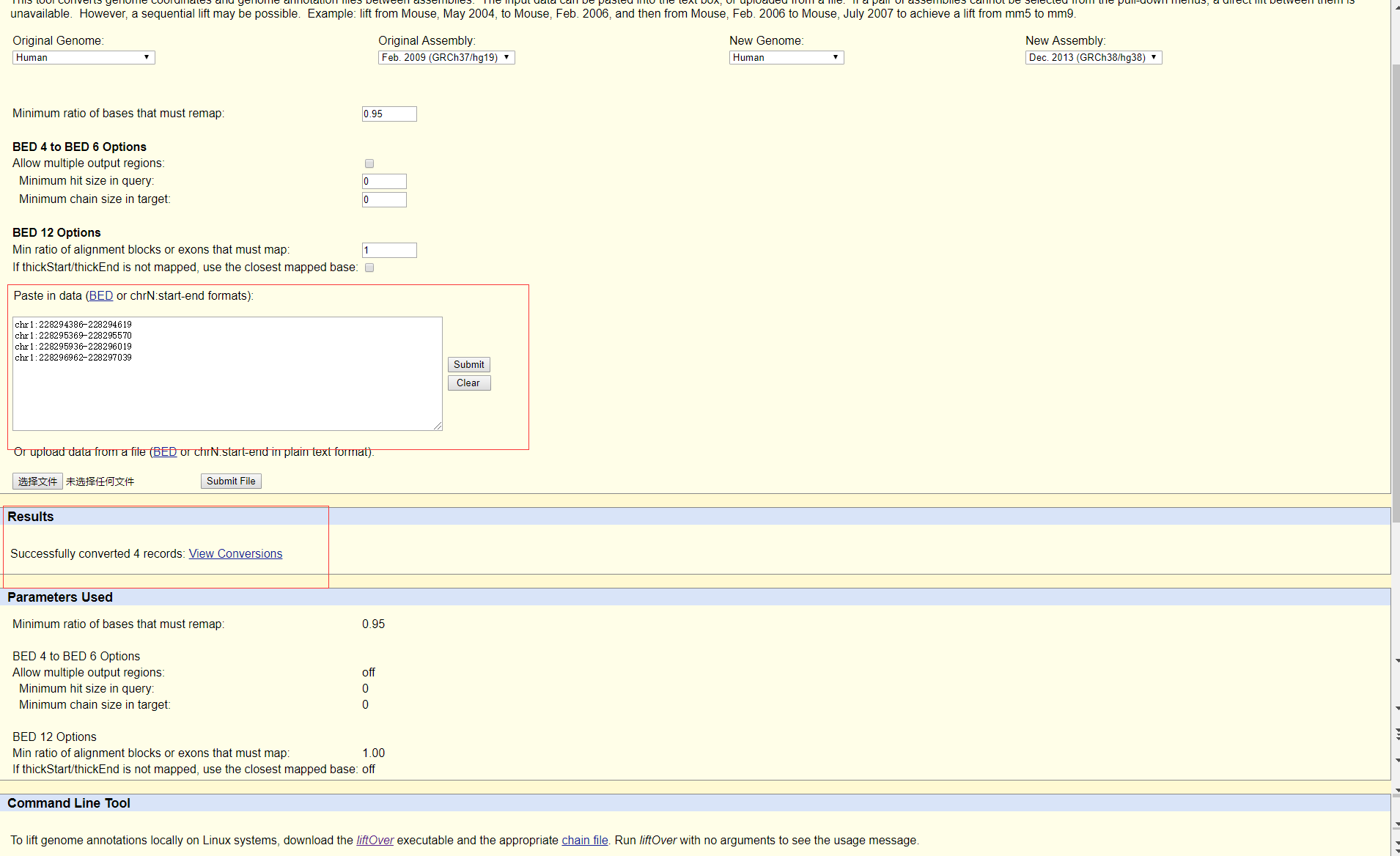
可以看到4个外显子坐标成功转换完成,点击View conversions就可以得到转换完成的hg38的外显子坐标,外显子坐标为:
chr1:228106685-228106918
chr1:228107668-228107869
chr1:228108235-228108318
chr1:228109261-228109338
将相应位置的DNA序列与LncRNA序列比对
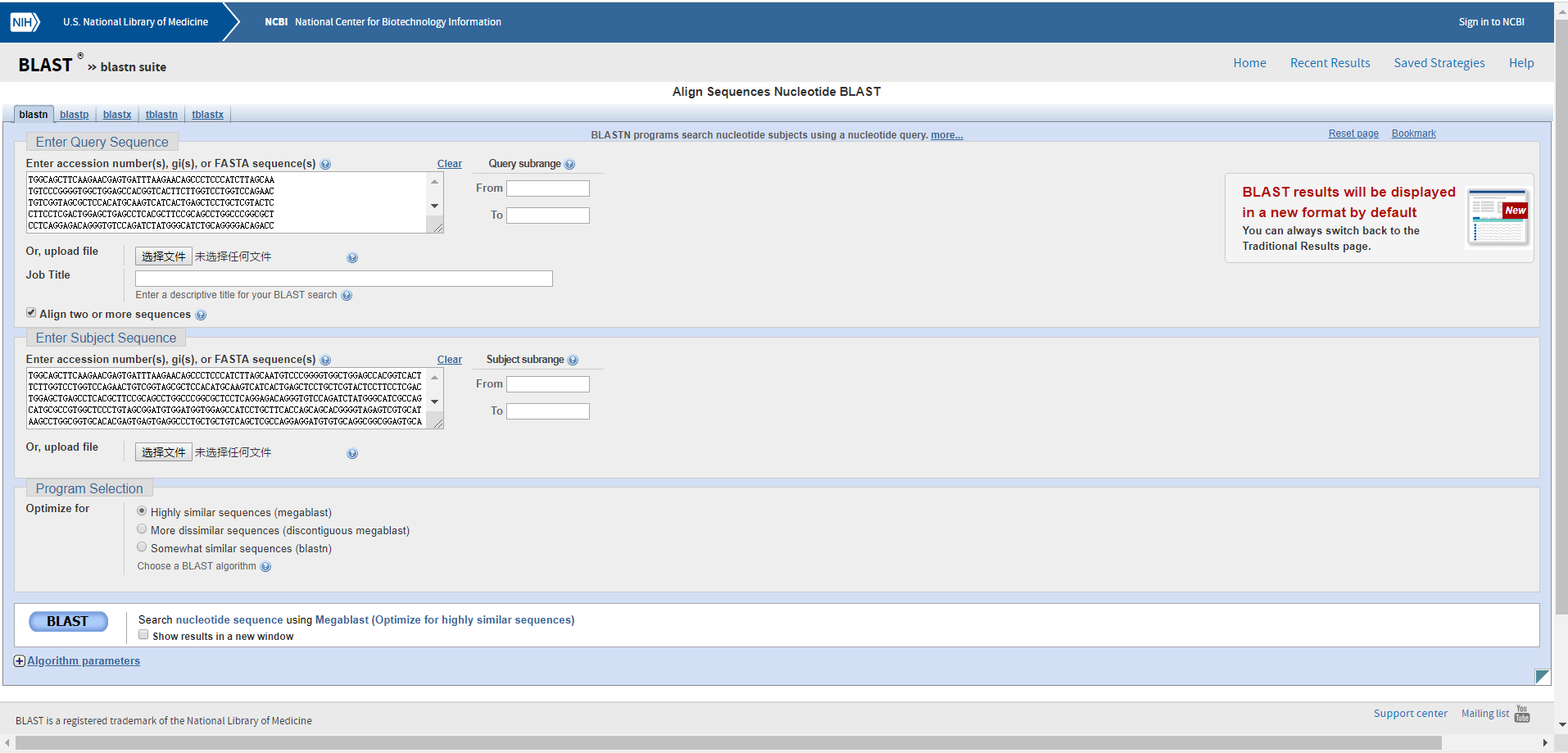

可以看到LNC_000332一共匹配了4段,染色体坐标分别为chr1:228106685-228106919,chr1:228107668-228107870,chr1:228108235-228108332,chr1:228109259-228109938
而外显子的染色体坐标分别为chr1:228106685-228106918,chr1:228107668-228107869,chr1:228108235-228108318,chr1:228109261-228109338
这里可以看到LNC_000332的序列基本都在外显子里面,所以不适合选择这个LncRNA来做PCR验证


