miRNA结合位点预测软件miRanda的使用教程
miRanda的介绍
miRanda 是由著名的 Memorial Sloan-Kettering 癌症研究中心的研究人员开发的,用于预测 miRNA 与其靶基因互作的结合位点。 miRanda 主要强调 miRNA 与靶基因连接位点的进化保守性,亦偏重于以 miRNA 的5′ 端序列搜索靶基因,并采用 RNAFold 计算热力学稳定性。
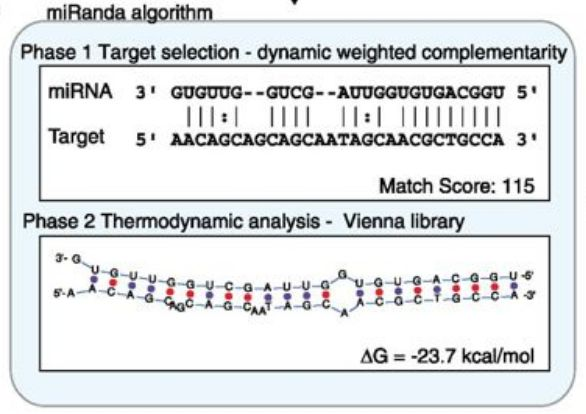
1.miRanda的下载与安装
1 wget http://cbio.mskcc.org/microrna_data/miRanda-aug2010.tar.gz 2 tar -xzvf miRanda-aug2010.tar.gz 3 cd /path/to/miRanda-3.3a4 ./configure --prefix=/path/to/miRandada-aug2010 #--prefix最好设置为当前文件夹的路径 5 make install
2.输入文件的准备
1.获取人类所有的miRNA的fasta文件hsa_miRNA.fa
进入miRBase的官网http://www.mirbase.org/
然后点击browse
然后点击expand all

然后选择Homo sapiens
然后再select sequence type这里选择 Mature sequence,接着点击select all,然后点击Fetch sequences就能得到人类所有成熟的miRNA的fasta文件了
2.获取差异的circRNA的fasta文件SFTSV_24vscontrol_circBase.fa
进入circBase的官网http://www.circbase.org/
然后点击list search
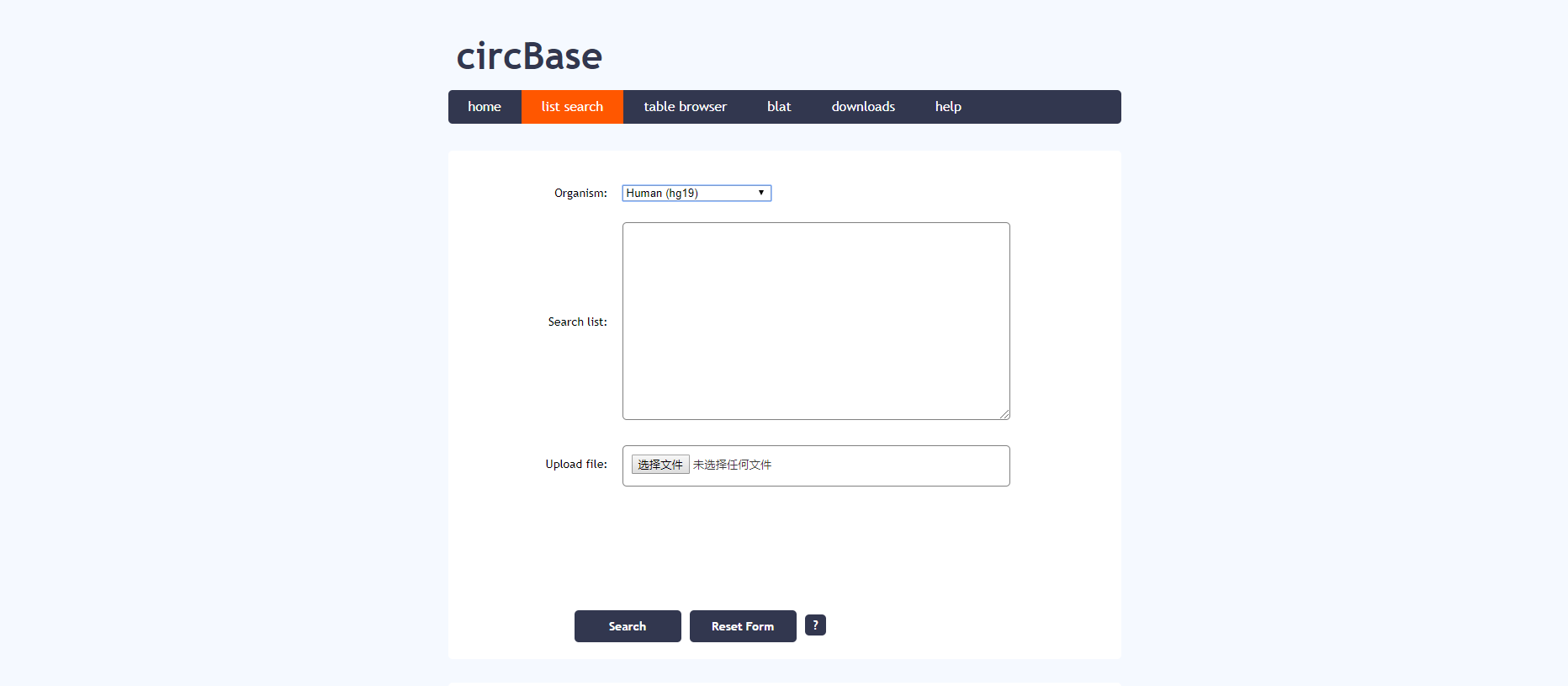
在organism选项选择Human (hg19),然后输入circBase的id,就可以得到相关circRNA的fasta文件了
3.miRanda的使用
miranda -sc 150 -en -7 hsa_miRNA.fa SFTSV_24vscontrol_circBase.fa >SFTSV_24vscontrol_circ_ miRNA_targeted_prediction.txt #-sc指定序列比对打分的阈值,小于该阈值的结合位点会被过滤掉;-en指定自由能的阈值,结果必须小于该阈值才可以
从中提取出关键信息
grep '>' SFTSV_24vscontrol_circ_miRNA_targeted_prediction.txt >SFTSV_24vscontrol_circ_miRNA_targeted_prediction_v1.txt
最后得到的文件即为circRNA有可能结合的靶miRNA,最后将结果输入cytoscape画ceRNA网络图。




