python语言绘图:绘制贝叶斯方法中最大后验密度(Highest Posterior Density, HPD)区间图的近似计算
代码源自:
https://github.com/PacktPublishing/Bayesian-Analysis-with-Python
===========================================================
贝叶斯方法中最大后验密度(Highest Posterior Density, HPD)区间,是指包含一定比例概率密度的最小区间。

主要判断所得的区间是否为HPD的依据为:

===========================================================
- 当概率分布为单峰分布时,如果分布为左右对称那么这个HPD是很好计算的,其中最典型的代表就是高斯分布。此时的HPD区间就等于频率学派的置信区间。
- 当概率分布为单峰分布时,如果分布为左右不是完全对称的那么这个HPD就很难计算,此时我们可以假设这个不对称的分布可以近似于一个对称的分布,其中比较典型的代表就是beta分布,不过这里需要知道既然是近似就一定存在偏差,而这个偏差随着不对称程度的增加而增加。既然我们这里把单峰不对称分布假设近似于单峰对称分布,自然我们采用的HPD方法类似于单峰对称分布。
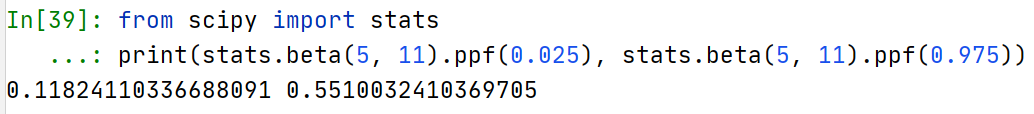
from scipy import stats print(stats.beta(5, 11).ppf(0.025), stats.beta(5, 11).ppf(0.975))
import matplotlib.pyplot as plt import numpy as np from scipy import stats import seaborn as sns palette = 'muted' sns.set_palette(palette); sns.set_color_codes(palette) def naive_hpd(post): sns.kdeplot(post) HPD = np.percentile(post, [2.5, 97.5]) plt.plot(HPD, [0, 0], label='HPD {:.2f} {:.2f}'.format(*HPD), linewidth=8, color='k') plt.legend(fontsize=16); plt.xlabel(r"$\theta$", fontsize=14) plt.gca().axes.get_yaxis().set_ticks([]) np.random.seed(1) post = stats.beta.rvs(5, 11, size=1000) naive_hpd(post) plt.xlim(0, 1) plt.savefig('B04958_01_07.png', dpi=300, figsize=(5.5, 5.5)) plt.show()
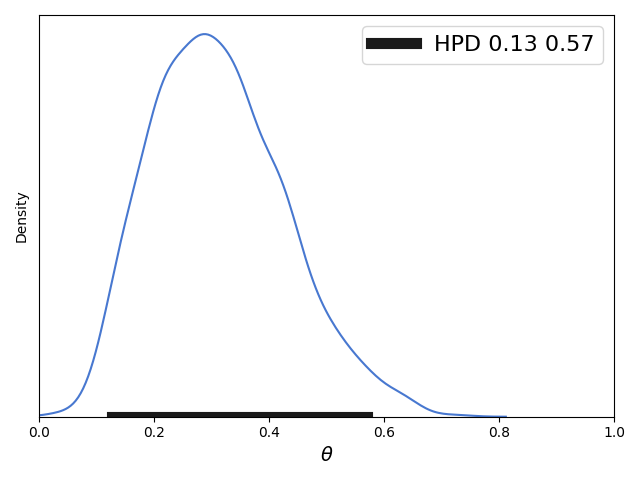
可以看到此时的区间[0.13, 0.57]距离[0.11824110336688091, 0.5510032410369705]还是存在一点偏差的,而这个偏差随着蒙特卡洛采样数据量的增加而减少,对此尝试代码如下:
import numpy as np from scipy import stats import seaborn as sns palette = 'muted' sns.set_palette(palette); sns.set_color_codes(palette) np.random.seed(1) for n in [10**3, 10**4, 10**5, 10**6, 10**7, 10**8, 10**9,]: post = stats.beta.rvs(5, 11, size=n) HPD = np.percentile(post, [2.5, 97.5]) print(HPD) print(stats.beta(5,11).cdf(HPD[0]), stats.beta(5,11).cdf(HPD[1]))
运行结果:

可以看到累积密度函数最终贴近于[0.025, 0.975]。
from scipy import stats import seaborn as sns palette = 'muted' sns.set_palette(palette); sns.set_color_codes(palette) a_per = 0.025 b_per = 0.975 ab_per = 0.95 alpha = 5 beta = 11 a = stats.beta(alpha, beta).ppf(a_per) b = stats.beta(alpha, beta).ppf(b_per) for i in range(10**8): a_p = stats.beta(alpha, beta).pdf(a) b_p = stats.beta(alpha, beta).pdf(b) print("{:6d} \t {:8f} {:8f} \t\t {:8f} {:8f}".format(i, a, b, a_p, b_p)) if abs(a_p - b_p)<0.0001: break if a_p > b_p: a = a - 0.000001 else: a = a + 0.000001 b = stats.beta(alpha, beta).ppf( stats.beta(alpha, beta).cdf(a)+ab_per )
运行结果:
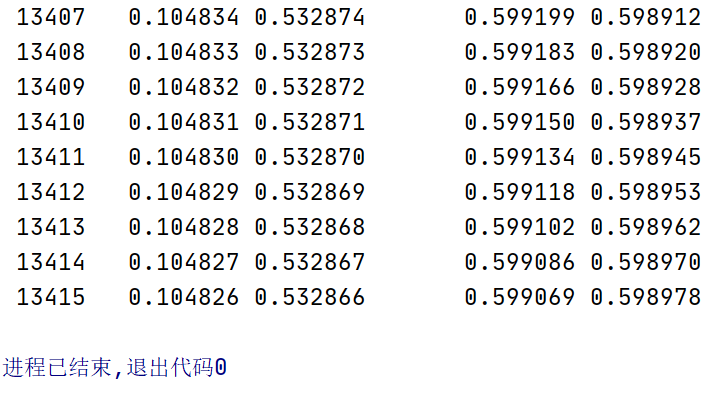
通过运行结果我们得到最后的区间为:
[0.104826, 0.532866]
可以看到这个最优的区间结果与第一种方法的结果还是有一定差距的。
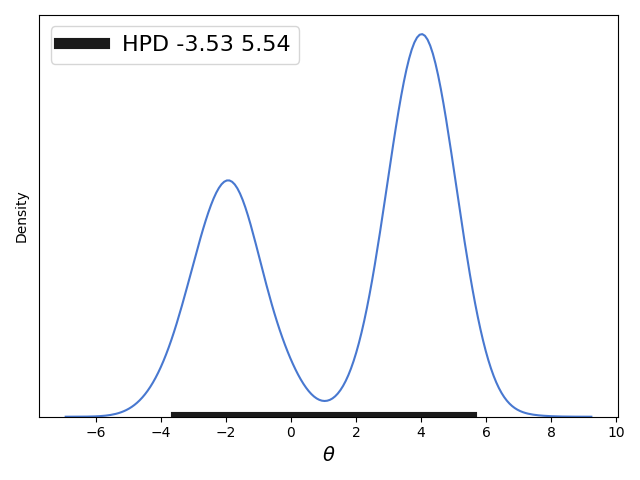
- 当概率分布为双峰分布时,并且HPD的区间包括在双峰的大部分并且双峰的外侧分布相似那么上面的第二种近似求解HPD区间的方式同样适用,但是其他的近似求解方式(第一种和第三种)已经不适应了,因为对于很多双峰分布我们是难以写出具体的概率密度函数rvs、累积分布函数cdf、累积分布函数的反函数ppf的。给出相应代码:
import matplotlib.pyplot as plt import numpy as np from scipy import stats import seaborn as sns palette = 'muted' sns.set_palette(palette); sns.set_color_codes(palette) def naive_hpd(post): sns.kdeplot(post) HPD = np.percentile(post, [2.5, 97.5]) plt.plot(HPD, [0, 0], label='HPD {:.2f} {:.2f}'.format(*HPD), linewidth=8, color='k') plt.legend(fontsize=16); plt.xlabel(r"$\theta$", fontsize=14) plt.gca().axes.get_yaxis().set_ticks([]) np.random.seed(1) gauss_a = stats.norm.rvs(loc=4, scale=0.9, size=3000) gauss_b = stats.norm.rvs(loc=-2, scale=1, size=2000) mix_norm = np.concatenate((gauss_a, gauss_b)) naive_hpd(mix_norm) plt.savefig('B04958_01_08.png', dpi=300, figsize=(5.5, 5.5)) plt.show()
注意:上面代码是存在一定问题的,两个高斯函数的采样并没有使用相同的采样数,这里建议进行修改,如:
gauss_a = stats.norm.rvs(loc=4, scale=0.9, size=30000)
gauss_b = stats.norm.rvs(loc=-2, scale=1, size=30000)
posted on 2022-06-10 15:37 Angry_Panda 阅读(774) 评论(0) 编辑 收藏 举报















【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 无需6万激活码!GitHub神秘组织3小时极速复刻Manus,手把手教你使用OpenManus搭建本
· Manus爆火,是硬核还是营销?
· 终于写完轮子一部分:tcp代理 了,记录一下
· 别再用vector<bool>了!Google高级工程师:这可能是STL最大的设计失误
· 单元测试从入门到精通
2021-06-10 【转载】 在边缘设备上拟合大型神经网络的方法总结