医学图像处理(一):医学图像格式(nii和dicom)
医疗图像是对解剖区域的内部结构和功能的一种表征。它以二维像素或者三维体素的形式呈现出来。映射到空间位置的数值是对采样过程和重建过程的离散表征。用来描述一个确定采样模态视野的像素数量是对解剖结构和功能的细节的表达。像素表达的数值取决于成像模式、采样协议、重建以及后续处理过程。
医疗数据的组成
医疗数据有四个关键的组成部分--像素深度、光度解释、元数据以及像素数据。这几部分决定了图像的大小和分辨率
a. 像素深度(Pixel Depth)或者位深度(Bit Depth)或者色深度(Color Depth)就是用来编码每一像素的信息所用的位数。例如,一个 8 位的栅格会拥有从 0 到 255 这 256 种各不相同的数值。
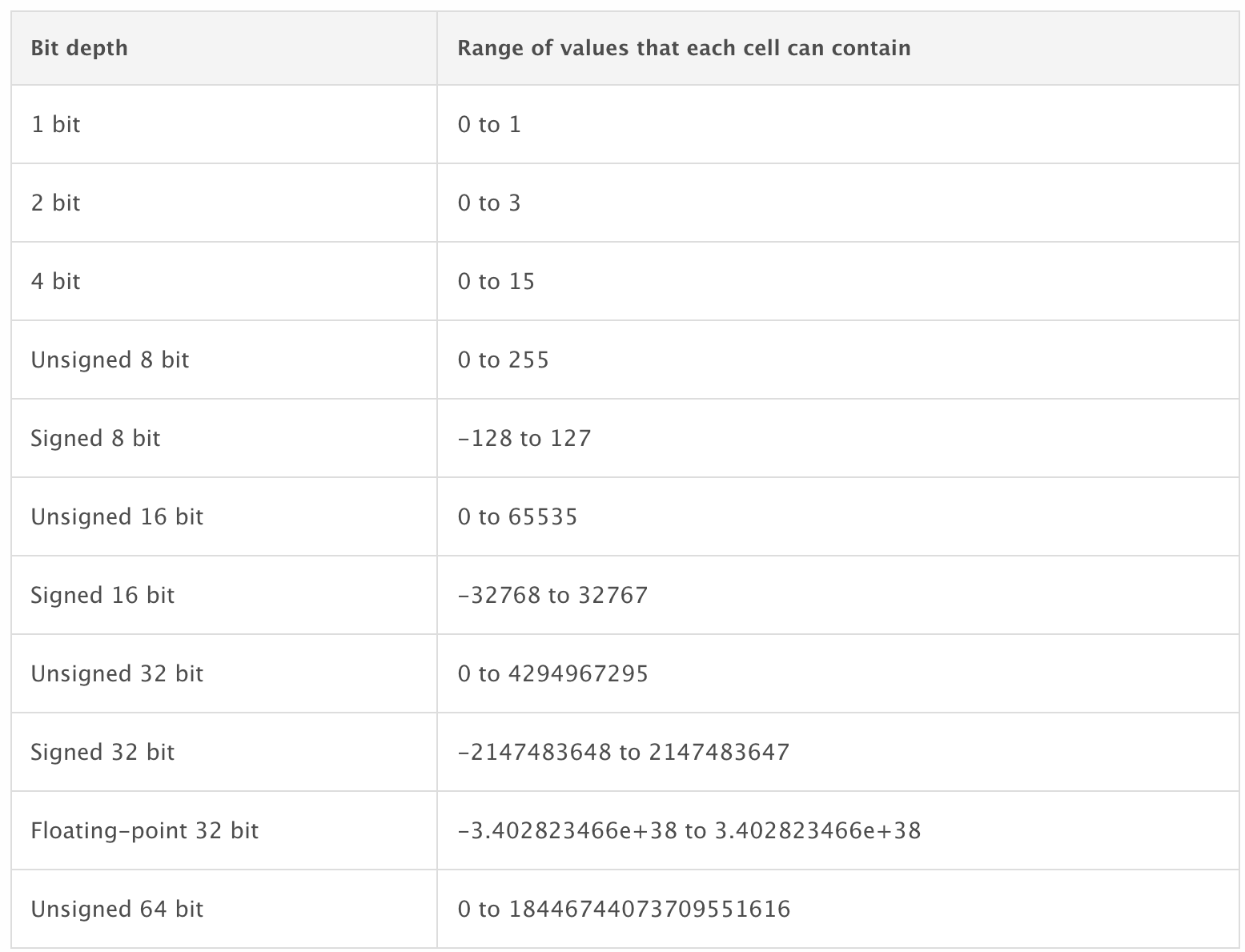
b. 光度解释具体化了像素数据被解释成正确的图片展示的方式,如单色图像或者彩色图像。为了确定像素值中是否存储了彩色信息,我们引入了每个像素的样本的概念,也就是大家都知道的通道数量。单色图像每个像素只有一个样本,图片中并没有存储彩色信息。我们使用从黑色到白色的灰度级别来展示这种图片。灰度的数量明显取决于用来存储这个样本的位数,在这种情况下,与像素深度是一致的。像 X 光片、CT和磁共振这样的放射医疗影像都有一个灰度光度解释。核医学图像都以彩色的形式展现,例如 PET 和 SPECT。
c. 元数据就是图片中所描述的信息。它看上去可能是很奇怪的,但是无论在什么格式的文件中,都存在一些超越像素数据并且和图像相关的信息。这类被称作元数据的信息通常都以头部的形式储在文件的起始部分,它至少会包含以下信息:图像矩阵的维度、空间精度、像素深度以及光度解释。
d. 像素数据--这里存储的是像素数值的大小。根据不同的数据类型,像素数据可能以整型或者浮点型的类型存储,使用表达数据所需的最少的数据位。
所以,图像的大小=头部大小(包含元数据)+行×列×像素深度×帧的数量
医疗图像的格式
放射生物图像中主要有六种格式--DICOM(医疗中的数字图像和通信),NIFTI(神经影像学信息技术计划),PAR/REC(飞利浦 MRI 扫描格式),ANALYZE(Mayo 医疗成像)以及 NRRD(近乎原始光栅数据)和 MNIC 格式。
其中 DICOM 和 NIFTI 是最常用的格式。
DICOM 格式的基本知识
DICOM 代表的是医疗数字成像和通信。DICOM 是由美国国家电气制造商协会(NEMA)制定的标准。它定义了医疗成像领域中关于信息处理、存储、打印以及传输的标准。这些都是你在扫描仪或者某家医院的图片归档和通信系统(PACS)能够立即得到的文件格式。
它包括了文件格式和能够接收图像和 DICOM 格式的病人数据的实体之间使用 TCP/IP 进行通信的协议。
一个 DICOM 文件包含文件头部和同文件名的*.dcm 图像数据。文件头部的大小取决于它所提供的信息的多少。文件头包含以下信息:病人的 ID,病人的姓名,图像的模态以及其他信息。它定义了帧的数量以及图像的精度。这些信息会被图像浏览器在显示图像时用到。对于一个单词采样,会有很多个 DICOM 文件。
pydicom 是一个读取 dicom 文件的 python 库
NIFTI 格式基本知识
Nifti 格式最初是为神经影像学发明的。神经影像信息学技术计划(NIFTI)将 NIfTI 格式预设为 ANALYZE7.5 格式的替代品。它最初的应用领域是神经影像,但是也被用在其他领域。这种格式的主要特点就是它包含两个能够将每个体素的索引(i,j,k)和它的空间位置(x,y,z)关联起来的仿射坐标。
DICOM 和 NIFTI 的区别
DICOM 和 NIfTI 这两种格式的主要区别是:NIfTI 中的图像原始数据被存储成了 3 维图像,而 dicom 一些 2 维的图层。这就使得 NIFTI 更加适合那些应用在 DICOM 上的机器学习的方法,因为它是以 3D 图像建模的。处理一个单独的 NIFTI 文件要比处理成百上千个 dicom 文件更加容易一些。与 DICOM 格式下的好多个文件相比,NIFTI 格式下,每个 3d 图像只有两个文件。
格式转换
dicom 转换成 NIFTI
dicom2nii是一个用来把 DICOM 转换为 NIFTI 的工具。nibabel 是一个读写 nifiti 文件的 python 库。如果你你想把 DICOM 转换成 NIFTI,可以使用自动转换的工具(例如,dcm2nii)。python2 下的库 dcmstack 可以将一系列的 DICOM 文件堆叠成多维数组。这些数组能够被写成 NIFTI 的文件,同时还加上一个可选的头部扩展,这个头文件包含原始 DICOM 文件的所有元数据。python3 提供了一个新的用来完成这个格式转换的库--dicom2nifti。我建议读者去查看一下 nipy 这个项目。
也可以使用python读取dicom文件后转换为nii文件
其他格式相关内容可查看引用文章
nii详解
nii基础
大部分医学领域导出dicom格式,但是太复杂了。很多时候,将dicom转换为nifti格式也就是nii格式
一个NIFTI格式主要包含三部分:hdr, ext, img

hdr/header
这部分数据长度是固定的,当然不同版本可能规定的长度不同,但是同一版本的多个nii文件是相同的。
header里包含的信息有:
--维度,x,y,z,单位是毫米。还有第四个维度,就是时间。这部分储存的主要是四个数字。
--voxel size(体素大小):毫米单位的x,y,z大小。(也就是spacing)
--数据类型,一般是int16,这个精度不够,最好使用double类型。
--Form和转换矩阵,每一个Form都对应一个转换矩阵。(暂时不知道Form是什么)
Extension
是自己可以随意定义数据的部分,可以自己用。但是通用的软件公司都无法使用这部分。
Image
储存3D或者4D的图像数据
坐标
dicom和nii格式定义了不同的方向,对于nii格式,坐标原点在大脑中某个部位上,方向可以从图上看出。
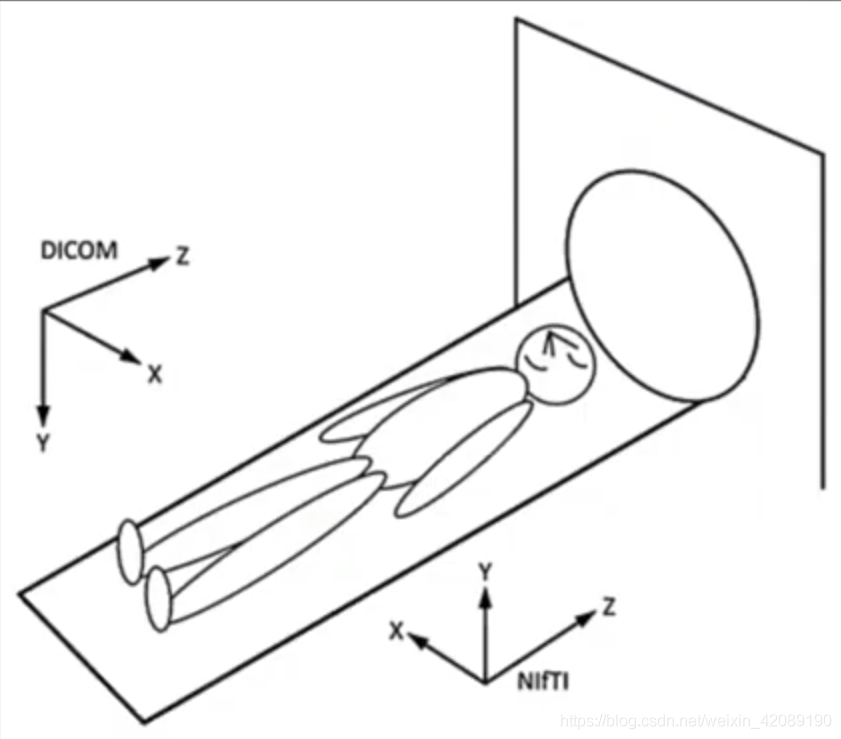
体素
转换矩阵
转换矩阵可以轻松分清图像的左右。转换矩阵是一个4X4的矩阵,作用是将体素索引(i,j,k)转换为空间位置(x,y,z)。具体使用方法是转换矩阵乘以一个包含(i,j,k)的矩阵,就可以得到一个包含(x,y,z)的矩阵。
转换矩阵用到了一些概念,在dicom格式上也是一样的
详情见引用文章
nii格式
后缀名为.nii的文件格式又叫NIfTI-1,它改编自广泛使用的ANALYZE™7.5格式。一些比NIfTI-1发展早的老软件也可以兼容NIfTI-1。
比ANALYZE 7.5新的特性如下:
--将体素索引(i,j,k)与空间位置(x,y,z)相关的仿射坐标定义
--表示FMRI(核磁共振)的时空切片顺序的代码;
--“完整”的8-128位数据类型集;
--在1-4维域上存储矢量值数据集的标准化方法;
--表示数据“含义”的代码;
--向标头添加“扩展”数据的标准化方法;
--双文件(.hdr和.img)或单文件(.nii)存储;
还有很多。 目的是在FMRI数据分析软件包之间的文件交换级别上促进互操作性。
AFNI,BrainVoyager,FSL和SPM的作者都致力于支持这种格式的输入和输出。
注意:该规范现在称为NIfTI-1.1。



