作业七 逻辑回归应用
1.逻辑回归是怎么防止过拟合的?为什么正则化可以防止过拟合?(大家用自己的话介绍下)
答:如果你的目标是为测试数据表上0,1两种标签。我们可以用一个线性函数h(x)来分割这个空间,一边的是良性的,一边是恶性的。由于最右边一个奇元的instance影响,我们得到的直线很可能是这样的,对于许多训练集的instance来说都分到了错误的一边。用linear model来做分类并不一定是个好主意。我们就有了新的hypothesis,这个的输出在(0,1)之间,当h’(x)>0.5时,我们认为肿瘤是恶性的(1),当h’(x)<0.5时为良性的。当h’(x)=0.5时结果random一个。拿上面这个作为最小化的目标,用gradient descent求得使它最小的ω’,这个就是最后训练出来的模型的参数了。对于新的数据x’,我们用新的f(x’)=h’(x’, ω’),如果f(x’)>0.5,预测结果就是恶性的;如果f(x’)<0.5,预测结果就是良性的。同时这个函数值也是结果是恶性的概率。
答:简单来说就是:以L2正则化为例,正则项会使权重趋于0,就意味着大量权重就和0没什么差别了,此时网络就变得很简单,拟合能力变弱,从高方差往高偏差的方向移动。激活函数的角度讲:以sigmoid或tanh为例,当w趋于0时(忽略偏置b),激活值趋于0,此时位于激活函数的线性趋于,神经网络就变成一个线性网络,不容易过拟合。
2.用logiftic回归来进行实践操作,数据不限。
import pandas as pd
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn.preprocessing import StandardScaler
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import classification_report
def logistic():
#加载数据集
names = ['Sample code number',' Clump Thickness','Uniformity of Cell Size','Uniformity of Cell Shape',
'Marginal Adhesion','Single Epithelial Cell Size','Bare Nuclei','Bland Chromatin',
'Normal Nucleoli','Mitoses','Class']
data = pd.read_csv('breast-cancer-wisconsin_3.csv',names=names)
#数据集预处理,缺失值删除
data = data.replace(to_replace='?',value=np.nan)
data = data.dropna()
#进行数据的分割
x_train,x_test,y_train,y_test = train_test_split(data.loc[:,'Sample code number':'Mitoses'],data.loc[:,'Class'],test_size=0.25)
#特征值的标准化
std = StandardScaler()
x_train = std.fit_transform(x_train)
x_test = std.transform(x_test)
#使用逻辑回归进行预测
lr = LogisticRegression(C=1.0)
lr.fit(x_train,y_train)
print(lr.coef_)
y_predict = lr.predict(x_test)
#输出准确率
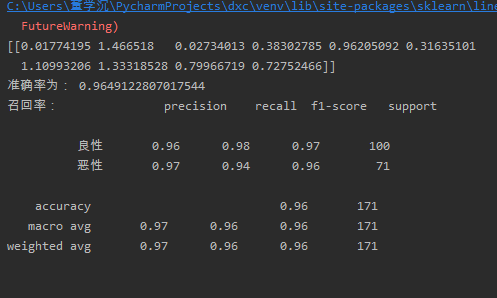
print("准确率为:",lr.score(x_test,y_test))
#输出召回率
print("召回率:",classification_report(y_test,y_predict,labels=[2,4],target_names=["良性","恶性"]))
# print(x_train)
return None
if __name__ == "__main__":
logistic()
截图: