【R笔记】glm函数报错原因及解析
R语言glm函数学习:
【转载时请注明来源】:http://www.cnblogs.com/runner-ljt/
Ljt
作为一个初学者,水平有限,欢迎交流指正。
glm函数介绍:
glm(formula, family=family.generator, data,control = list(...))
family:每一种响应分布(指数分布族)允许各种关联函数将均值和线性预测器关联起来。
常用的family:
binomal(link='logit') ----响应变量服从二项分布,连接函数为logit,即logistic回归
binomal(link='probit') ----响应变量服从二项分布,连接函数为probit
poisson(link='identity') ----响应变量服从泊松分布,即泊松回归
control:控制算法误差和最大迭代次数
glm.control(epsilon = 1e-8, maxit = 25, trace = FALSE)
-----maxit:算法最大迭代次数,改变最大迭代次数:control=list(maxit=100)
glm函数使用:
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
|
>> data<-iris[1:100,]> samp<-sample(100,80)> names(data)<-c('sl','sw','pl','pw','species')> testdata<-data[samp,]> traindata<-data[-samp,]>> lgst<-glm(testdata$species~pl,binomial(link='logit'),data=testdata)Warning messages:1: glm.fit:算法没有聚合2: glm.fit:拟合機率算出来是数值零或一> summary(lgst)Call:glm(formula = testdata$species ~ pl, family = binomial(link = "logit"), data = testdata)Deviance Residuals: Min 1Q Median 3Q Max -1.836e-05 -2.110e-08 -2.110e-08 2.110e-08 1.915e-05 Coefficients: Estimate Std. Error z value Pr(>|z|)(Intercept) -83.47 88795.25 -0.001 0.999pl 32.09 32635.99 0.001 0.999(Dispersion parameter for binomial family taken to be 1) Null deviance: 1.1085e+02 on 79 degrees of freedomResidual deviance: 1.4102e-09 on 78 degrees of freedomAIC: 4Number of Fisher Scoring iterations: 25> |
注意在使用glm函数就行logistic回归时,出现警告:
Warning messages:
1: glm.fit:算法没有聚合
2: glm.fit:拟合機率算出来是数值零或一
同时也可以发现两个系数的P值都为0.999,说明回归系数不显著。
第一个警告:算法不收敛。
由于在进行logistic回归时,依照极大似然估计原则进行迭代求解回归系数,glm函数默认的最大迭代次数 maxit=25,当数据不太好时,经过25次迭代可能算法 还不收敛,所以可以通过增大迭代次数尝试解决算法不收敛的问题。但是当增大迭代次数后算法仍然不收敛,此时数据就是真的不好了,需要对数据进行奇异值检验等进一步的处理。
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
|
>> lgst<-glm(testdata$species~pl,binomial(link='logit'),data=testdata,control=list(maxit=100))Warning message:glm.fit:拟合機率算出来是数值零或一> summary(lgst)Call:glm(formula = testdata$species ~ pl, family = binomial(link = "logit"), data = testdata, control = list(maxit = 100))Deviance Residuals: Min 1Q Median 3Q Max -1.114e-05 -2.110e-08 -2.110e-08 2.110e-08 1.162e-05 Coefficients: Estimate Std. Error z value Pr(>|z|)(Intercept) -87.18 146399.32 -0.001 1pl 33.52 53808.49 0.001 1(Dispersion parameter for binomial family taken to be 1) Null deviance: 1.1085e+02 on 79 degrees of freedomResidual deviance: 5.1817e-10 on 78 degrees of freedomAIC: 4Number of Fisher Scoring iterations: 26> |
如上,通过增加迭代次数,解决了第一个警告,此时算法收敛。
但是第二个警告仍然存在,且回归系数P=1,仍然不显著。
第二个警告:拟合概率算出来的概率为0或1
首先,这个警告是什么意思?
我们先来看看训练样本的logist回归结果,拟合出的每个样本属于'setosa'类的概率为多少?
|
1
2
3
4
5
|
>>lgst<-glm(testdata$species~pl,binomial(link='logit'),data=testdata,control=list(maxit=100))>p<-predict(lgst,type='response')>plot(seq(-2,2,length=80),sort(p),col='blue')> |

可以看出训练样本为'setosa'类的概率不是几乎为0,就是几乎为1,并不是我们预想中的logistic模型的S型曲线,这就是第二个警告的意思。
那么问题来了,为什么会出现这种情况?
(以下内容只是本人参考一些解释的个人理解)
这种情况的出现可以理解为一种过拟合,由于数据的原因,在回归系数的优化搜索过程中,使得分类的种类属于某一种类(y=1)的线性拟合值趋于大,分类种类为另一 类(y=0)的线性拟合值趋于小。
由于在求解回归系数时,使用的是极大似然估计的原理,即回归系数在搜索过程中使得似然函数极大化:
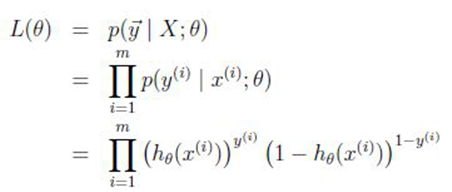
所以在搜索过程中偏向于使得y=1的h(x)趋向于大,而使得y=0的h(x)趋向于小。
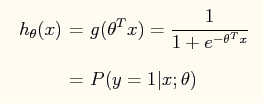
即系数Θ使得 Y=1类的 -ΘTX 趋向于大,使得Y=0类的 -ΘTX 趋向于小。而这样的结果就会导致P(y=1|x;Θ)-->1 ; P(y=0|x;Θ)-->0 .
那么问题又来了,什么样的数据会导致这样的过拟合产生呢?
先来看看上述logistic回归中种类为setosa和versicolor的样本pl值的情况。(横轴代表pl值,为了避免样本pl数据点叠加在一起,增加了一个无关的y值使样本点展开)

可以看出两类数据明显的完全线性可分。
故在回归系数搜索过程中只要使得一元线性函数h(x)的斜率的绝对值偏大,就可以实现y=1类的h(x)趋向大,y=0类的h(x)趋向小。
所以当样本数据完全可分时,logistic回归往往会导致过拟合的问题,即出现第二个警告:拟合概率算出来的概率为0或1。
出现了第二个警告后的logistic模型进行预测时往往是不适用的,对于这种线性可分的样本数据,其实直接使用规则判断的方法则简单且适用(如当pl<2.5时则直接判断为setosa类,pl>2.5时判断为versicolor类)。
以下,对于不完全可分的二维训练数据展示logistic回归过程。
|
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
|
>> data<-iris[51:150,]> samp<-sample(100,80)> names(data)<-c('sl','sw','pl','pw','species')> testdata<-data[samp,]> traindata<-data[-samp,]>> lgst<-glm(testdata$species~sw+pw,binomial(link='logit'),data=testdata)> summary(lgst)Call:glm(formula = testdata$species ~ sw + pw, family = binomial(link = "logit"), data = testdata)Deviance Residuals: Min 1Q Median 3Q Max -1.82733 -0.16423 0.00429 0.11512 2.12846 Coefficients: Estimate Std. Error z value Pr(>|z|) (Intercept) -12.915 5.021 -2.572 0.0101 * sw -3.796 1.760 -2.156 0.0310 * pw 14.735 3.642 4.046 5.21e-05 ***---Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1(Dispersion parameter for binomial family taken to be 1) Null deviance: 110.85 on 79 degrees of freedomResidual deviance: 24.40 on 77 degrees of freedomAIC: 30.4Number of Fisher Scoring iterations: 7>#画拟合概率曲线图> p<-predict(lgst,type='response')> plot(seq(-2,2,length=80),sort(p),col='blue')>>#画训练样本数据散点图>a<-testdata$species=='versicolor'> x1<-testdata[a,'sw']> y1<-testdata[a,'pw']> x2<-testdata[!a,'sw']> y2<-testdata[!a,'pw']> summary(testdata$sw) Min. 1st Qu. Median Mean 3rd Qu. Max. 2.000 2.700 2.900 2.881 3.100 3.800> summary(testdata$pw) Min. 1st Qu. Median Mean 3rd Qu. Max. 1.000 1.300 1.600 1.672 2.000 2.500>> plot(x1,y1,xlim=c(1.5,4),ylim=c(.05,3),xlab='sw',ylab='pw',col='blue')> points(x2,y2,col='red')>> #画分类边界图,即画h(x)=0.5的图像> x3<-seq(1.5,4,length=100)> y3<-(3.796/14.735)*x3+13.415/14.735> lines(x3,y3) |
拟合概率曲线图:
(基本上符合logistic模型的S型曲线)

训练样本散点图及分类边界:
(画logistic回归的分类边界即画曲线h(x)=0.5)



 浙公网安备 33010602011771号
浙公网安备 33010602011771号