citup软件安装过程及克隆分析
citup软件 https://github.com/amcpherson/citup 安装需要conda环境
首先要将citup的安装包路径放到conda的默认channel里
conda config --add channels http://conda.anaconda.org/dranew
然后安装cplex依赖,直接安装的话也比较麻烦,因为cplex也有很多依赖,所以我们也把ceplx的channel加进去
conda config --add channels https://conda.anaconda.org/IBMDecisionOptimization/linux-64
我们看一下现在的默认channel
$ conda config --show-source ==> /home/.condarc <== channels: - http://conda.anaconda.org/dranew - defaults - https://conda.anaconda.org/IBMDecisionOptimization/linux-64
然后我们就可以安装citup了
conda install citup
如果我们没有默认安装路径权限,也可以指定目录安装
conda install citup --prefix .
一般会提示我们要安装很多依赖包,而且有些依赖特别容易断,像这样
$ conda install citup --prefix ./ Fetching package metadata ................. Solving package specifications: . Package plan for installation in environment /home/test: The following NEW packages will be INSTALLED: blas: 1.0-mkl blosc: 1.15.0-hd408876_0 boost_source: 1.60.0-0 dranew bzip2: 1.0.6-h14c3975_5 ca-certificates: 2019.1.23-0 certifi: 2019.3.9-py27_0 citup: 0.1.1-py27_1 dranew cplex: 12.8-py27_0 IBMDecisionOptimization decorator: 4.4.0-py27_1 hdf5: 1.10.4-hb1b8bf9_0 intel-openmp: 2019.3-199 libedit: 3.1.20181209-hc058e9b_0 libffi: 3.2.1-hd88cf55_4 libgcc-ng: 8.2.0-hdf63c60_1 libgfortran-ng: 7.3.0-hdf63c60_0 libstdcxx-ng: 8.2.0-hdf63c60_1 lzo: 2.10-h49e0be7_2 mkl: 2019.3-199 mkl_fft: 1.0.12-py27ha843d7b_0 mkl_random: 1.0.2-py27hd81dba3_0 ncurses: 6.1-he6710b0_1 networkx: 2.2-py27_1 numexpr: 2.6.9-py27h9e4a6bb_0 numpy: 1.16.3-py27h7e9f1db_0 numpy-base: 1.16.3-py27hde5b4d6_0 openssl: 1.1.1b-h7b6447c_1 pandas: 0.24.2-py27he6710b0_0 pip: 19.1.1-py27_0 pypeliner: 0.5.0-py27h1453be2_0 dranew pytables: 3.5.1-py27h71ec239_0 python: 2.7.16-h9bab390_0 python-dateutil: 2.8.0-py27_0 pytz: 2019.1-py_0 readline: 7.0-h7b6447c_5 scikit-learn: 0.20.3-py27hd81dba3_0 scipy: 1.2.1-py27h7c811a0_0 setuptools: 41.0.1-py27_0 six: 1.12.0-py27_0 snappy: 1.1.7-hbae5bb6_3 sqlite: 3.28.0-h7b6447c_0 tk: 8.6.8-hbc83047_0 wheel: 0.33.2-py27_0 zlib: 1.2.11-h7b6447c_3 Proceed ([y]/n)?
其实我们只需要几个依赖就够了,可以不安装全部依赖,网快的话还是尽量完整安装
conda install --no-deps cplex --prefix ./ conda install --no-deps pypeliner --prefix ./
conda install --no-deps hdf5 --prefix ./
然后将安装后的各个路径都加入到Python的环境变量里就可以安装citup
安装后就可以在lib/python2.7/site-packages路径下找到相应安装包了
这里citup-0.1.0-py2.7.egg需要解压一下
unzip citup-0.1.0-py2.7.eg
这样就基本完成了
这样的话就安装完成了
要注意的就是Python环境变量的问题了,确保Python可以找到这些安装包
如果-h有上面的结果而真正运行报错的话就是环境变量的问题
解决方案:
用citup安装的2.7版本的Python输出sys.path把这些路径加到环境变量里
export sys.path输出路径:$PATH
citup分析运行比较简单可以参考官方网页:https://shahlab.ca/projects/citup/
需要注意的是加这个参数:--submit local
输出结果是一个HDF格式的h5文件:results.h5
这是一个基于键引用的树型结构,可以用Python的h5py包读取处理数据还需要numpy和pandas
hf=h5py.File(results.h5,'r')
用hf.keys()命令我们可以看到有两个键results和trees,这两个键下面又包含很多键,具体结构:

我看从上述结果中取最佳拟合树:
opnum=hf["results/optimal/index"][0]
获取此树的节点克隆频率
cellfreq=hf["trees/" + str(opnum) + "/clone_freq/block0_values"][:]
获取此树的分支进化:
tree=hf["trees/" + str(opnum) + "/adjacency_list/block0_values"][:]
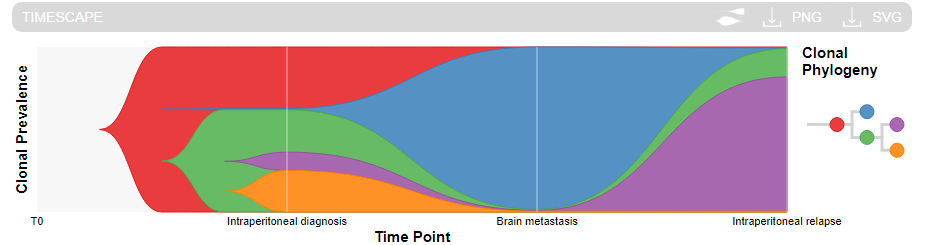
将这两个文件整理成TimeScape的输入文件:https://bioconductor.org/packages/release/bioc/vignettes/timescape/inst/doc/timescape_vignette.html
就可以得到这样的图了:
have fine!




