在本地成功部署 AlphaFold 3:完整指南
在本地成功部署 AlphaFold 3:完整指南
感谢AlphaFold 3开源,自己也尝试本地部署,中间经历了各自报错,最终完成了AlphaFold 3 本地部署,分享给大家,互相学习
AlphaFold 是由 DeepMind 开发的革命性工具,它通过深度学习为蛋白质折叠问题提供了前所未有的解决方案。自从 AlphaFold 发布以来,它已经改变了生命科学领域,尤其是蛋白质结构预测方面的研究。AlphaFold 3 是最新的版本,优化了预测精度,并且开源了,为广大科研人员提供了极大的便利。
这篇博客将分享本人如何在本地部署 AlphaFold 3,解决可能遇到的错误,最终成功运行 AlphaFold 3 的详细过程。
环境准备
在开始部署 AlphaFold 3 之前,确保已经准备好了以下环境和依赖:
- 操作系统 AlphaFold 3只支持Linux操作系统
- 硬件要求 有条件最好能按照官方的要求来
步骤 1:安装 Docker
AlphaFold 3 强烈推荐使用 Docker 部署。Docker 可以帮助避免环境配置上的问题,并确保系统兼容性。
在 Ubuntu 上,可以通过以下命令安装 Docker:
sudo apt-get update
sudo apt-get install -y docker.io
安装完成后,启动 Docker 服务并检查 Docker 是否成功安装:
sudo systemctl start docker
sudo systemctl enable docker
docker --version
sudo docker run hello-world
确保 Docker 安装无误,并且能够正常运行。
步骤 2:获取 AlphaFold 3 源代码
AlphaFold 3 的源代码已经开源,你可以通过 git 克隆代码库:
git clone https://github.com/google-deepmind/alphafold3.git
你也可以通过直接下载压缩包来获取代码,具体方法可以参考官方文档。
步骤 3:为A100安装NVIDIA驱动程序
docker run --rm --gpus all nvidia/cuda:12.6.0-base-ubuntu22.04 nvidia-smi
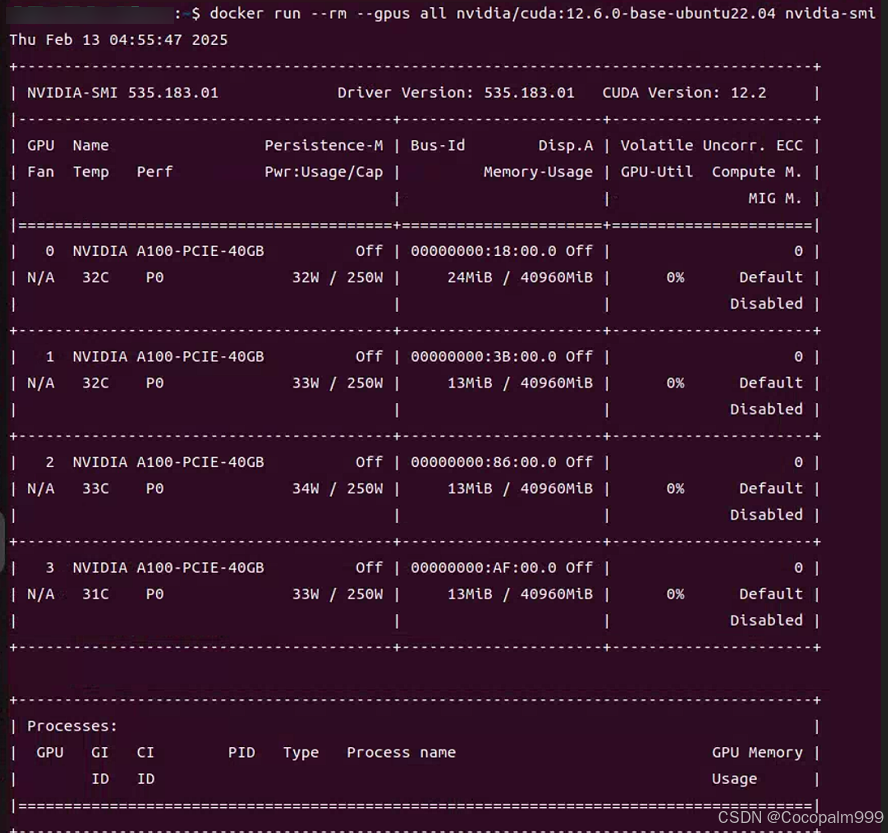
显示类似结果说明为A100安装NVIDIA驱动程序正确
步骤 4:下载依赖数据库
cd alphafold3 #alphafold3是上文步骤3的文件夹
python3 fetch_databases.py --download_destination=<DATABASES_DIR>
下载成功后,<DATABASES_DIR>文件夹下包含以下文件,
pdb_2022_09_28_mmcif_files.tar
bfd-first_non_consensus_sequences.fasta
mgy_clusters_2022_05.fa
nt_rna_2023_02_23_clust_seq_id_90_cov_80_rep_seq.fasta
pdb_seqres_2022_09_28.fasta
rfam_14_9_clust_seq_id_90_cov_80_rep_seq.fasta
rnacentral_active_seq_id_90_cov_80_linclust.fasta
uniprot_all_2021_04.fa
uniref90_2022_05.fa
步骤 5:获取模型参数
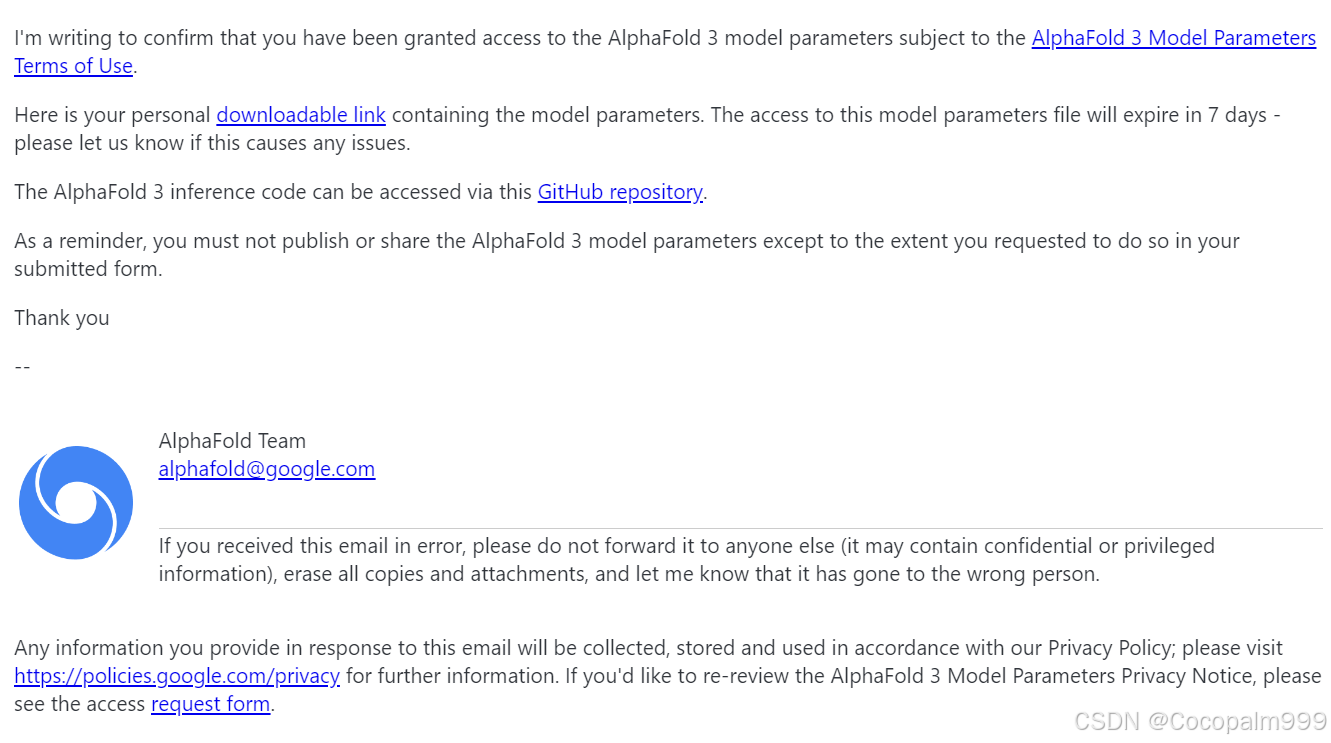
AlphaFold 3的模型参数需要向Google DeepMind申请,获得权限后不能转手、不能商用、输出结果只能自己用等等,
步骤 6:构建AlphaFold3 Docker容器
docker build -t alphafold3 -f docker/Dockerfile .
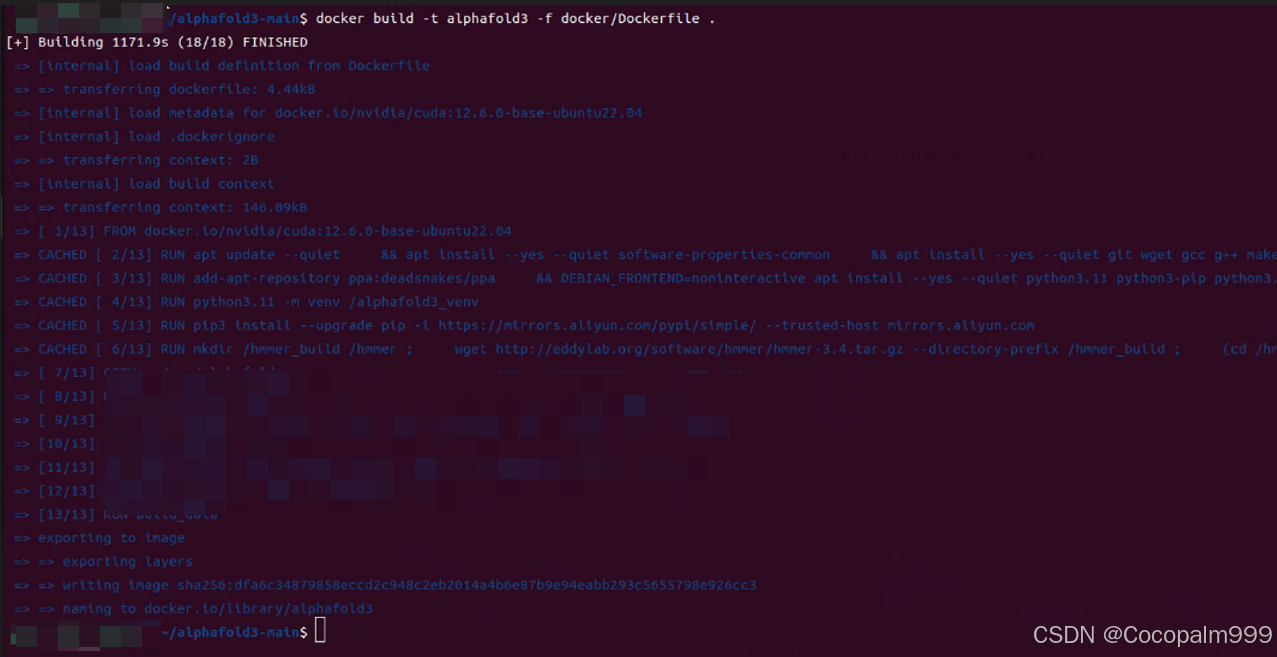
现在可以使用AlphaFold 3了,
docker run -it \
--volume $HOME/af_input:/root/af_input \
--volume $HOME/af_output:/root/af_output \
--volume <MODEL_PARAMETERS_DIR>:/root/models \
--volume <DATABASES_DIR>:/root/public_databases \
--gpus all \
alphafold3 \
python run_alphafold.py \
--json_path=/root/af_input/fold_input.json \
--model_dir=/root/models \
--output_dir=/root/af_output
步骤 7:处理错误和常见问题
在安装过程中,我遇到了一些常见的错误和解决方案,分享给大家:
错误 1:Docker 镜像构建失败
- 原因:可能是由于系统环境问题(如缺少依赖、网络问题等)。
- 解决方案:检查 Dockerfile 中的依赖,确保网络没有被防火墙或代理限制。可以尝试手动安装缺失的依赖。
错误 2:数据库下载失败
- 原因:下载过程中网络不稳定,或者下载链接不可用。
- 解决方案:手动下载数据库文件并将其放置在指定目录。可以从 AlphaFold 的 GitHub 页面或官方链接中获取备用下载地址。
错误 3:GPU 内存不足
- 原因:AlphaFold 需要大量的显存,特别是在处理较大的蛋白质时。
- 解决方案:减小输入数据的规模,或将 AlphaFold 配置为只使用部分 GPU(如果有多个 GPU)。
步骤 8:测试查看结果
AlphaFold 会将预测结果保存到指定的输出目录。可以通过以下命令查看预测结果:
ls /mnt/data/output
fold_input.json
{
"name": "4PV8",
"sequences": [
{
"protein": {
"id": ["A", "B"],
"sequence": "GMRESYANENQFGFKTINSDIHKIVIVGGYGKLGGLFARYLRASGYPISILDREDWAVAESILANADVVIVSVPINLTLETIERLKPYLTENMLLADLTSVKREPLAKMLEVHTGAVLGLHPMFGADIASMAKQVVVRCDGRFPERYEWLLEQIQIWGAKIYQTNATEHDHNMTYIQALRHFSTFANGLHLSKQPINLANLLALSSPIYRLELAMIGRLFAQDAELYADIIMDKSENLAVIETLKQTYDEALTFFENNDRQGFIDAFHKVRDWFGDYSEQFLKESRQLLQQANDLKQG"
}
}
],
"modelSeeds": [1],
"dialect": "alphafold3",
"version": 1
}
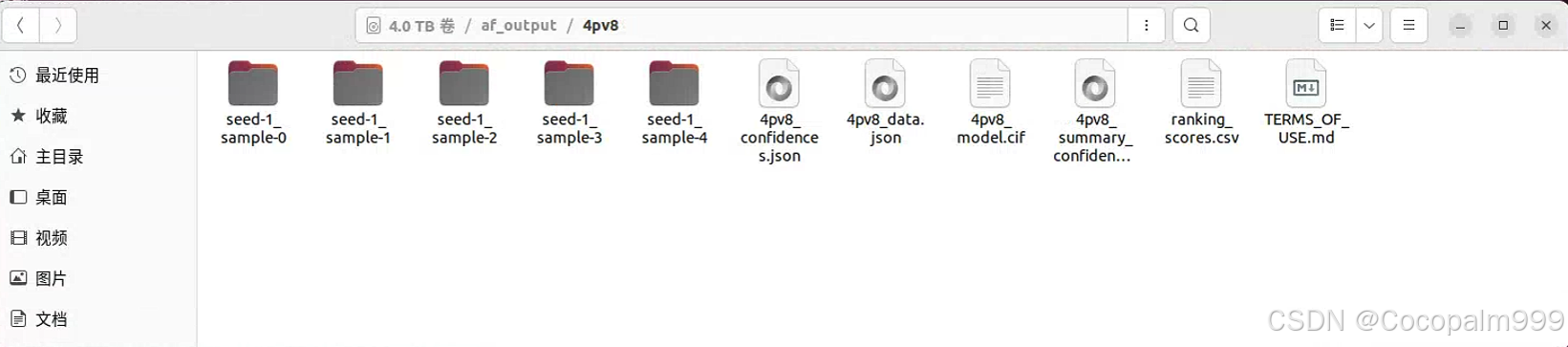
AlphaFold 会生成一个包含多个文件的输出文件夹,其中最重要的是 .pdb 文件,它包含了蛋白质的三维结构数据。
你可以使用 PyMOL、Chimera 等分子可视化工具打开这些 .pdb 文件,查看蛋白质的结构。
总结
成功部署 AlphaFold 3 是一个有挑战但值得的过程,本人一路试错,中途遇到很多BUG差点就放弃了,尤其是如果你第一次在本地进行生物学计算工具的部署。通过这篇博客,希望能够提供一些部署过程中可能遇到的常见问题的解决方法。
如果你在部署过程中遇到问题,欢迎在评论区留言,我们一起探讨学习!





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· 10年+ .NET Coder 心语 ── 封装的思维:从隐藏、稳定开始理解其本质意义
· 地球OL攻略 —— 某应届生求职总结
· 提示词工程——AI应用必不可少的技术
· 字符编码:从基础到乱码解决
· SpringCloud带你走进微服务的世界