LUNA16数据集(二)肺结节可视化
在检测到肺结节后,还需要可视化,这样才能为诊断服务。
我使用的项目地址为:https://github.com/wentaozhu/DeepLung
项目基于论文:DeepLung: Deep 3D Dual Path Nets for Automated Pulmonary Nodule Detection and Classification
该项目论文地址为:https://arxiv.org/abs/1801.09555
检测出肺结节可疑区域后,将其在原始CT图像上展示出来,原理比较简单,就是找出肺结节对应的切片,并圈出结节。贴下代码
首先是读取原始CT数据,需要sitk包,读取的filename是.mhd格式,里面包含CT图像的信息,不过数据其实在同名的.raw文件。
itkimage = sitk.ReadImage(filename)#读取.mhd文件
numpyImage = sitk.GetArrayFromImage(itkimage)#获取数据,自动从同名的.raw文件读取
之后,将其传入下面的函数,ct_scan就是上面的numpyImage。
def show_nodules(ct_scan,nodules,radius=20,pad=5,max_show_num=3):#radius是正方形边长一半,pad是边的宽度,max_show_num最大展示数
show_index = []
for idx in xrange(nodules.shape[0]):#lable是一个nx4维的数组,n是肺结节数目,4代表x,y,z,以及直径
if idx<max_show_num:
if abs(nodules[idx,0])+abs(nodules[idx,1])+abs(nodules[idx,2])+abs(nodules[idx,3])==0: continue
x, y, z = int(nodules[idx,0]), int(nodules[idx,1]), int(nodules[idx,2])
data = ct_scan[z]
#注意 y代表纵轴,x代表横轴
data[max(0,y-radius):min(data.shape[0],y+radius), max(0,x-radius-pad):max(0,x-radius)] = 3000#竖线
data[max(0,y-radius):min(data.shape[0],y+radius), min(data.shape[1],x+radius):min(data.shape[1],x+radius+pad)] = 3000#竖线
data[max(0,y-radius-pad):max(0,y-radius), max(0,x-radius):min(data.shape[1],x+radius)] = 3000#横线
data[min(data.shape[0],y+radius):min(data.shape[0],y+radius+pad), max(0,x-radius):min(data.shape[1],x+radius)] = 3000#横线
if z in show_index:#检查是否有结节在同一张切片,如果有,只显示一张
continue
show_index.append(z)
plt.figure
plt.imshow(data,cmap='gray')
plt.show()
需要强调的是,检测出的肺结节有不止一个,分布于不同切片,有些结节位于同一张切片,一般来讲,单个CT的肺结节数不会超过三个,所以只展示可能性最高的三个就应该够了。
此处可视化的办法是直接修改像素值,其它方法我还没有试过,相信只要能画出线条就可以。
结果展示

论文官方也给出了展示demo,不过略有不同,官方展示的是预处理后的切片,肺结节的位置自然也是与之对应,展示一下
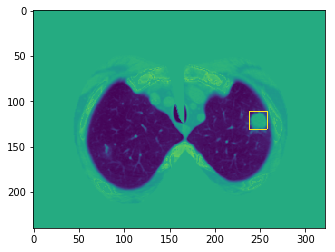
LUNA16切片的大小统一为512X512,预处理后的尺寸明显不同了。



