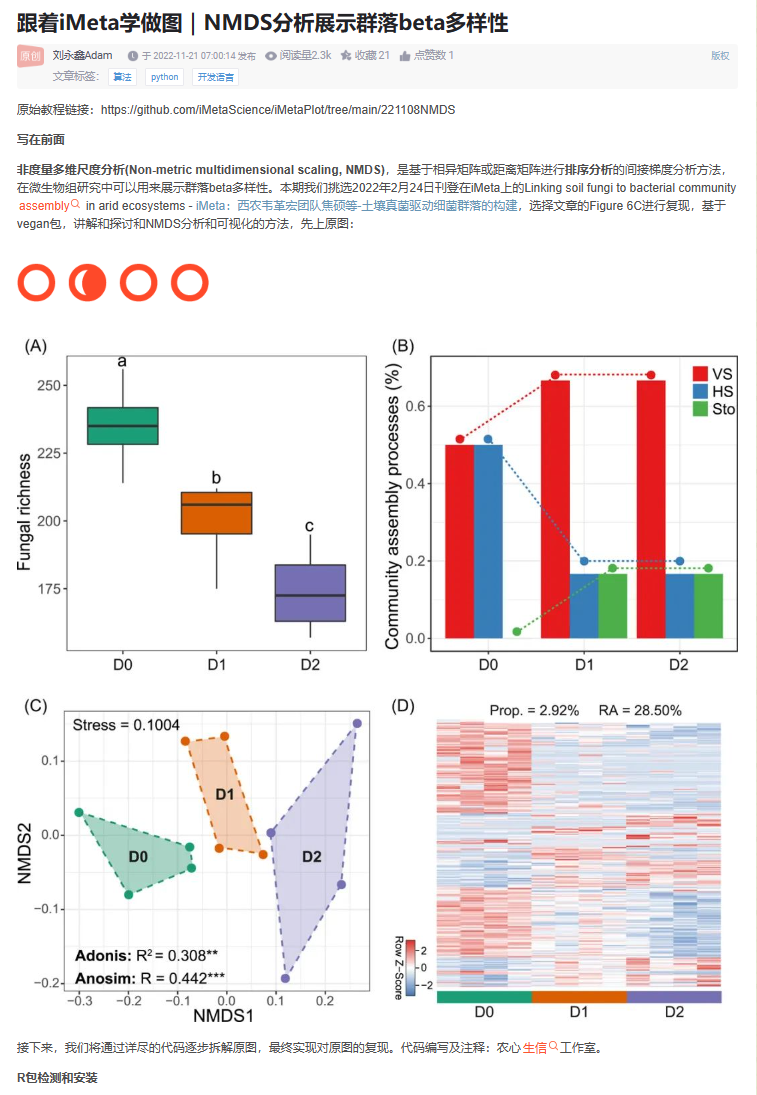
R : NMDS分析
以上事学习的源头,根据课题实际做了一点修改
# 加载必要的包
library(vegan)
library(ggplot2)
# 从txt文件载入数据
mydata <- read.delim("mydata.txt", header = TRUE, sep = "\t")
mygroups <- read.delim("mygroups.txt", header = TRUE, sep = "\t")
# 将数据中的NA值替换为0
mydata[is.na(mydata)] <- 0
# 计算bray_curtis距离
distance <- vegdist(mydata[, -1], method = 'bray') # 排除SampleID列
# NMDS排序分析
nmds <- metaMDS(distance, k = 2)
# 获得应力值(stress)
stress <- nmds$stress
# 将绘图数据转化为数据框
df <- as.data.frame(nmds$points)
# 与分组数据合并
df$SampleID <- rownames(df)
df <- merge(df, mygroups, by = "SampleID")
# 设置随机种子
set.seed(123)
# 基于bray-curtis距离进行PERMANOVA分析
adonis <- adonis2(as.dist(distance) ~ Group, data = df, permutations = 999)
# 基于bray-curtis距离进行ANOSIM分析
anosim <- anosim(distance, df$Group, permutations = 999)
# 应力值、Adonis R2与显著性、Anosim R与显著性
stress_text <- paste("Stress =", round(stress, 4))
adonis_text <- paste("Adonis R2 =", round(adonis$R2[1], 2), "p =", adonis$`Pr(>F)`[1])
anosim_text <- paste("Anosim R =", round(anosim$statistic, 2), "p =", anosim$signif)
my_colors <- c("#7FFFAA","#FFD700", "#FFA07A", "#DA70D6") # 你可以根据需要修改这些颜色
# 数据可视化
p <- ggplot(df, aes(x = MDS1, y = MDS2)) +
geom_point(aes(color = Group), size = 5) +
geom_polygon(aes(x = MDS1, y = MDS2, fill = Group, group = Group, color = Group),
alpha = 0.3, linetype = "longdash", linewidth = 1.5) +
scale_color_manual(values = my_colors) + # 添加这行代码来自定义点的颜色
scale_fill_manual(values = my_colors) + # 添加这行代码来自定义填充色
theme(plot.margin = unit(rep(1, 4), 'lines'),
panel.border = element_rect(fill = NA, color = "black", size = 0.5, linetype = "solid"),
panel.grid = element_blank(),
panel.background = element_rect(fill = 'white')) +
#guides(color = "none", fill = "none") +
ggtitle(paste(stress_text, adonis_text, anosim_text, sep = "\n"))
# 保存图形
ggsave("NMDS.pdf", p, height = 5.69, width = 7.42)这段代码主要针对生态学或生物信息学领域,涉及多样性分析,特别是计算和可视化基于bray-curtis距离的非度量多维尺度排序(NMDS)。以下是逐行的分析:
1-2: 加载必要的包
library(vegan)
library(ggplot2)加载vegan(用于生态多样性分析)和ggplot2(用于数据可视化)两个R包。
4-6: 从txt文件载入数据
mydata <- read.delim("mydata.txt", header = TRUE, sep = "\t")
mygroups <- read.delim("mygroups.txt", header = TRUE, sep = "\t")从txt文件中读取数据,并存储到mydata和mygroups两个数据框中。
8: 将数据中的NA值替换为0
mydata[is.na(mydata)] <- 0将mydata数据框中的所有NA值替换为0。
10-11: 计算bray_curtis距离
distance <- vegdist(mydata[, -1], method = 'bray')计算bray-curtis距离,排
set.seed(123)除第一列(假设为SampleID)。
13-14: NMDS排序分析
nmds <- metaMDS(distance, k = 2)使用非度量多维尺度排序(NMDS)进行排序分析。
16: 获得应力值(stress)
stress <- nmds$stress从nmds结果中提取应力值。
18-21: 将绘图数据转化为数据框并与分组数据合并
df <- as.data.frame(nmds$points)
df$SampleID <- rownames(df)
df <- merge(df, mygroups, by = "SampleID")将nmds排序后的点转化为数据框,并与分组数据mygroups合并。
23: 设置随机种子
set.seed(123)设置随机种子以确保结果可重复。
25-26: 基于bray-curtis距离进行PERMANOVA分析
adonis <- adonis2(as.dist(distance) ~ Group, data = df, permutations = 999)进行PERMANOVA分析以检验群组之间的差异是否显著。
28-29: 基于bray-curtis距离进行ANOSIM分析
anosim <- anosim(distance, df$Group, permutations = 999)进行ANOSIM分析以检验群组之间的差异。
31-34: 应力值、Adonis R2与显著性、Anosim R与显著性
这部分提取了PERMANOVA和ANOSIM的统计结果,并为后续的可视化准备文本信息。
36: 定义颜色
my_colors <- c("#7FFFAA","#FFD700", "#FFA07A", "#DA70D6")定义一个颜色向量以供后续使用。
38-54: 数据可视化
使用ggplot2进行数据可视化,展示NMDS结果并按组进行颜色区分。同时,使用geom_polygon绘制了群组的凸包。
56-57: 保存图形
ggsave("NMDS.pdf", p, height = 5.69, width = 7.42)将可视化的结果保存为PDF文件。
总结:这段代码主要实现了多样性数据的NMDS排序、群组间差异性统计分析,并将结果进行了可视化。


