enzyme design 整体流程及感想
2018-08-13 23:43 丨o聽乄雨o丨 阅读(1875) 评论(5) 编辑 收藏 举报想起什么来写什么吧。
整体流程(以Ceas2, TPP, G3P为例):
准备蛋白即配体参数文件(pdb文件需要有header,header的顺序符合cst block的顺序,且residue1和residue2的顺序也必须符合cst block);
设置CST文件;
准备protocol和flag文件;
运行enzyme_design;
结果处理。
CST文件准备:
要想准备好cst文件,需要的先决条件有两个,一个是对CST文件格式的了解,一个是对rosetta参数文件(.params)的了解。
首先,CST格式可见另外一篇博客https://www.cnblogs.com/wq242424/p/9463703.html;
其次,.params文件格式里面,主要注意我们感兴趣的原子,比如HIS的Nhis,确定A1,A2和A3,Nhis作为A1,A2和A3则由.params文件里对应ICOOR行的parent原子决定,A2是A1的parent,A3是A2的parent。如HIS里Nhis对应的原子及A1是ND1,A2为A1的parent为CG,A3为A2的parent为CB,.params文件格式参见.param文件格式,标准氨基酸的.params文件存放在$ROSETTAHOME/main/database/chemical/residue_type_sets/下。
最后,设置好CST文件后,一定要用 CstfileToTheozymePDB.linuxgccrelease 对CST进行验证,因为有时候随着标准氨基酸.params文件的更新,不同原子的定义可能会有改变,命令用法如下:
CstfileToTheozymePDB.linuxgccrelease -extra_res_fa G3P.params -match:geometric_constraint_file G3P.cst
命令会产生出 PDB_Model_G3P.cst_1.pdb 文件,删除pdb文件里的CONNECT行后,使用 pymol PDB_Model_G3P.cst_1.pdb 命令查看motif是否符合设定。
下面列举我遇到过的几种氨基酸的原子信息:
侧链信息:
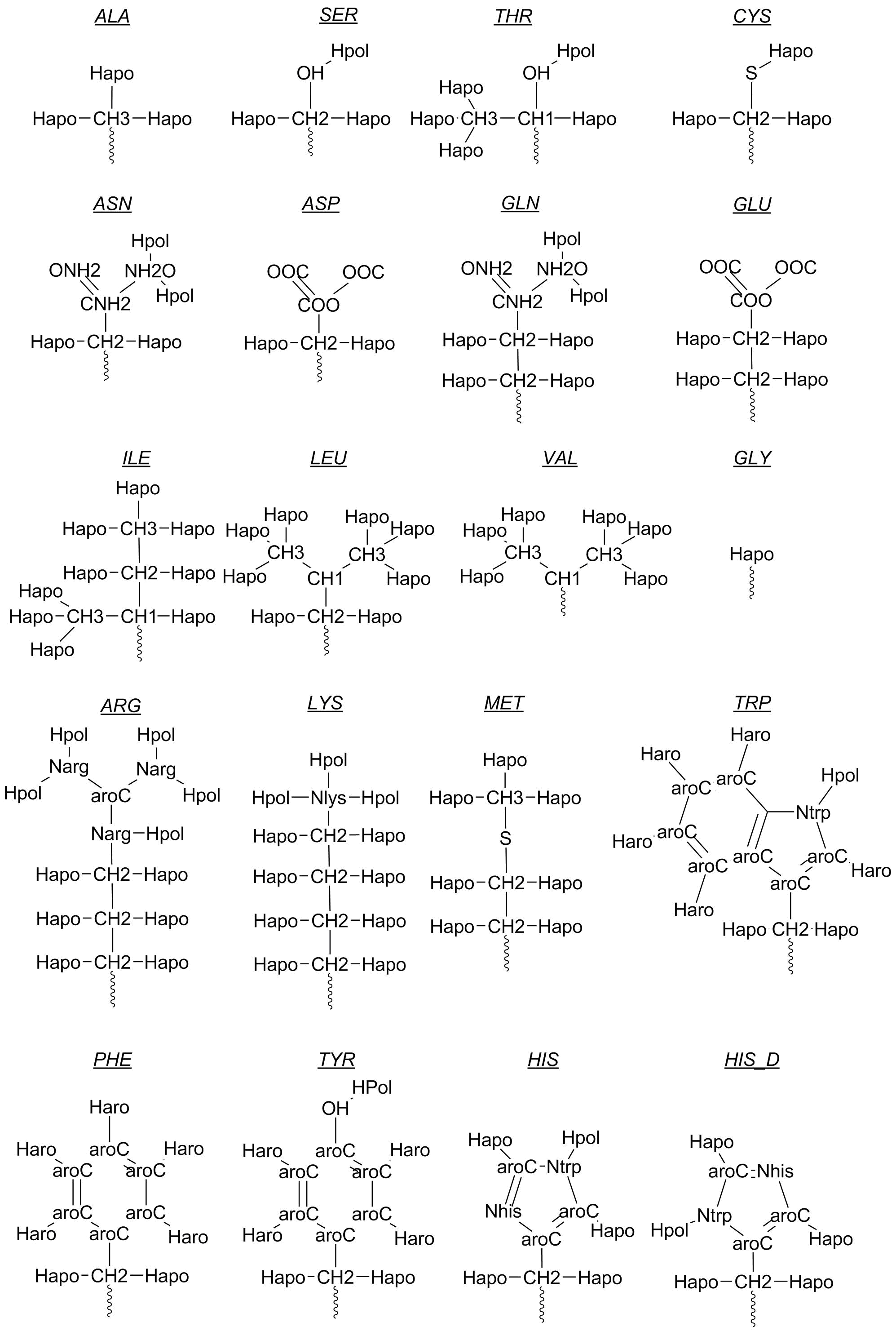
骨架信息:
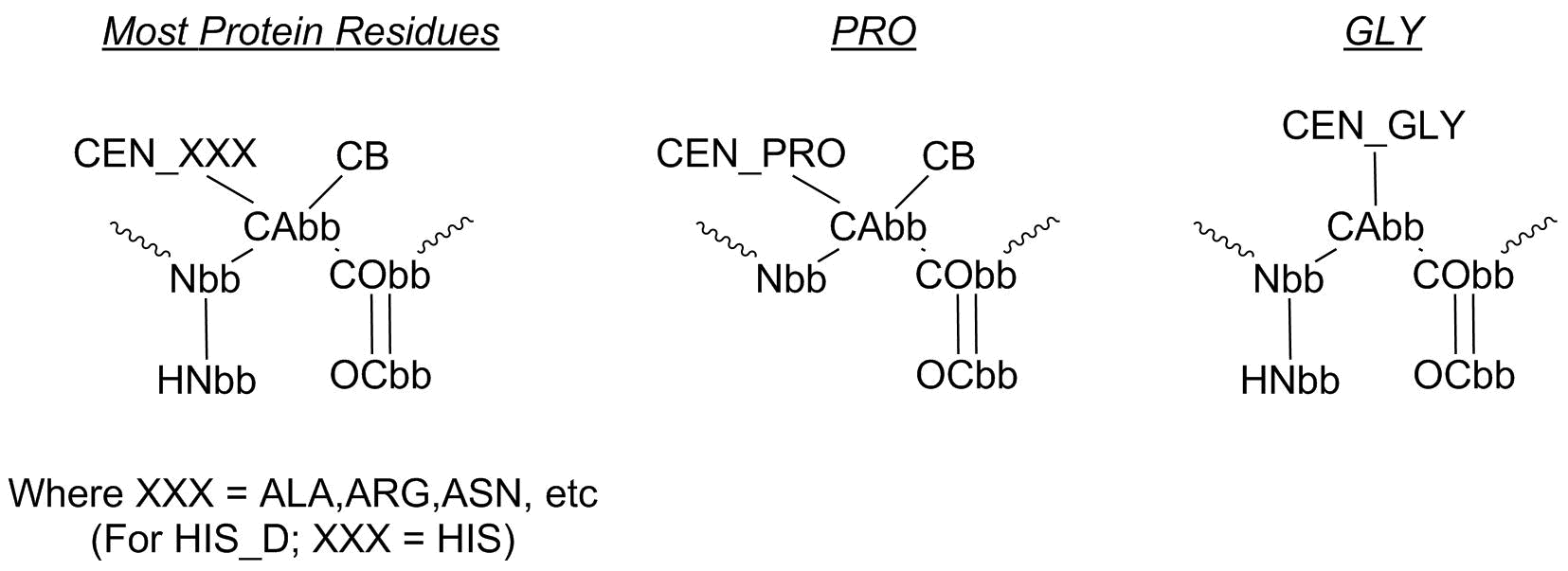
上述是列举的官方文档种的残基原子信息。
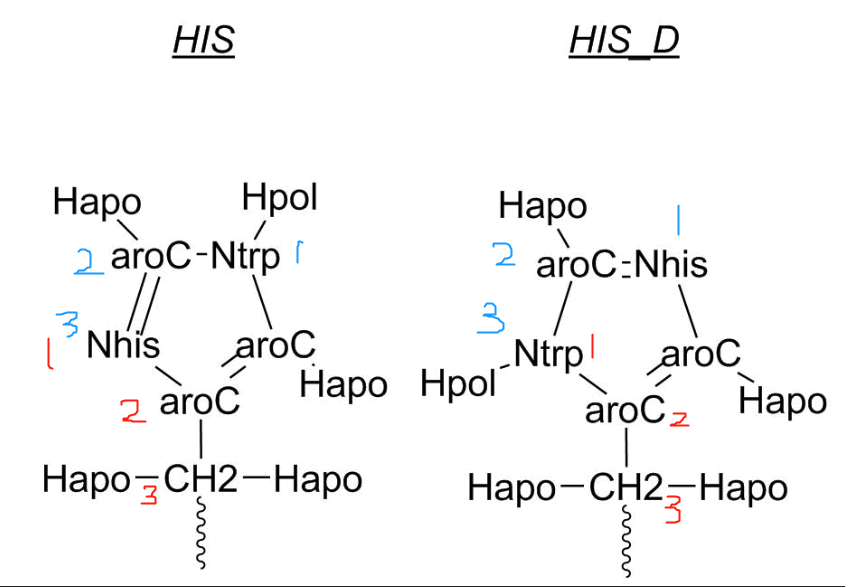
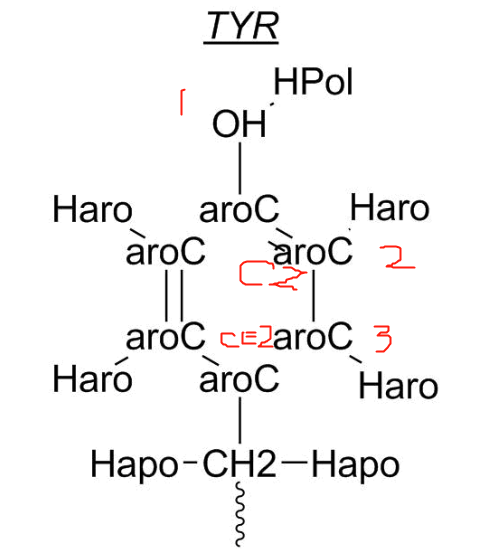
下面是自己应用过程种用到的:
Ntrp,和Nhis,
OH ,(注意逗号前有空格)
Narg,
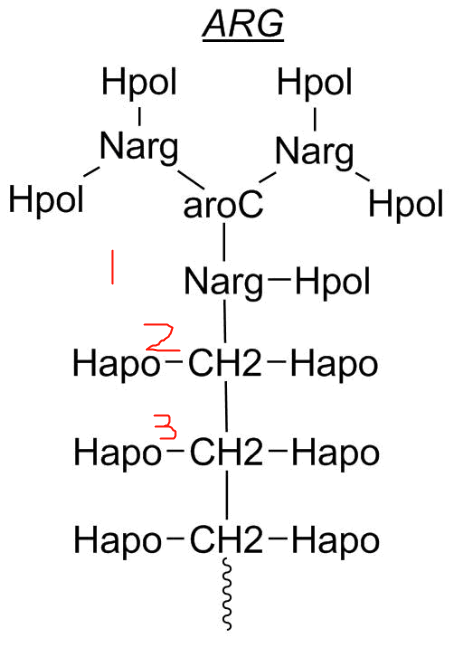
结果处理:
对设计出来的众多结果进行筛选,筛选规则的原文如下:
One approach currently used in the Baker group is the following: first, a subset of the 4-5 most important criteria is picked, i.e. total_score, ligand binding energy/SR_interface_E_1_2, total constraint score of the catalytic residues (all_cst), packstat, and buried unsatisfied polars of the ligand. Then, for each of these criteria, a minimum value is decided, which all designs considered for expression have to exceed ( i.e. total_score has to be lower than the corresponding Rosetta score of the undesigned scaffold, ligand_binding energy has to be < -10.0, and all_cst has to be < 1.0 ).
大体过程是:
首先,确定4-5个重要的对设计重要的特征,如total_score, ligand binding energy/SR_interface_E_1_2, total constraint score of the catalytic residues (all_cst), packstat, buried unsatisfied polars of the ligand等;
然后,确定每一个特征标准或者称为临界值,例如ligand_binding energy has to be < -10.0,又如 all_cst has to be < 1.0等;
最后,使用 DesignSelect.pl 脚本筛选出符合标准的设计。
DesignSelect.pl脚本的用法:
首先,需要准备一个标准或者临界值文件,如下:
req all_cst value < 1.0 req SR_4_interf_E_1_2 value < -10.0 output sortmin total_score
然后,使用命令:
DesignSelect.pl -d design.out -c <requirements file> -tag_column last > filtered_designs.out
对结果处理,得到的文件filtered_designs.out里包含符合我们条件的entry。
故障报错:
1. ERROR: unknown atom_name: TPP C
准备好flag文件,xml文件等后,运行 rosetta_scripts.linuxgccrelease @flags :
出现错误:ERROR: unknown atom_name: TPP C
没有排查出错误出处,由低版本(2016)换高版本rosetta_scripts(2018)运行后,问题解决。




