超参数调节
目录
1.前言
我刚开始搞这玩意的时候,我觉得这都有中文手册了,应该不难吧。结果我发现有的人不仅连中文都说不明白,而且不维护自己的网站,导致大部分外站链接都是404,真是气死我了。不仅如此,我发现调参数这个东西如果不配着具体的模型去讲的话跟没讲差不多。所以我爆肝了一下午代码,整了几个模型的样例,供各位参考。本文将先给出调参数的方法和函数并简单讲解让后通过几个具体的模型来进行具体验证。关于参数问题请前往网站https://scikit-learn.org.cn/view/704.html。这个网站相当于英文网站的中文机翻版本,比那什么中文手册好。
但是我还是建议直接去翻英文官方手册。把内容复制到deepl里,deepl的学术翻译效果很不错。
理论上的超参数调节
在sklearn中,为我们提供了四个用来调参的方法。这是前两个,共同点是要迭代每一种组合
给出指定的模型和参数列表后。这俩玩意在读入训练集训练时 fit(x_train,y_train) 会遍历所有的参数组合并计算训练的score,最终形成一个score最高的参数组合存在对象中等待取用。其中第二个(Halving啥啥啥的)采用锦标赛算法,比第一个快。
给出指定的模型和参数列表后。这俩玩意在读入训练集训练时 fit(x_train,y_train) 会随机遍历所有的参数>组合并计算训练的score,最终形成一个score最高的参数组合存在对象中等待取用。其中第二个(Halving啥啥啥的)采用锦标赛算法,比第一个快。
这两组比较来看,下面这种随机访问的方式比上面迭代的方式速度要快的多。
当然了,我们需要一套评价标准来评价参数的好坏,在这里我们把它称之为score。
sklearn的score大致如下:
显然,你要根据你的模型去选择合适的score。
一些模型的一些超参数可以通过自带交叉验证的模型来自动确定,有些模型甚至没有超参数。剩下的就需要自己写了。
好了讲完了,请问你学会了吗????????????????
3.根据模型讲解超参数调节
下面,我将会从几编译环境:vscode使用python 3.9.1(64bt)和vscode的jupyter插件。所有包都是最新版的。
3.1广义线性回归模型
广义线性回归模型是为了解决如下问题而准备的。说白了就是数理统计里的那个线性回归模型,只不过这里面写成矩阵了罢了。
现在我们一边看代码一边解释(以下所有讲解代码省略import)
data = datasets.fetch_california_housing() #加利福尼亚房价数据集
x_train,x_test,y_train,y_test = train_test_split(data.data,data.target,test_size = 0.2,random_state =20)
解释一下数据集划分函数train_test_split的参数(请注意返回值的顺序和参数顺序)
test_size:float or int, default=None
测试集的大小,如果是小数的话,值在(0,1)之间,表示测试集所占有的比例.如果是整数,表示的是测试集的具体样本数。如果train_size也是None的话,会有一个默认值0.25
train_size:float or int, default=None
和test_size一样,同上
random_state:int or RandomState instance, default=None
这个参数表示随机状态,因为每次分割都是随机的。为了保证分割时不改变分割的数据集,必须指定一个特定的参数。
shuffle: bool, default=True
是否重洗数据(洗牌),就是说在分割数据前,是否把数据打散重新排序这样子,看上面我们分割完的数据,都不是原始数据集的顺序,默认是要重洗的
讲完这个划分数据集,继续看
st = StandardScaler()
st.fit(x_train)
x_train = st.transform(x_train)
x_test = st.transform(x_test)
这是数据的标准化,方法是用(x-u)/σ,u是所有x的均值,σ是标准差。相当于把数据弄成标准正态分布
还有一个标准化是MinMaxScaler,就是用极大值和极小值缩放,MinMaxScaler有一个重要参数,feature_range,控制我们希望把数据压缩到的范围,默认是feature_range = [0,1]
regr = linear_model.LinearRegression()
regr.fit(x_train, y_train)
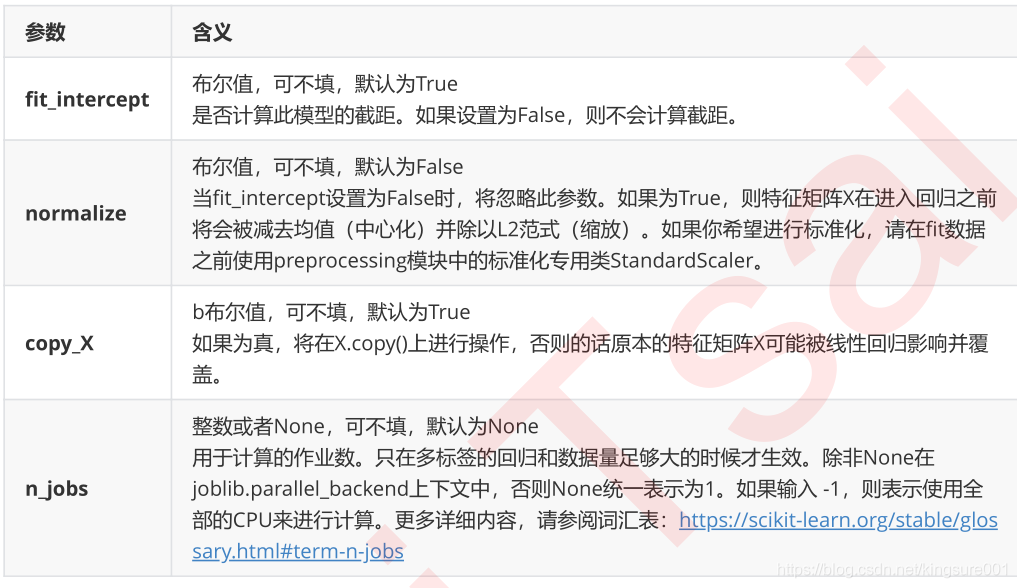
线性回归模型的参数如下图
可以看到没有超参数,所以这种线性模型对数据的要求很高很高。当然也不用调参数。
注意,这四个参数几乎每个模型都有,意思也基本差不多,之后这些参数会被省略不讲。这四个参数基本上不用调整。还有normalize参数在未来的更新中不是默认值为False就是被移除,目的是强制手写StandardScaler的使用。
y_pred = regr.predict(x_test)
print("系数:\n",regr.coef_)
print("截距:\n",regr.intercept_)
print("决定系数(越接近一意味着模型的线性程度越好,如果为负数,那就意味着跟瞎jb猜差距不大):\n",r2_score(y_test,y_pred))
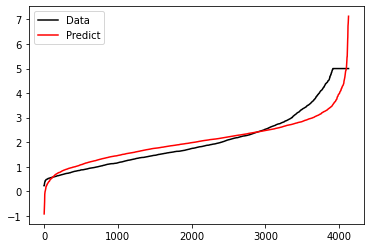
plt.plot(range(len(y_test)),sorted(y_test),c="black",label= "Data")
plt.plot(range(len(y_pred)),sorted(y_pred),c="red",label = "Predict")
plt.legend()
plt.show()
输出:
系数:
[ 0.83275185 0.1173856 -0.27597663 0.29900186 -0.00795271 -0.03963673
-0.88241635 -0.85338011]
截距:
2.0678235537788865
决定系数(越接近一意味着模型的线性程度越好,如果为负数,那就意味着跟瞎jb猜差距不大):
0.6121654293404898
这个R2_score的计算可以在网上搜一下,我感觉数理统计回归分析那里的那个r就是这个R2_score。
3.2岭回归模型
岭回归模型通过引入损失函数来防止广义线性模型带来的对测试集的过拟合以及当属性出现多重共线性时导致的结果不稳定。(我也不知道上面那句话在说啥,但是我知道当出现过拟合或者多重共线性的时候可以试试岭回归模型)
从此往下的数据集分割,数据标准化的代码也省略了。
#model = linear_model.Ridge(alpha=0.5)
model = linear_model.RidgeCV(alphas=np.arange(1,1001,100),store_cv_values=True)
#采用带交叉验证的岭回归,alphas就是测试的alpha值的元组,最后会得到最佳的alpha值。
model.fit(data.data,data.target)
print(model.score(data.data,data.target))
#这里是不进行交叉验证的R方系数
print(model.alpha_)
岭回归参数如下:
alpha:正则化项系数,较大的值指定更强的正则化
max_iter:共轭梯度求解器的最大迭代次数,需要与solver求解器配合使用。solver为sparse_cg和lsqr时,默认由scipy.sparse.linalg确定,solver为sag时,默认值为1000
tol:计算精度,默认=1e-3
solver:求解器{auto,svd,cholesky,lsqr,sparse_cg,sag,saga}
auto:根据数据类型自动选择求解器
svd:使用X的奇异值分解计算岭系数,奇异矩阵比cholesky更稳定
cholesky:使用标准的scipy.linalg.solve函数获得收敛的系数
sparse_cg:使用scipy.sparse.linalg.cg中的共轭梯度求解器。比cholesky更适合大规模数据(设置tol和max_iter的可能性)
lsqr:专用的正则化最小二乘方法scipy.sparse.linalg.lsqr
sag:随机平均梯度下降;仅在fit_intercept为True时支持密集数据
saga:sag改进,无偏版.采用SAGA梯度下降法可以使模型快速收敛
random_state:随机数生成器的种子,仅在solver="sag"时使用,默认None
这里面的超参数不少呀:alpha,精度,求解器,随机数种子。
1.如果需要随机数种子,必须显式指定一个数,不然的话种子也随机了。
2.alpha:正则化系数,是比较关键的超参数。
3.精度:这个只能看情况了,一般来说1e-6——1e-3就差不多了,大部分时候不用管。
4.solver:有个auto看到没?不进行严格的模型建立的话auto就完了。反之就要慢慢试了。
5.max_iter:这个超参数最好调整了,如果跑完fit跳出警告说数据未收敛,那就往大了写就行。
本演示只讲alpha的调参。
alpharange = np.arange(1,1001,100)
ridge, lr = [], []
for alpha in alpharange:
reg = linear_model.Ridge(alpha=alpha)
linear = linear_model.LinearRegression()
regs = cross_val_score(reg,data.data,data.target,cv=5,scoring = "r2").mean()
linears = cross_val_score(linear,data.data,data.target,cv=5,scoring = "r2").mean()
ridge.append(regs)
lr.append(linears)
这个相当于手动实现的上面那个RidgeCV的计算过程,当然这里加上了跟广义线性回归的比较。
param_grid = {'alpha':np.arange(1,1001,100)}
model = linear_model.Ridge()
gridsearch = GridSearchCV(model,param_grid,n_jobs = -1,scoring = "r2")
gridsearch.fit(data.data,data.target)
print(gridsearch.best_estimator_)
print(gridsearch.best_score_)
#在此处使用girdsearch结果基本一样。
这里是通过GridSearchCV实现。首先建造一个param_grid的字典。字典内部的结构是'参数':[值,值,值,……]其中参数必须与对应模型的参数名一致,参数值必须合法。当然你只需要写你要进行调整的参数就行了。然后用GridSearchCV建立model。再然后进行fit,fit完成后模型的最佳参数在best_estimator_中,其中这个值就是一个模型,可以直接用等号取出来直接用。
这里有一个规律,当模型本身有参数alpha时,对应的cv函数则是alphas。一般来说能用CV就不用自己写的gridsearch
GridSearchCV的参数介绍如下,其他三个的基本差不多。可以类推。
1.estimator
选择使用的模型,并且传入除需要确定最佳的参数之外的其他参数。(一般先建立模型再进行传参,不要直接在内部建立模型)
2.param_grid
需要最优化的参数的取值,值为字典或者列表。(列表是列表里面套字典,在讲到SVC和SVR,NCA的时候会见到)
1. scoring=None
模型评价标准,默认None。
根据所选模型不同,评价准则不同。自行学习评价标准的好坏。
4.n_jobs=1
n_jobs: 并行数,int:个数,-1:跟CPU核数一致, 1:默认值
5.cv=None
交叉验证参数,默认None,使用三折交叉验证。指定fold数量,默认为3,也可以是yield产生训练/测试数据的生成器。
进行预测的常用方法和属性
grid.fit():运行网格搜索
grid_scores_:给出不同参数情况下的评价结果
best_params_:描述了已取得最佳结果的参数的组合
best_score_:成员提供优化过程期间观察到的最好的评分
best_esi
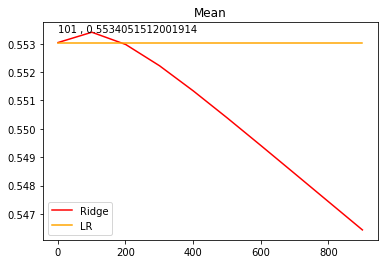
对上述的每个α计算它们的score,并且和广义线性模型的score进行比较。
可以看到其实吧,对于这个数据集来说提升不大,因为一般来说这些数据集都经过了去多重共线性处理。但是自己的数据集那就不一定了。
岭回归讲完了。
3.3lasso回归
lasso回归跟岭回归一样也是为了解决同样的问题,只不过换了一个损失函数。 但是,Lasso能有效拟合出稀疏系数的线性模型,因为它倾向于使用具有较少参数值的情况,有效地减少给定解决方案所依赖变量的数量。并且Lasso对alpha的值十分敏感,必须调好参数。关于基于最小角的lassolars自行学习。
lasso参数如下:(重复的想max_iter之类的省略了啊)
alpha : float, 可选,默认 1.0。
fit_intercept : boolean
是否进行拦截计算(intercept)。若 false,则不计算(比如数据已经经过集中了)。
precompute : True | False | array-like, 默认=False
是否使用预计算的 Gram 矩阵来加速计算。如果设置为 ‘auto’ 则机器决定。Gram 矩阵也可以 pass。对于 sparse input 这个选项永远为 True。
warm_start : bool, 可选
为 True 时, 重复使用上一次学习作为初始化,否则直接清除上次方案。
positive : bool, 可选
设为 True 时,强制使系数为正。
selection : str, 默认 ‘cyclic’
若设为 ‘random’, 每次循环会随机更新参数。
调alpha就行了啊。
alpha = np.logspace(-10,1,500,base = 10)(10^-10 到 10^1,等步长生成500个数)
model = linear_model.LassoCV(alphas = alpha,cv = 5)
model.fit(data.data,data.target)
print(model.alpha_)#0.0006222570836730231,这个值确实很小。
print(model.score(x_test,y_test))
LassoCV 类比上面的那个什么RidgeCV就行,但是注意,LassoCV的模型评估指标选用的是均方误差,而岭回归的模型评估指标是可以自己设定的,并且默认是R^2。
Lasoo讲完了,使用GridSearchCV的方法照着上面岭回归抄就行。
3.4弹性网络回归
弹性网络回归结合了岭回归和LASSO算法,通过两个超参数 λ(alpha) 和 ρ(l1_ratio) 来控制惩罚项的大小。对了,弹性网络回归还要设置一个random_state参数保证结果一致性。
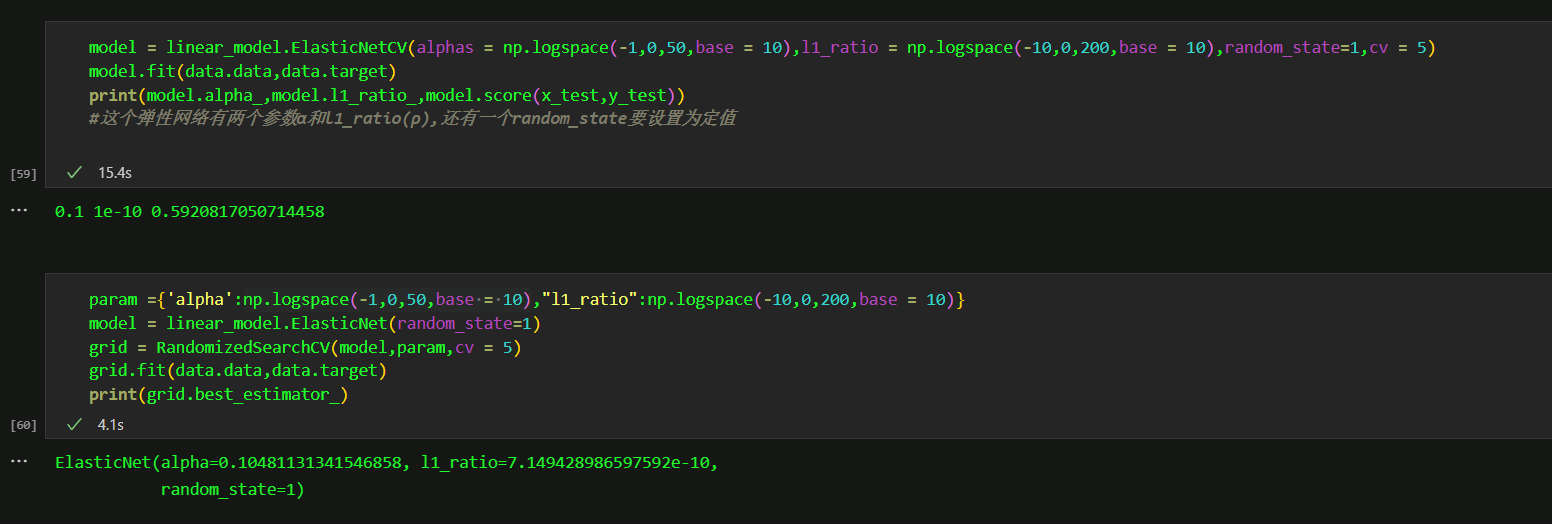
model = linear_model.ElasticNetCV(alphas = np.logspace(-1,0,50,base = 10),l1_ratio = np.logspace(-10,0,200,base = 10),random_state=1,cv = 5)
model.fit(data.data,data.target)
print(model.alpha_,model.l1_ratio_,model.score(x_test,y_test))
param ={'alpha':np.logspace(-1,0,50,base = 10),"l1_ratio":np.logspace(-10,0,200,base = 10)}
model = linear_model.ElasticNet(random_state=1)
grid = RandomizedSearchCV(model,param,cv = 5)
grid.fit(data.data,data.target)
print(grid.best_estimator_)
在这里我记得我用GirdSearchCV,结果半个小时了也没出个结果,气死偶类!
弹性网络回归请慎重调节参数,很容易使这个运行时间那叫一个长呀。注意这里的l1_ratio属于[0,1]闭区间
下面这张图展示了使用netcv,randomcv+net两种不同的调参方式的结果和时间消耗
3.45 Logit回归分类器
这玩意虽然叫回归模型,但是用起来更像一个分类器。重要参数如下图所示:这一堆参数写成grid的话太长太乱,不如自己手调。
还有一个正则化系数C,跟上面的alpha类似。其实吧有很多参数的名字(英文名)和意思在不同的模型里都基本差不多。
test = LogisticRegressionCV(penalty="l2",multi_class="auto",solver = "sag",max_iter=5050)
test.fit(X, y)
y_pred = test.predict(X_test)
print(accuracy_score(y_pred,y_test))
这里我出现了一个新的参数max_iter,就是迭代次数,有的时候数据会出现迭代次数不足导致数据未收敛(换句话说默认的次迭代有点少),这个参数弄大一点就行(照死里弄也行)。(这也算超参数吧)
3.5 knn分类器
KNN的全称是K Nearest Neighbors。KNN的原理就是当预测一个新的值x的时候,根据它距离最近的K个点是什么类别来判断x属于哪个类别。
knn分类器参数如下。
n_neighbors:这个值就是指 KNN 中的 “K”了。
weights(权重)
• 'uniform':不管远近权重都一样,就是最普通的 KNN 算法的形式。
• 'distance':权重和距离成反比,距离预测目标越近具有越高的权重。
• 自定义函数:自定义一个函数,根据输入的坐标值返回对应的权重,达到自定义权重的目的。
algorithm:
• 'brute' :蛮力实现
• 'kd_tree':KD 树实现 KNN
• 'ball_tree':球树实现 KNN
• 'auto': 默认参数,自动选择合适的方法构建模型
leaf_size:如果是选择蛮力实现,那么这个值是可以忽略的,当使用KD树或球树,它就是是停止建子树的叶子节点数量的阈值。默认30,但如果数据量增多这个参数需要增大,否则速度过慢不说,还容易过拟合。
p:和metric结合使用的,当metric参数是"minkowski"的时候,p=1为曼哈顿距离, p=2为欧式距离。默认为p=2。
metric:指定距离度量方法,一般都是使用欧式距离。
• 'euclidean' :欧式距离
• 'manhattan':曼哈顿距离
• 'chebyshev':切比雪夫距离
• 'minkowski': 闵可夫斯基距离(默认)
这里面的参数可不少,下面演示的是n_neighbors,weights,metric的超参数调节。我觉得其实想metric这种没必要交给系统呀。
param_grid = {"n_neighbors":range(1,25),"weights":["uniform","distance"],'metric': ['euclidean','manhattan','chebyshev','minkowski']}
kn = neighbors.KNeighborsClassifier(n_neighbors = 5)
grid = RandomizedSearchCV(kn,param_grid,cv = 10,scoring = "accuracy")
grid.fit(x_train,y_train)
print('随机搜索-最佳度量值:',grid.best_score_) # 获取最佳度量值
print('随机搜索-最佳参数:',grid.best_params_) # 获取最佳度量值时的代定参数的值。是一个字典
print('随机搜索-最佳模型:',grid.best_estimator_) # 获取最佳度量时的分类器模型
随机搜索-最佳度量值: 0.9666666666666666
随机搜索-最佳参数: {'weights': 'distance', 'n_neighbors': 17, 'metric': 'euclidean'}
随机搜索-最佳模型: KNeighborsClassifier(metric='euclidean', n_neighbors=17, weights='distance')
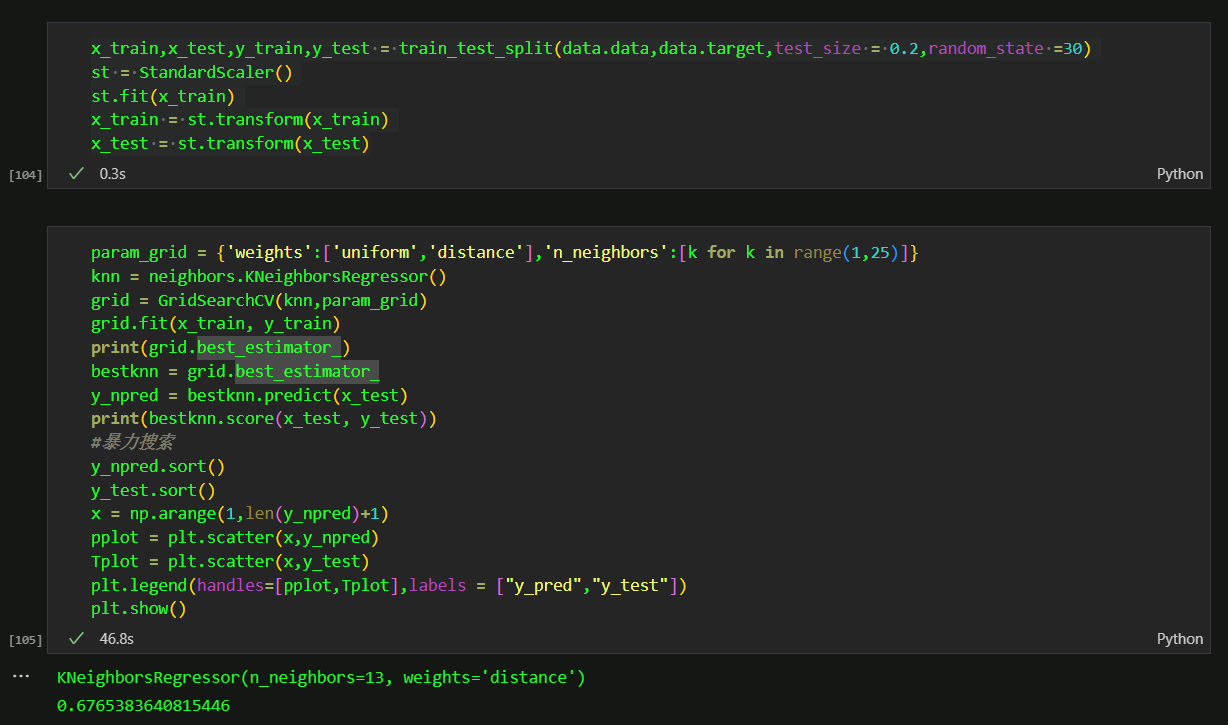
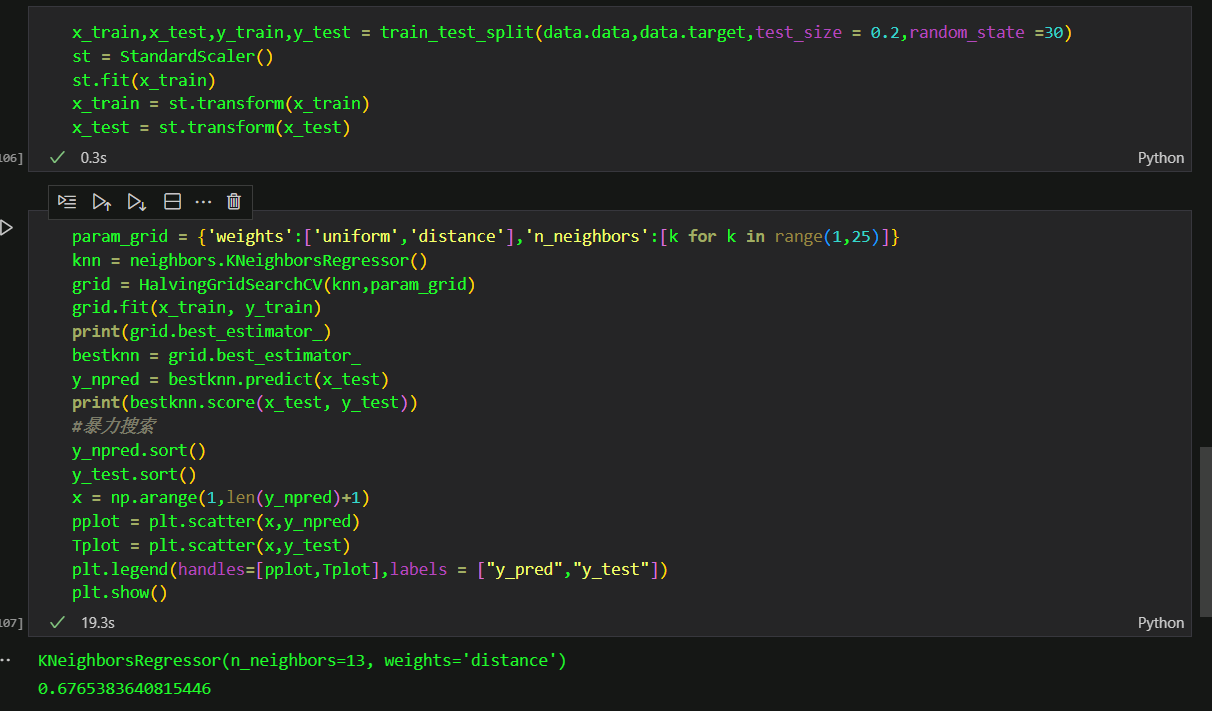
3.6knn回归
knn回归的参数和knn分类器差不多。要调整的也差不多。这里我使用GridSearchCV和HalvingGridSearchCV进行同样的测试,时间结果如下。对了对了,boston房价数据集现在官方说不推荐使用,请使用加利福尼亚房价数据集
data = datasets.fetch_california_housing()。
这是普通的网格搜索
这是锦标赛搜索
这才多少数据呀,就已经有一倍的差距了。
3.7Rnn分类器
3.8Rnn回归
Rnn分类器和knn分类器,Rnn回归和knn回归的原理都差不多,只不过一个是求K最近邻,一个是求半径罢了。参数也差不多,就是n_neighbors变成了radius。
param_grid = {"radius":range(1,25),"weights":["uniform","distance"]}
rn = neighbors.RadiusNeighborsClassifier(radius = 5)
grid = RandomizedSearchCV(rn,param_grid,cv = 10)
grid.fit(x_train,y_train)
print('随机搜索-最佳度量值:',grid.best_score_) # 获取最佳度量值
print('随机搜索-最佳参数:',grid.best_params_) # 获取最佳度量值时的代定参数的值。是一个字典
print('随机搜索-最佳模型:',grid.best_estimator_) # 获取最佳度量时的分类器模型
随机搜索-最佳度量值: 0.95
随机搜索-最佳参数: {'weights': 'distance', 'radius': 4}
随机搜索-最佳模型: RadiusNeighborsClassifier(radius=4, weights='distance')
3.8NCA
邻域成分分析(NCA)是一种用于度量学习的机器学习算法。 它以监督方式学习线性变换,以提高变换空间中随机最近邻规则的分类精度。
大白话就是:使用NCA可以提高knn/Rnn的分类精度。
nca = NeighborhoodComponentsAnalysis(random_state=25)
knn = KNeighborsClassifier(n_neighbors=3)
nca_pipe = Pipeline([("sc",StandardScaler()),('nca', nca), ('knn', knn)])
grid = {"nca":[nca],"nca__random_state":[25],'knn':[knn],'knn__n_neighbors':[i for i in range(1,30)],'knn__weights':['uniform']}
grid = GridSearchCV(nca_pipe,grid,cv = 3)
grid.fit(X_train, y_train)
print(grid.best_estimator_)
best_nca = grid.best_estimator_
best_nca.fit(X_train, y_train)
print(best_nca.score(X_test, y_test))
#Pipeline(steps=[('sc', StandardScaler()),
('nca', NeighborhoodComponentsAnalysis(random_state=25)),
('knn', KNeighborsClassifier(n_neighbors=18))])
这里遇到了一种新的方法Pipeline,我们很多时候对数据的处理是有顺序要求的,比如说先归一化,再用NCA分析,最后构建knn模型。Pipeline允许我们将所有的步骤整合为一个流水线管道。好处是既保证了取样的数据的统一处理,防止过程中出现的覆盖现象,又提供了一种联合调参是的机制。 此时grid的写法为 "管道变量":[管道变量],'管道变量__变量名',[参数列表]。那道杠是两个“_”。
3.9SVC
SVC, NuSVC 和 LinearSVC 能在数据集中实现多元分类。后两个我就不写了,懒。
SVC主要参数如下:
C: 浮点数。正则化参数。正则化的强度与C成反比。必须严格为正。默认1.0。
kernel:默认=’rbf’。指定算法中使用的内核类型。它必须是“linear”,“poly”,“rbf”,“sigmoid”,“precomputed”或者“callable”中的一个。如果没有给出,将默认使用“rbf”。如果给定了一个可调用函数,则用它来预先计算核矩阵。该矩阵应为形状数组(n_samples,n_samples)
degree:整数,默认=3。kernel为"poly"时有用。多项式核函数的次数(' poly ')。
gamma:浮点数。默认=’scale’。kernel为‘rbf’, ‘poly’ 和‘sigmoid’时有用。如果gamma='scale',则它使用1 / (n_features * X.var())作为gamma的值,如果是auto,则使用1 / n_features。gamma还可是一个浮点数。
coef0:浮点数,默认=0.0。核函数中的独立项。它只在' poly '和' sigmoid '中有意义。
class_weight:默认=None
在SVC中,将类i的参数C设置为class_weight [i] * C。如果没有给出值,则所有类都将设置为单位权重。“balanced”模式使用y的值自动将权重与类频率成反比地调整为n_samples / (n_classes * np.bincount(y))
decision_function_shape {‘ovo’, ‘ovr’}, 默认=’ovr’。二分类/多分类,默认多分类。且不建议调整此参数。
这个时候我们就会发现SVC的参数存在组合规则。这个时候显然GridSearchCV就很有必要了。
param_grid = [
{'C':[0.001,0.01,0.1,1,10,100],'kernel':['linear']},
{'C':[0.001,0.01,0.1,1,10,100],'kernel':['rbf'],'gamma':[1,0.1,0.01,0.001,0.0001]}
]
clr = svm.SVC(kernel = "linear",probability=True)
grid = RandomizedSearchCV(clr,param_grid,cv = 10)
grid.fit(x_train,y_train)
print(grid.best_estimator_)
SVC(C=10, kernel='linear', probability=True)
3.10SVR
SVR, NuSVR 和 LinearSVR 实现回归。后两个我就不写了,懒。
SVR参数如下:
kernel:默认=’rbf’指定算法中使用的内核类型。它必须是“linear”,“poly”,“rbf”,“sigmoid”,“precomputed”或者“callable”中的一个。如果没有给出,将默认使用“rbf”。如果给定了一个可调用函数,则用它来预先计算核矩阵。
degree:整数型,默认=3。多项式核函数的次数(' poly ')。将会被其他内核忽略。
gamma:浮点数或者{‘scale’, ‘auto’} , 默认=’scale’。核系数包含‘rbf’, ‘poly’ 和‘sigmoid’
coef0:浮点数,默认=0.0。核函数中的独立项。它只在' poly '和' sigmoid '中有意义。
C:浮点数,默认= 1.0
正则化参数。正则化的强度与C成反比。必须严格为正。此惩罚系数是l2惩罚系数的平方
(∪。∪)。。。zzz
param_grid = [
{'C':[0.001,0.01,0.1,1,10,100],'kernel':['linear']},
{'C':[0.001,0.01,0.1,1,10,100],'kernel':['rbf'],'gamma':[1,0.1,0.01,0.001,0.0001]}
]
clr = svm.SVR()
grid = RandomizedSearchCV(clr,param_grid,cv = 10)
grid.fit(x_train,y_train)
print(grid.best_estimator_)
SVR(C=100, gamma=0.1)
4.结语
写这么多乱七八糟的,主要是想留个档案,万一以后有用呢。