主成分分析PCA
主成分分析PCA
简介
降维就是一种对高维度特征数据预处理方法。降维是将高维度的数据保留下最重要的一些特征,去除噪声和不重要的特征,从而实现提升数据处理速度的目的。利用正交变换把由线性相关变量表示的数据转换为少数几个由线性无关变量表示的数据,线性无关变量称为主成分。主成分的数量通常小于原始变量数量,因此主成分分析常用于高维数据的降维,提取数据的主要特征分量。
主成分分析通常会得到协方差矩阵和相关矩阵。这些矩阵可以通过原始数据计算出来。协方差矩阵包含平方和与向量积的和。相关矩阵与协方差矩阵类似,但是第一个变量,也就是第一列,是标准化后的数据。如果变量之间的方差很大,或者变量的量纲不统一,我们必须先标准化再进行主成分分析。
PCA的具体实施的过程
将数据进行“去中心化”。
计算数据的协方差矩阵。
计算协方差矩阵的特征值和所对应的特征向量。
将特征值从大到小排序,保留前k kk个特征值,即前k kk个主成分。
将数据转换到上述的k kk个特征向量构建的新空间中。
Python代码实现
import numpy as np
import matplotlib.pyplot as plt
# 实验数据
Data = np.array([[10, 16], [3, 9], [1, 4], [7, 12], [2, 7]])
print(Data)
'''
[[10 16]
[ 3 9]
[ 1 4]
[ 7 12]
[ 2 7]]
'''
# 计算均值
meanVals = np.mean(Data, axis=0)
print(meanVals) # [4.6 9.6]
# 数据“去中心化”
DataMeanRemoved = Data - meanVals
print(DataMeanRemoved)
'''
[[ 5.4 6.4]
[-1.6 -0.6]
[-3.6 -5.6]
[ 2.4 2.4]
[-2.6 -2.6]]
'''
# 计算数据的协方差矩阵
CovData = np.cov(DataMeanRemoved, rowvar=False)
'''
rowvar:布尔值,可选
如果rowvar为True(默认值),
则每行代表一个变量X,另一个行为变量Y。
否则,转换关系:每列代表一个变量X,另一个列为变量Y。
'''
CovData = np.mat(CovData) # 必须转化为矩阵形式才能做后续的运算
print("协方差矩阵是:\n", CovData)
'''
[[14.3 17.05]
[17.05 21.3 ]]
'''
# 计算特征值和特征向量
eigVals, eigVects = np.linalg.eig(CovData) # 必须是矩阵形式
print("特征值是:\n", eigVals)
print("特征向量是:\n", eigVects)
'''
特征值是:
[ 0.39446927 35.20553073]
特征向量是:
[[-0.77494694 -0.6320263 ]
[ 0.6320263 -0.77494694]]
'''
maxEigVect = eigVects[:, 1] # 打印最大的特征值所对应的特征向量
print("第一主成分所对应的特征向量为:\n", maxEigVect)
'''
第一主成分所对应的特征向量为:
[[-0.6320263 ]
[-0.77494694]]
'''
print("去中心化后的数据是:\n", DataMeanRemoved)
'''
去中心化后的数据是:
[[ 5.4 6.4]
[-1.6 -0.6]
[-3.6 -5.6]
[ 2.4 2.4]
[-2.6 -2.6]]
'''
# 第一主成分向量与去中心化后的数据进行点积得到降维后的数据
LowDData = DataMeanRemoved * maxEigVect
print("降维后的数值为:\n", LowDData)
'''
降维后的数值为:
[[-8.37260242]
[ 1.47621024]
[ 6.61499753]
[-3.37673577]
[ 3.65813042]]
'''
# 原始数据
reconMat = (LowDData * maxEigVect.T) + meanVals
print("原始数据降维后的二维坐标表示为:\n", reconMat)
'''
原始数据降维后的二维坐标表示为:
[[ 9.89170494 16.0883226 ]
[ 3.6669963 8.45601539]
[ 0.41914758 4.47372793]
[ 6.73418582 12.21679104]
[ 2.28796536 6.76514304]]
'''
实现参考 https://blog.csdn.net/bwqiang/article/details/110407382
sklearn中PCA实现
from sklearn.decomposition import PCA
import numpy as np
X = np.array([[10, 16], [3, 9], [1, 4], [7, 12], [2, 7]])
pca = PCA(n_components = 1)
pca.fit(X)
print(pca.transform(X))
'''
[[ 8.37260242]
[-1.47621024]
[-6.61499753]
[ 3.37673577]
[-3.65813042]]
'''
数据降维并可视化
from sklearn import datasets
import matplotlib.pyplot as plt
# 加载数据
plt.switch_backend('TkAgg')
data = datasets.load_digits()
# 数据划分x/y
x = data.data
y = data.target
# 直接调用sklearn的主成分分析函数
from sklearn.decomposition import PCA
pca = PCA(n_components=2)
newdata = pca.fit_transform(x, y)
# 利用主成分分析步骤计算,或者使用numpy
# import numpy as np
# n = 2 # 确定降维维数
# mean = np.mean(x, axis=0) # 按列取平均
# norm_data = x - mean # 均值归一化
# cov = np.cov(norm_data, rowvar=False) # 计算协方差矩阵,每一列代表一个特征
# evalue, evector = np.linalg.eig(cov) # 计算特征值和特征向量
# index = np.argsort(evalue) # 特征值从大到小排序,index为对应下标
# n_index = index[-n:]
# n_vec = evector[:, n_index] # 取最大的n维特征值对应的特征向量构成映射P矩阵
# newdata = np.dot(norm_data, n_vec)
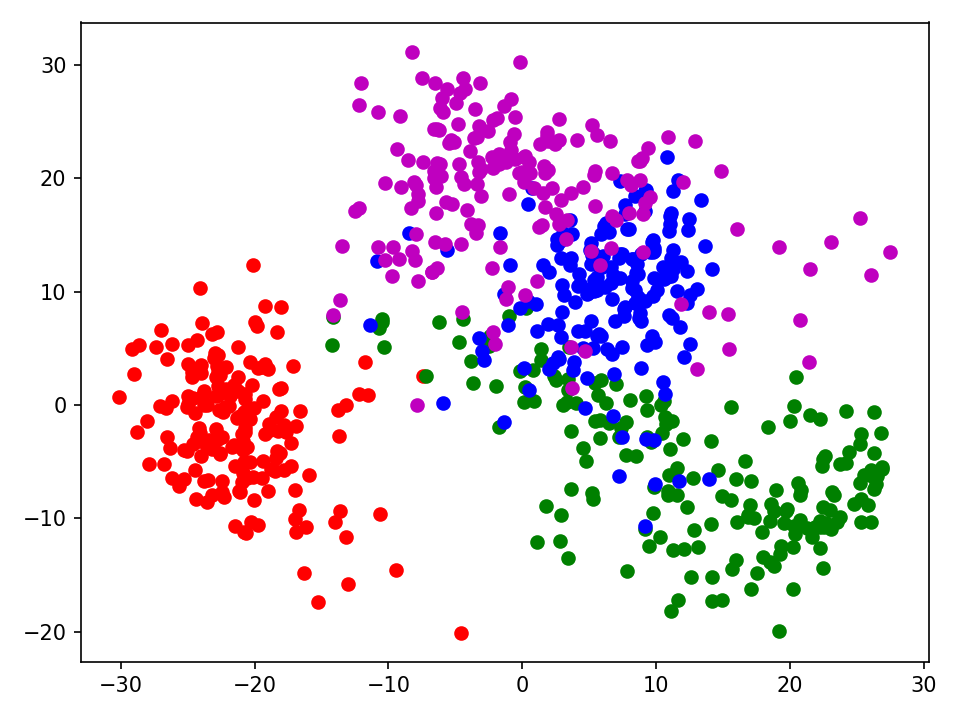
x0 = newdata[y == 0]
x1 = newdata[y == 1]
x2 = newdata[y == 2]
x3 = newdata[y == 3]
# x4 = newdata[y == 4]
# x5 = newdata[y == 5]
# x6 = newdata[y == 6]
# x7 = newdata[y == 7]
# x8 = newdata[y == 8]
# x9 = newdata[y == 9]
plt.scatter(x0[:, 0], x0[:, 1], c="red", marker='o')
plt.scatter(x1[:, 0], x1[:, 1], c="green", marker='o')
plt.scatter(x2[:, 0], x2[:, 1], c="blue", marker='o')
plt.scatter(x3[:, 0], x3[:, 1], c="m", marker='o')
# plt.scatter(x4[:, 0], x4[:, 1], c="yellow", marker='o')
# plt.scatter(x5[:, 0], x5[:, 1], c="cyan", marker='o')
# plt.scatter(x6[:, 0], x6[:, 1], c="black", marker='o')
# plt.scatter(x7[:, 0], x7[:, 1], c="white", marker='o')
# plt.scatter(x8[:, 0], x8[:, 1], c="purple", marker='o')
# plt.scatter(x9[:, 0], x9[:, 1], c="brown", marker='o')
plt.tight_layout()
plt.show()
PCA进行图像压缩
import numpy as np
import cv2
# 数据中心化
def centere_data(dataMat):
rows, cols = dataMat.shape
meanVal = np.mean(dataMat, axis=0) # 按列求均值,即求各个特征的均值
meanVal = np.tile(meanVal, (rows, 1))
newdata = dataMat - meanVal
return newdata, meanVal
# 最小化降维造成的损失,确定k
def Percentage2n(eigVals, percentage):
sortArray = np.sort(eigVals) # 升序
sortArray = sortArray[-1::-1] # 逆转,即降序
arraySum = sum(sortArray)
temp_Sum = 0
num = 0
for i in sortArray:
temp_Sum += i
num += 1
if temp_Sum >= arraySum * percentage:
return num
# 得到最大的k个特征值和特征向量
def EigDV(covMat, p):
D, V = np.linalg.eig(covMat) # 得到特征值和特征向量
k = Percentage2n(D, p) # 确定k值
print("保留99%信息,降维后的特征个数:" + str(k) + "\n")
eigenvalue = np.argsort(D)
K_eigenValue = eigenvalue[-1:-(k + 1):-1]
K_eigenVector = V[:, K_eigenValue]
return K_eigenValue, K_eigenVector
# PCA算法
def PCA(data, p):
dataMat = np.float32(np.mat(data))
# 数据中心化
dataMat, meanVal = centere_data(dataMat)
# 计算协方差矩阵
covMat = np.cov(dataMat, rowvar=False)
# 选取最大的k个特征值和特征向量
D, V = EigDV(covMat, p)
# 得到降维后的数据
lowDataMat = dataMat * V
# 重构数据
reconDataMat = lowDataMat * V.T + meanVal
return reconDataMat
if __name__ == '__main__':
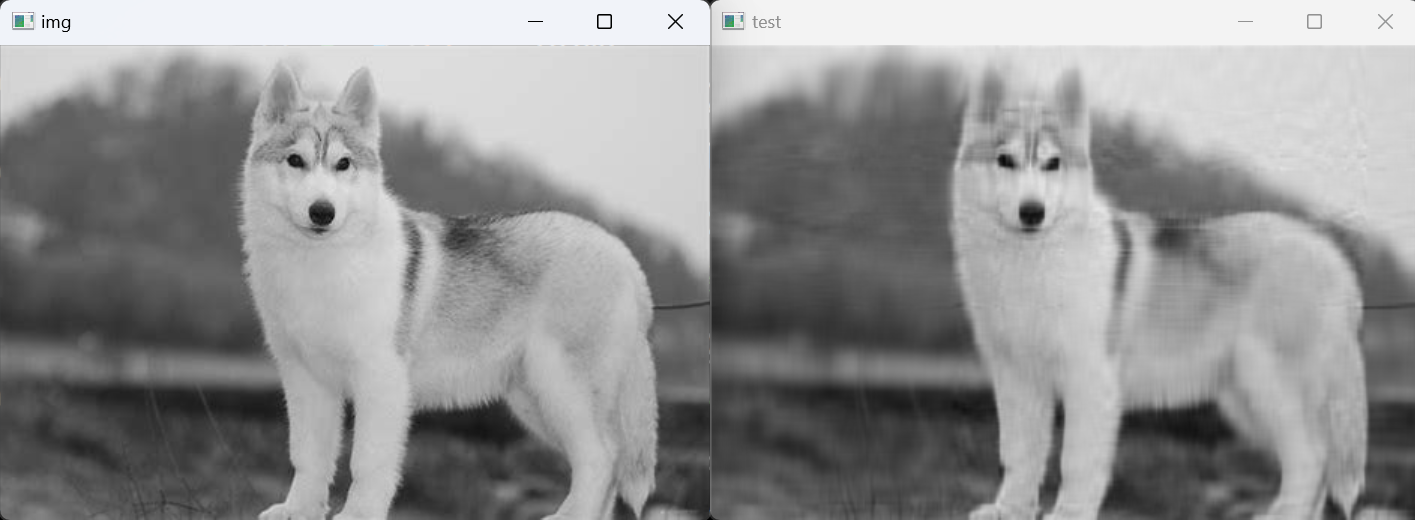
img = cv2.imread('../../assets/dog.jpg',0)
rows, cols, = img.shape
# pca = decomposition.PCA()
print("降维前的特征个数:" + str(cols) + "\n")
print(img.shape)
print('----------------------------------------')
PCA_img = PCA(img, 0.99)
print(PCA_img.shape)
print(PCA_img)
PCA_img = PCA_img.astype(np.uint8)
# print(PCA_img)
cv2.imshow('img', img) # 原图像
cv2.imshow('test', PCA_img) # 降维后的图像
cv2.waitKey(0)
cv2.destroyAllWindows()
参考资料
https://www.cnblogs.com/charlotte77/p/5625984.html
https://www.cnblogs.com/codewitness/p/15794652.html (图像降维重构)





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· winform 绘制太阳,地球,月球 运作规律
· 超详细:普通电脑也行Windows部署deepseek R1训练数据并当服务器共享给他人
· 上周热点回顾(3.3-3.9)
· AI 智能体引爆开源社区「GitHub 热点速览」
· 写一个简单的SQL生成工具