【机器学习】确定最佳聚类数目的10种方法
【机器学习】确定最佳聚类数目的10种方法
在聚类分析的时候确定最佳聚类数目是一个很重要的问题,比如kmeans函数就要你提供聚类数目这个参数,总不能两眼一抹黑乱填一个吧。之前也被这个问题困扰过,看了很多博客,大多泛泛带过。今天把看到的这么多方法进行汇总以及代码实现并尽量弄清每个方法的原理。
数据集选用比较出名的wine数据集进行分析
library(gclus)
data(wine)
head(wine)
Loading required package: cluster
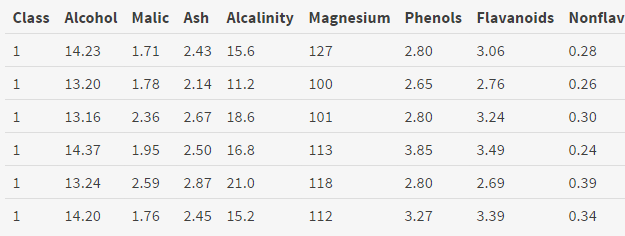 因为我们要找一个数据集进行聚类分析,所以不需要第一列的种类标签信息,因此去掉第一列。
因为我们要找一个数据集进行聚类分析,所以不需要第一列的种类标签信息,因此去掉第一列。
同时注意到每一列的值差别很大,从1到100多都有,这样会造成误差,所以需要归一化,用scale函数
dataset <- wine[,-1] #去除分类标签
dataset <- scale(dataset)
去掉标签之后就可以开始对数据集进行聚类分析了,下面就一一介绍各种确定最佳聚类数目的方法
判定方法
1.mclust包
mclust包是聚类分析非常强大的一个包,也是上课时老师给我们介绍的一个包,每次导入时有一种科技感 :) 帮助文档非常详尽,可以进行聚类、分类、密度分析
Mclust包方法有点“暴力”,聚类数目自定义,比如我选取的从1到20,然后一共14种模型,每一种模型都计算聚类数目从1到20的BIC值,最终确定最佳聚类数目,这种方法的思想很直接了当,但是弊端也就显然易见了——时间复杂度太高,效率低。
library(mclust)
m_clust <- Mclust(as.matrix(dataset), G=1:20) #聚类数目从1一直试到20
summary(m_clust)
Gaussian finite mixture model fitted by EM algorithm
Mclust EVE (ellipsoidal, equal volume and orientation) model with 3 components:
log.likelihood n df BIC ICL
-3032.45 178 156 -6873.257 -6873.549
Clustering table:
1 2 3
63 51 64
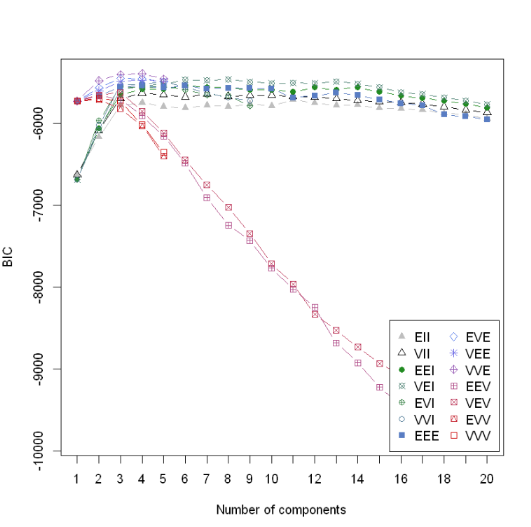
可见该函数已经把数据集聚类为3种类型了。数目分别为63、51、64。再画出14个指标随着聚类数目变化的走势图
plot(m_clust, "BIC")
下表是这些模型的意义
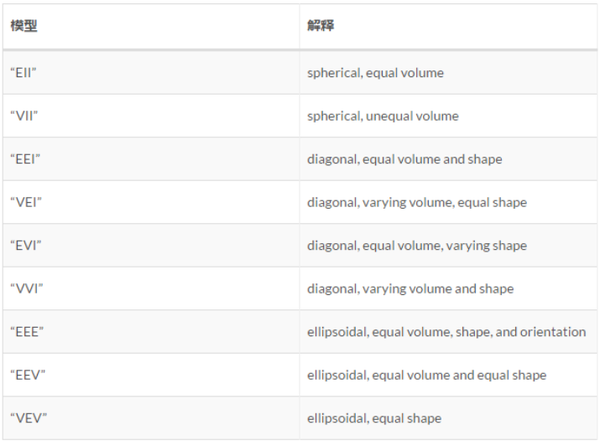
它们应该分别代表着相关性(完全正负相关——对角线、稍强正负相关——椭圆、无关——圆)等参数的改变对应的模型,研究清楚这些又是非常复杂的问题了,先按下不表,知道BIC值越大则说明所选取的变量集合拟合效果越好 上图中除了两个模型一直递增,其他的12模型数基本上都是在聚类数目为3的时候达到峰值,所以该算法由此得出最佳聚类数目为3的结论。
mclust包还可以用于分类、密度估计等,这个包值得好好把玩。
注意:此BIC并不是贝叶斯信息准则!!!
最近上课老师讲金融模型时提到了BIC值,说BIC值越小模型效果越好,顿时想起这里是在图中BIC极大值为最佳聚类数目,然后和老师探讨了这个问题,之前这里误导大家了,Mclust包里面的BIC并不是贝叶斯信息准则。
1.维基上的贝叶斯信息准则定义
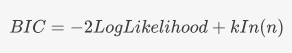
与log(likelihood)成反比,极大似然估计是值越大越好,那么BIC值确实是越小模型效果越好
2.Mclust包中的BIC定义[3]
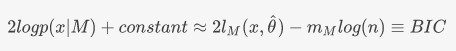 这是Mclust包里面作者定义的“BIC值”,此BIC非彼BIC,这里是作者自己定义的BIC,可以看到,这里的BIC与极大似然估计是成正比的,所以这里是BIC值越大越好,与贝叶斯信息准则值越小模型越好的结论并不冲突
这是Mclust包里面作者定义的“BIC值”,此BIC非彼BIC,这里是作者自己定义的BIC,可以看到,这里的BIC与极大似然估计是成正比的,所以这里是BIC值越大越好,与贝叶斯信息准则值越小模型越好的结论并不冲突
2.Nbclust包
Nbclust包是我在《R语言实战》上看到的一个包,思想和mclust包比较相近,也是定义了几十个评估指标,然后聚类数目从2遍历到15(自己设定),然后通过这些指标看分别在聚类数为多少时达到最优,最后选择指标支持数最多的聚类数目就是最佳聚类数目。
library(NbClust)
set.seed(1234) #因为method选择的是kmeans,所以如果不设定种子,每次跑得结果可能不同
nb_clust <- NbClust(dataset, distance = "euclidean",
min.nc=2, max.nc=15, method = "kmeans",
index = "alllong", alphaBeale = 0.1)
*** : The Hubert index is a graphical method of determining the number of clusters.
In the plot of Hubert index, we seek a significant knee that corresponds to a
significant increase of the value of the measure i.e the significant peak in Hubert
index second differences plot.
*** : The D index is a graphical method of determining the number of clusters.
In the plot of D index, we seek a significant knee (the significant peak in Dindex
second differences plot) that corresponds to a significant increase of the value of
the measure.
*******************************************************************
* Among all indices:
* 5 proposed 2 as the best number of clusters
* 16 proposed 3 as the best number of clusters
* 1 proposed 10 as the best number of clusters
* 1 proposed 12 as the best number of clusters
* 1 proposed 14 as the best number of clusters
* 3 proposed 15 as the best number of clusters
***** Conclusion *****
* According to the majority rule, the best number of clusters is 3
*******************************************************************
barplot(table(nb_clust$Best.nc[1,]),xlab = "聚类数",ylab = "支持指标数")
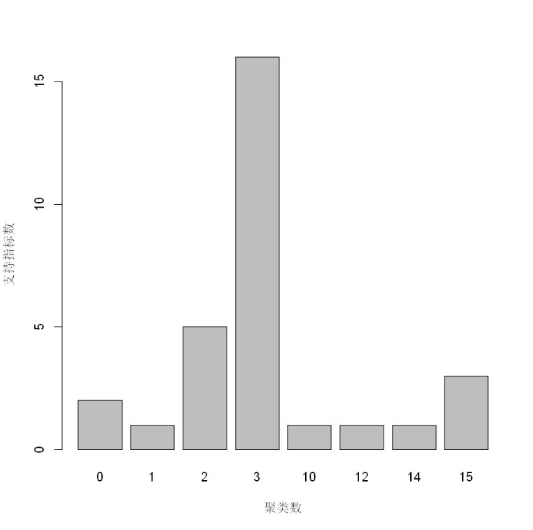
可以看到有16个指标支持最佳聚类数目为3,5个指标支持聚类数为2,所以该方法推荐的最佳聚类数目为3.
3. 组内平方误差和——拐点图
想必之前动辄几十个指标,这里就用一个最简单的指标——sum of squared error (SSE)组内平方误差和来确定最佳聚类数目。这个方法也是出于《R语言实战》,自定义的一个求组内误差平方和的函数。
wssplot <- function(data, nc=15, seed=1234){
wss <- (nrow(data)-1)*sum(apply(data,2,var))
for (i in 2:nc){
set.seed(seed)
wss[i] <- sum(kmeans(data, centers=i)$withinss)
}
plot(1:nc, wss, type="b", xlab="Number of Clusters",
ylab="Within groups sum of squares")}
wssplot(dataset)
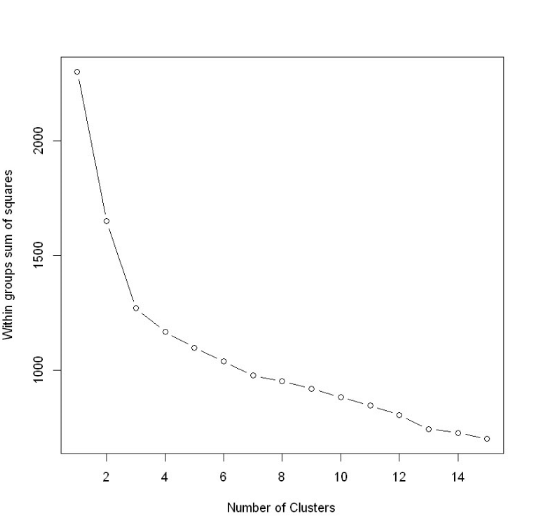
随着聚类数目增多,每一个类别中数量越来越少,距离越来越近,因此WSS值肯定是随着聚类数目增多而减少的,所以关注的是斜率的变化,但WWS减少得很缓慢时,就认为进一步增大聚类数效果也并不能增强,存在得这个“肘点”就是最佳聚类数目,从一类到三类下降得很快,之后下降得很慢,所以最佳聚类个数选为三
另外也有现成的包(factoextra)可以调用
library(factoextra)
library(ggplot2)
set.seed(1234)
fviz_nbclust(dataset, kmeans, method = "wss") +
geom_vline(xintercept = 3, linetype = 2)
Loading required package: ggplot2
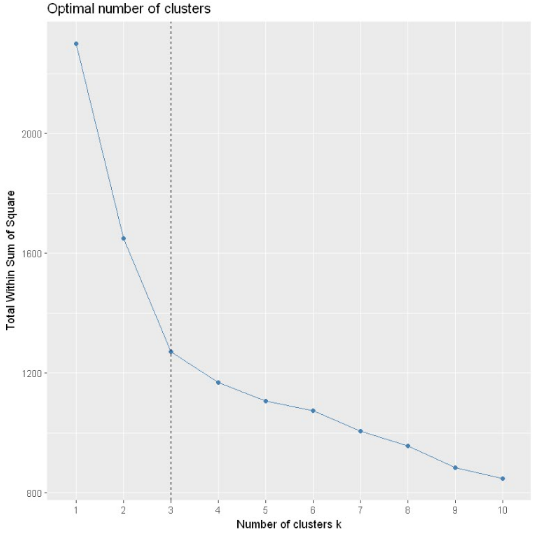
选定为3类为最佳聚类数目
用该包下的fviz_cluster函数可视化一下聚类结果
km.res <- kmeans(dataset,3)
fviz_cluster(km.res, data = dataset)
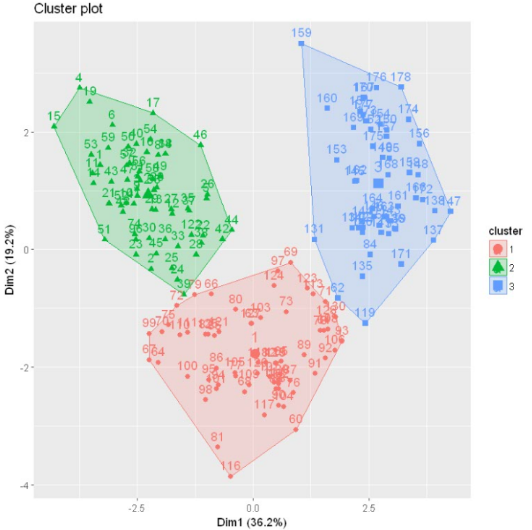
4. PAM(Partitioning Around Medoids) 围绕中心点的分割算法
k-means算法取得是均值,那么对于异常点其实对其的影响非常大,很可能这种孤立的点就聚为一类,一个改进的方法就是PAM算法,也叫k-medoids clustering
首先通过fpc包中的pamk函数得到最佳聚类数目
library(fpc)
pamk.best <- pamk(dataset)
pamk.best$nc
3
pamk函数不需要提供聚类数目,也会直接自动计算出最佳聚类数,这里也得到为3
得到聚类数提供给cluster包下的pam函数并进行可视化
library(cluster)
clusplot(pam(dataset, pamk.best$nc))
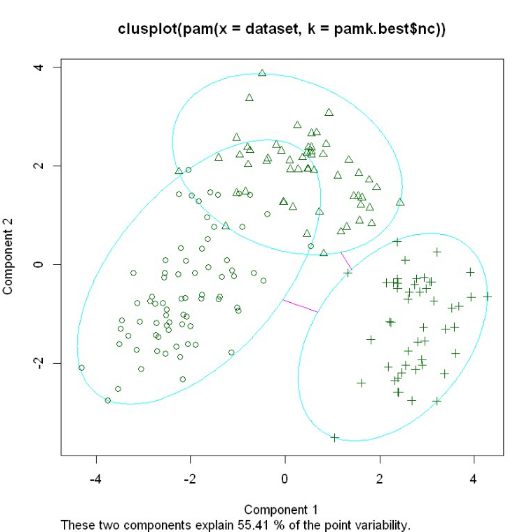
5.Calinsky criterion
这个评估标准定义[5]如下:
其中,k是聚类数,N是样本数,SSw是我们之前提到过的组内平方和误差, SSb是组与组之间的平方和误差,SSw越小,SSb越大聚类效果越好,所以Calinsky criterion值一般来说是越大,聚类效果越好
library(vegan)
ca_clust <- cascadeKM(dataset, 1, 10, iter = 1000)
ca_clust$results

可以看到该函数把组内平方和误差和Calinsky都计算出来了,可以看到calinski在聚类数为3时达到最大值。
calinski.best <- as.numeric(which.max(ca_clust$results[2,]))
calinski.best
3
画图出来观察一下
plot(fit, sortg = TRUE, grpmts.plot = TRUE)
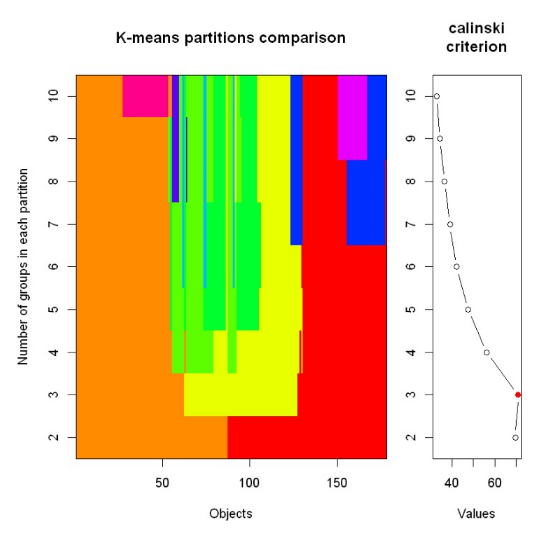
注意到那个红点就是对应的最大值,自带的绘图横轴纵轴取的可能不符合我们的直觉,把数据取出来自己单独画一下
calinski<-as.data.frame(ca_clust$results[2,])
calinski$cluster <- c(1:10)
library(ggplot2)
ggplot(calinski,aes(x = calinski[,2], y = calinski[,1]))+geom_line()
Warning message:
"Removed 1 rows containing missing values (geom_path)."
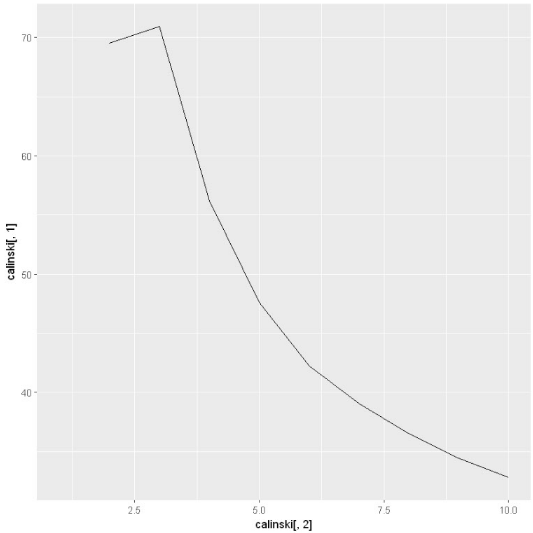
这个看上去直观多了。这就很清晰的可以看到在聚类数目为3时,calinski指标达到了最大值,所以最佳数目为3
6.Affinity propagation (AP) clustering
这个本质上是类似kmeans或者层次聚类一样,是一种聚类方法,因为不需要像kmeans一样提供聚类数,会自动算出最佳聚类数,因此也放到这里作为一种计算最佳聚类数目的方法。
AP算法的基本思想是将全部样本看作网络的节点,然后通过网络中各条边的消息传递计算出各样本的聚类中心。聚类过程中,共有两种消息在各节点间传递,分别是吸引度( responsibility)和归属度(availability) 。AP算法通过迭代过程不断更新每一个点的吸引度和归属度值,直到产生m个高质量的Exemplar(类似于质心),同时将其余的数据点分配到相应的聚类中[7]
library(apcluster)
ap_clust <- apcluster(negDistMat(r=2), dataset)
length(ap_clust@clusters)
15
该聚类方法推荐的最佳聚类数目为15,再用热力图可视化一下
heatmap(ap_clust)
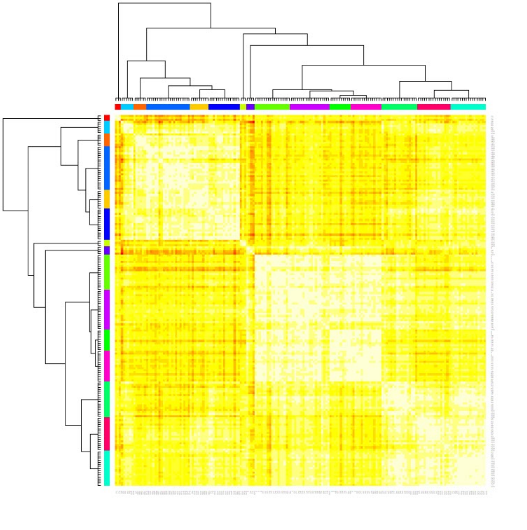
选x或者y方向看(对称),可以数出来“叶子节点”一共15个
7. 轮廓系数Average silhouette method
轮廓系数是类的密集与分散程度的评价指标。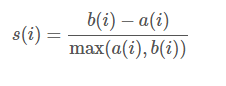 a(i)是测量组内的相似度,b(i)是测量组间的相似度,s(i)范围从-1到1,值越大说明组内吻合越高,组间距离越远——也就是说,轮廓系数值越大,聚类效果越好[9]
a(i)是测量组内的相似度,b(i)是测量组间的相似度,s(i)范围从-1到1,值越大说明组内吻合越高,组间距离越远——也就是说,轮廓系数值越大,聚类效果越好[9]
require(cluster)
library(factoextra)
fviz_nbclust(dataset, kmeans, method = "silhouette")
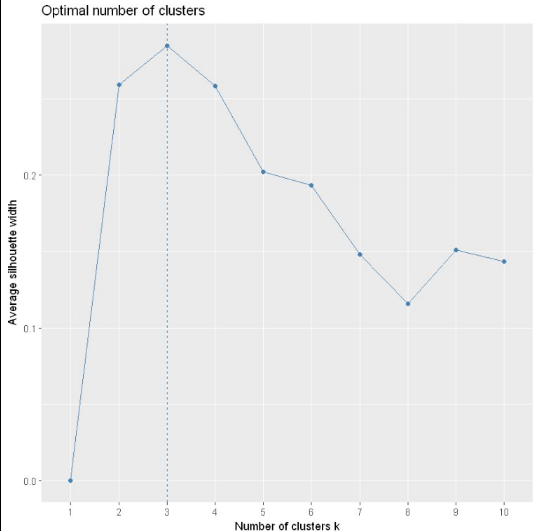
可以看到也是在聚类数为3时轮廓系数达到了峰值,所以最佳聚类数为3
8. Gap Statistic
之前我们提到了WSSE组内平方和误差,该种方法是通过找“肘点”来找到最佳聚类数,肘点的选择并不是那么清晰,因此斯坦福大学的Robert等教授提出了Gap Statistic方法,定义的Gap值为[9]
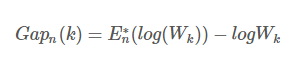
取对数的原因是因为Wk的值可能很大
通过这个式子来找出Wk跌落最快的点,Gap最大值对应的k值就是最佳聚类数
library(cluster)
set.seed(123)
gap_clust <- clusGap(dataset, kmeans, 10, B = 500, verbose = interactive())
gap_clust
Clustering Gap statistic ["clusGap"] from call:
clusGap(x = dataset, FUNcluster = kmeans, K.max = 10, B = 500, verbose = interactive())
B=500 simulated reference sets, k = 1..10; spaceH0="scaledPCA"
--> Number of clusters (method 'firstSEmax', SE.factor=1): 3
logW E.logW gap SE.sim
[1,] 5.377557 5.863690 0.4861333 0.01273873
[2,] 5.203502 5.758276 0.5547745 0.01420766
[3,] 5.066921 5.697322 0.6304006 0.01278909
[4,] 5.023936 5.651618 0.6276814 0.01243239
[5,] 4.993720 5.615174 0.6214536 0.01251765
[6,] 4.962933 5.584564 0.6216311 0.01165595
[7,] 4.943241 5.556310 0.6130690 0.01181831
[8,] 4.915582 5.531834 0.6162518 0.01139207
[9,] 4.881449 5.508514 0.6270646 0.01169532
[10,] 4.855837 5.487005 0.6311683 0.01198264
library(factoextra)
fviz_gap_stat(gap_clust)
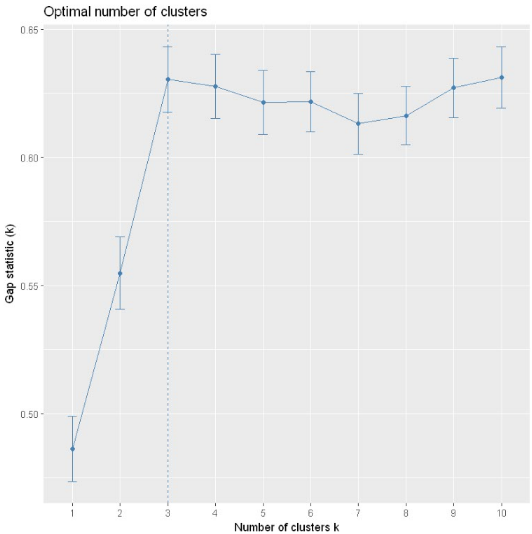
可以看到也是在聚类数为3的时候gap值取到了最大值,所以最佳聚类数为3
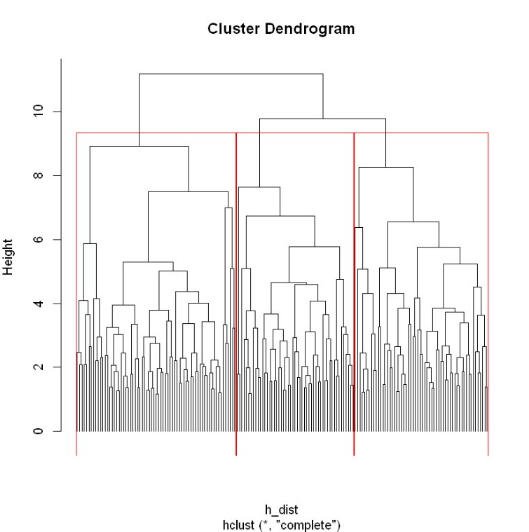
9.层次聚类
层次聚类是通过可视化然后人为去判断大致聚为几类,很明显在共同父节点的一颗子树可以被聚类为一个类
h_dist <- dist(as.matrix(dataset))
h_clust<-hclust(h_dist)
plot(h_clust, hang = -1, labels = FALSE)
rect.hclust(h_clust,3)
10.clustergram
最后一种算法是Tal Galili[10]大牛自己定义的一种聚类可视化的展示,绘制随着聚类数目的增加,所有成员是如何分配到各个类别的。该代码没有被制作成R包,可以去Galili介绍页面)里面的github地址找到源代码跑一遍然后就可以用这个函数了,因为源代码有点长我就不放博客里面了,直接放出运行代码的截图。
clustergram(dataset, k.range = 2:8, line.width = 0.004)
Loading required package: colorspace
Loading required package: plyr
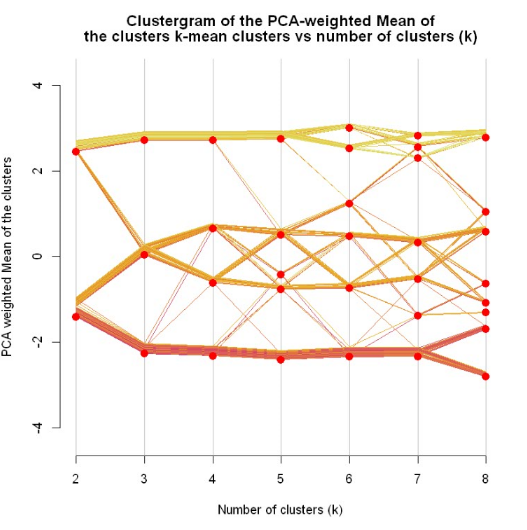
随着K的增加,从最开始的两类到最后的八类,图肯定是越到后面越密集。通过这个图判断最佳聚类数目的方法应该是看随着K每增加1,分出来的线越少说明在该k值下越稳定。比如k=7到k=8,假设k=7是很好的聚类数,那分成8类时应该可能只是某一类分成了两类,其他6类都每怎么变。反应到图中应该是有6簇平行线,有一簇分成了两股,而现在可以看到从7到8,线完全乱了,说明k=7时效果并不好。按照这个分析,k=3到k=4时,第一股和第三股几本没变,就第二股拆成了2类,所以k=3是最佳聚类数目
方法汇总与比较
wine数据集我们知道其实是分为3类的,以上10种判定方法中:
- 层次聚类和clustergram方法、肘点图法,需要人工判定,虽然可以得出大致的最佳聚类数,但算法本身不会给出最佳聚类数
- 除了Affinity propagation (AP) clustering 给出最佳聚类数为15,剩下6种全都是给出最佳聚类数为3
- 选用上次文本挖掘的矩阵进行分析(667*1623)
- mclust效果很差,14种模型只有6种有结果
- bclust报错
- SSE可以运行
- fpc包中的pamk函数聚成2类,明显不行
- Calinsky criterion聚成2类
- Affinity propagation (AP) clustering 聚成28类,相对靠谱
- 轮廓系数Average silhouette聚类2类
- gap-Statistic跑不出结果
可见上述方法中有的因为数据太大不能运行,有的结果很明显不对,一个可能是数据集的本身的原因(缺失值太多等)但是也告诉了我们在确定最佳聚类数目的时候需要多尝试几种方法,并没有固定的套路,然后选择一种可信度较高的聚类数目。
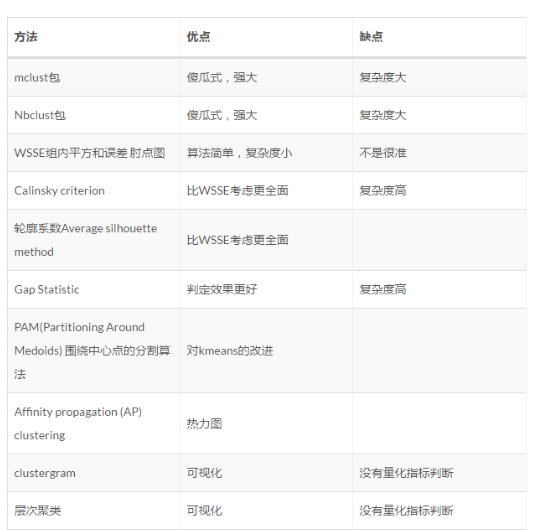
最后再把这10种方法总结一下:
参考文献
[1]R语言实战第二版
[2]Partitioning cluster analysis: Quick start guide - Unsupervised Machine Learning
[3]BIC:http://www.stat.washington.edu/raftery/Research/PDF/fraley1998.pdf
[4]Cluster analysis in R: determine the optimal number of clusters
[5]Calinski-Harabasz Criterion:Calinski-Harabasz criterion clustering evaluation object
[6]Determining the optimal number of clusters: 3 must known methods - Unsupervised Machine Learning
[7] affinity-propagation:聚类算法Affinity Propagation(AP)
[8]轮廓系数https://en.wikipedia.org/wiki/Silhouette(clustering))
[9]gap statistic-Tibshirani R, Walther G, Hastie T. Estimating the number of clusters in a data set via the gap statistic[J]. Journal of the Royal Statistical Society: Series B (Statistical Methodology), 2001, 63(2): 411-423.[10]ClustergramsClustergram: visualization and diagnostics for cluster analysis (R code)



 浙公网安备 33010602011771号
浙公网安备 33010602011771号