层次聚类
- stats::hclust
stats::dist
R使用dist()函数来计算距离,Usage: dist(x, method = "euclidean", diag = FALSE, upper = FALSE, p = 2)
- x: 是样本矩阵或者数据框
- method: 表示计算哪种距离
- euclidean 欧几里德距离,就是平方再开方。
- maximum 切比雪夫距离
- manhattan 绝对值距离
- canberra Lance 距离
- minkowski 明科夫斯基距离,使用时要指定p值
- binary 定性变量距离
- diag: 为TRUE的时候给出对角线上的距离
- upper: 为TURE的时候给出上三角矩阵上的值.
base::scale
R使用scale()函数对数据矩阵做中心化和标准化变换, Usage: scale(x, center = TRUE, scale = TRUE)
base::sweep
R语言中使用sweep() 函数对矩阵进行运算. Usage: sweep(x, MARGIN, STATS, FUN = "-", check.margin = TRUE, ...)
- MARGIN: 为1,表示行的方向上进行运算,为2表示列的方向上运算。
- STATS: 是运算的参数
- FUN为: 运算函数,默认是减法
stats::hclust
Usage:hclust(d, method = "complete", members = NULL)
- d:为距离矩阵
- method: 表示类的合并方法
- single: (单联动)最短距离法: 一个类中的点和另一个类中的点的最小距离
- complete: (全联动) 最长距离法: 一个类中的点和另一个类中的点的最大距离
- median: (平均联动) 中间距离法: 一个类中的点和另一个类中的点的平均距离
- mcquitty: 相似分析法
- average: 平均距离法,测量两类每对观测间的平均距离
- centroid:重心法, 两类间的距离定义为两类重心之间的距离
- ward:离差平方和法,基于方差分析思想,如果分类合理,则同类样品间离差平方和应当较小,类与类间离差平方和应当较大
示例 代码:
> library(stats)
> data=iris[,-5]
> dist.e=dist(data,method='euclidean')
> iris.hc <- hclust( dist.e,"single")
> plot( iris.hc, hang = -1,cex=.8) #hang小于0时,树将从底部画起
效果图如下:
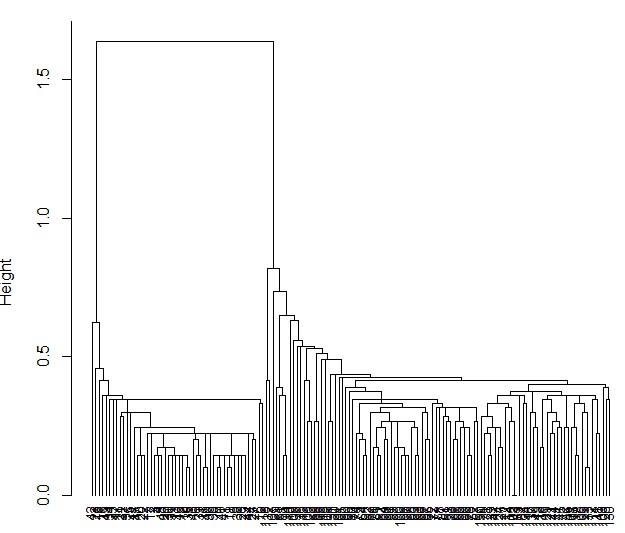
用矩形画出分为3类的区域
> #用矩形画出分为3类的区域
> re <- rect.hclust(iris.hc, k = 3)
> re
[[1]]
[1] 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 24 25 26 27 28 29 30 31 32 33 34 35 36 37 38 39 40 41 42 43 44 45 46
[47] 47 48 49 50
[[2]]
[1] 118 132
[[3]]
[1] 51 52 53 54 55 56 57 58 59 60 61 62 63 64 65 66 67 68 69 70 71 72 73 74 75 76 77 78 79 80 81 82 83 84 85
[36] 86 87 88 89 90 91 92 93 94 95 96 97 98 99 100 101 102 103 104 105 106 107 108 109 110 111 112 113 114 115 116 117 119 120 121
[71] 122 123 124 125 126 127 128 129 130 131 133 134 135 136 137 138 139 140 141 142 143 144 145 146 147 148 149 150
效果图
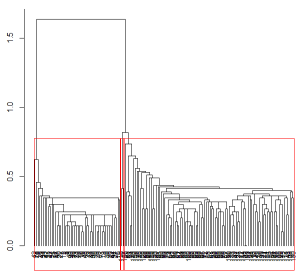
输出结果
> ##得到分为3类的数值,输出结果
> iris.id <- cutree(iris.hc, 3)
> table(iris.id, iris$Species)
iris.id setosa versicolor virginica
1 50 0 0
2 0 50 48
3 0 0 2
BIRCH
BIRCH(Balanced Iterative Reducing and Clustering Using Hierarchies)全称是:利用层次方法的平衡迭代规约和聚类。BIRCH是一种聚类算法,它最大的特点是能利用有限的内存资源完成对大数据集的高质量的聚类,同时通过单遍扫描数据集能最小化I/O代价。BIRCH算法有如下特点:
- BIRCH试图利用可用的资源来生成最好的聚类结果,给定有限的主存,一个重要的考虑是最小化I/O时间。
- BIRCH采用了一种多阶段聚类技术:数据集的单边扫描产生了一个基本的聚类,一或多遍的额外扫描可以进一步改进聚类质量。
- BIRCH是一种增量的聚类方法,因为它对每一个数据点的聚类的决策都是基于当前已经处理过的数据点,而不是基于全局的数据点。
- 如果簇不是球形的,BIRCH不能很好的工作,因为它用了半径或直径的概念来控制聚类的边界。
BIRCH算法中引入了两个概念:聚类特征和聚类特征树,详细参见:http://www.cnblogs.com/tiaozistudy/p/6129425.html
说明:birch 包在cran上很多年没更新,如果要使用,必须安装R3.0以前的版本,此处先略过,参见:https://cran.r-project.org/src/contrib/Archive/birch/
CURE
CURE(Clustering Using REprisentatives)算法即使用代表点的聚类方法。该算法先把每个数据点看成一类,然后合并距离最近的类直至类个数为所要求的个数为止。CURE算法将传统对类的 表示方法进行了改进,回避了用所有点或用中心和半径来表示一个类,而是从每一个类中抽取固定数量、分布较好的点作为描述此类的代表点,并将这些点乘以一个 适当的收缩因子,使它们更靠近类的中心点。将一个类用代表点表示,使得类的外延可以向非球形的形状扩展,从而可调整类的形状以表达那些非球形的类。另外, 收缩因子的使用减小了嗓音对聚类的影响。CURE算法采用随机抽样与分割相结合的办法来提高算法的空间和时间效率,并且在算法中用了堆和K-d树结构来提 高算法效率
说明:未找着下载包
参考资料:
- http://www.cnblogs.com/tiaozistudy/p/6129425.html
- https://www.douban.com/note/274189091/
- http://artax.karlin.mff.cuni.cz/r-help/library/birch/html/birch.html
- https://www.douban.com/note/514091031/


