R语言生态学JAGS模拟数据、线性回归、Cormack-Jolly-Seber (CJS) 模型拟合MCMC 估计动物存活率和可视化
原文链接:http://tecdat.cn/?p=24721
原文出处:拓端数据部落公众号
本文,我通过两个种群生态学家可能感兴趣的例子来说明使用“JAGS”来模拟数据:首先是线性回归,其次是估计动物存活率(公式化为状态空间模型)。
最近,我一直在努力模拟来自复杂分层模型的数据。我现在正在使用 JAGS。
模拟数据 JAGS 很方便,因为你可以使用(几乎)相同的代码进行模拟和推理,并且你可以在相同的环境(即JAGS)中进行模拟研究(偏差、精度、区间覆盖 )。
线性回归示例
我们首先加载本教程所需的包:
-
library(R2jags)
-
-
然后直接切入正题,让我们从线性回归模型生成数据。使用一个 data 块,并将参数作为数据传递。
-
data{
-
# 似然函数:
-
for (i in 1:N){
-
y[i] ~ # tau是精度(1/方差)。
-
-
}
-
这里, alpha 和 beta 是截距和斜率、 tau 方差的精度或倒数、 y 因变量和 x 解释变量。
我们为用作数据的模型参数选择一些值:
-
-
# 模拟的参数
-
N # 样本
-
x <- 1:N # 预测因子
-
alpha # 截距
-
beta # 斜率
-
sigma# 残差sd
-
1/(sigma*sigma) # 精度
-
# 在模拟步骤中,参数被当作数据处理
-
现在运行 JAGS; 请注意,我们监控因变量而不是参数,就像我们在进行标准推理时所做的那样:
-
# 运行结果
-
out

输出有点乱,需要适当格式化:
-
# 重新格式化输出
-
mcmc(out)

dim
![]()
dat

现在让我们将我们用来模拟的模型拟合到我们刚刚生成的数据中。不再赘述,假设读者熟悉 JAGS 线性回归。
-
-
# 用BUGS语言指定模型
-
model <-
-
-
for (i in 1:N){
-
y[i] ~ dnorm(mu[i], tau) # tau是精度(1/方差)
-
-
-
alpha 截距
-
beta # 斜率
-
sigma # 标准差
-
-
-
# 数据
-
dta <- list(y = dt, N = length(at), x = x)
-
-
# 初始值
-
inits
-
-
-
# MCMC设置
-
ni <- 10000
-
-
-
# 从R中调用JAGS
-
jags()
-
![]()

让我们看看结果并与我们用来模拟数据的参数进行比较(见上文):
-
# 总结后验
-
print(res)
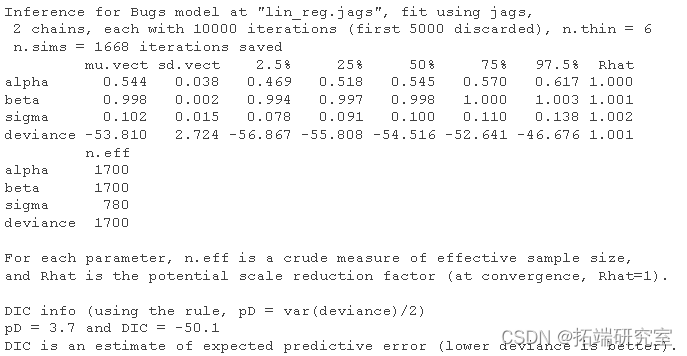
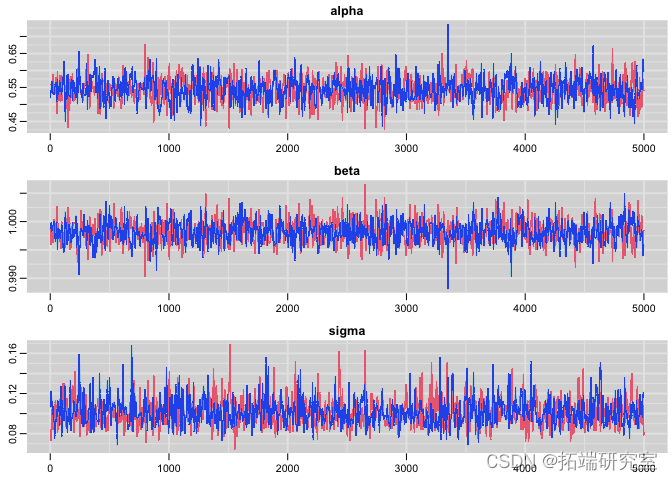
检查收敛:
-
# 追踪图
-
plot(res)
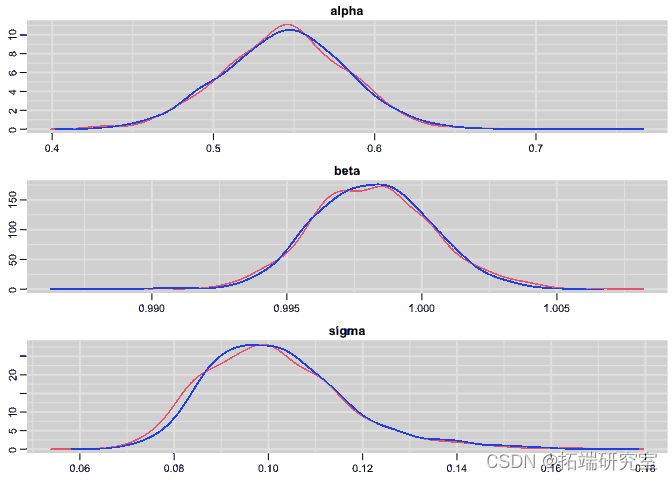
绘制回归参数和残差标准差的后验分布:
-
# 后验分布
-
plot(res)
模拟示例
我现在说明如何使用 JAGS 来模拟来自具有恒定生存和重新捕获概率的模型的数据。我假设读者熟悉这个模型及其作为状态空间模型的公式。
让我们模拟一下!
-
-
# 恒定的生存和重新捕获概率
-
for (i in 1:nd){
-
for (t in f:(on-1)){
-
-
#概率
-
for (i in 1:nid){
-
# 定义潜伏状态和第一次捕获时的观察值
-
z[i,f[i] <- 1
-
mu2[i,1] <- 1 * z[i,f[i] # 在第一次捕获时检测为1("以第一次捕获为条件")。
-
# 然后处理以后的情况
-
for (t in (f[i]+1):non){
-
# 状态进程
-
mu1[i,t] <- phi * z
-
# 观察过程
-
mu2[i,t] <- p * z
-
-
让我们为参数选择一些值并将它们存储在数据列表中:
-
-
# 用于模拟的参数
-
n = 100 # 个体的数量
-
meanhi <- 0.8 # 存活率
-
meap <- 0.6 # 重捕率
-
data<-list
现在运行 JAGS:
out 
格式化输出:
as.mcmc(out)
![]()
head(dat)

我只监测了检测和非检测,但也可以获得状态的模拟值,即个人在每种情况下是生是死。你只需要修改对JAGS 的调用 monitor=c("y","x") 并相应地修改输出。
现在我们将 Cormack-Jolly-Seber (CJS) 模型拟合到我们刚刚模拟的数据中,假设参数不变:
-
-
# 倾向性和约束
-
for (i in 1:nd){
-
for (t in f[i]:(nn-1)){
-
-
-
mehi ~ dunif(0, 1) # 平均生存率的先验值
-
Me ~ dunif(0, 1) # 平均重捕的先验值
-
# 概率
-
for (i in 1:nd){
-
# 定义第一次捕获时的潜伏状态
-
z[i]] <- 1
-
for (t in (f[i]+1):nions){
-
# 状态过程
-
z[i,t] ~ dbern(mu1[i,t])
-
# 观察过程
-
y[i,t] ~ dbern(mu2[i,t])
准备数据:
-
-
# 标记的场合的向量
-
gerst <- function(x) min(which(x!=0))
-
# 数据
-
jagta
-
-
# 初始值
-
for (i in 1:dim]){
-
min(which(ch[i,]==1))
-
max(which(ch[i,]==1))
-
-
function(){list(meaphi, mep , z ) }
-
我们想对生存和重新捕获的概率进行推断:
标准 MCMC 设置:
ni <- 10000
准备运行 JAGS!
-
# 从R中调用JAGS
-
jags(nin = nb, woy = getwd() )

总结后验并与我们用来模拟数据的值进行比较:
print(cj3)

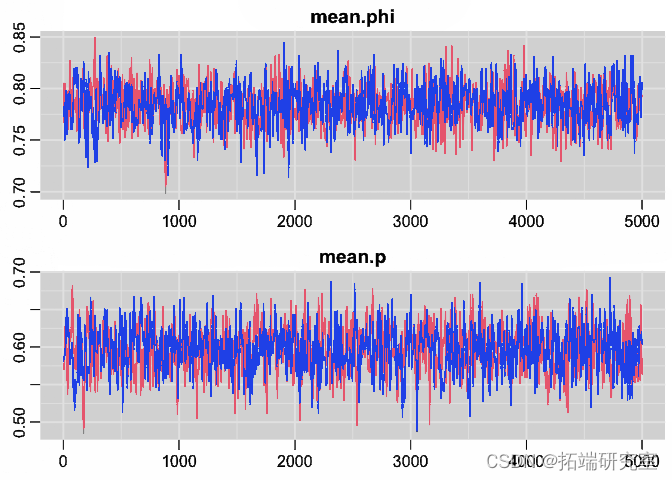
非常接近!
跟踪图
trplot
后验分布图
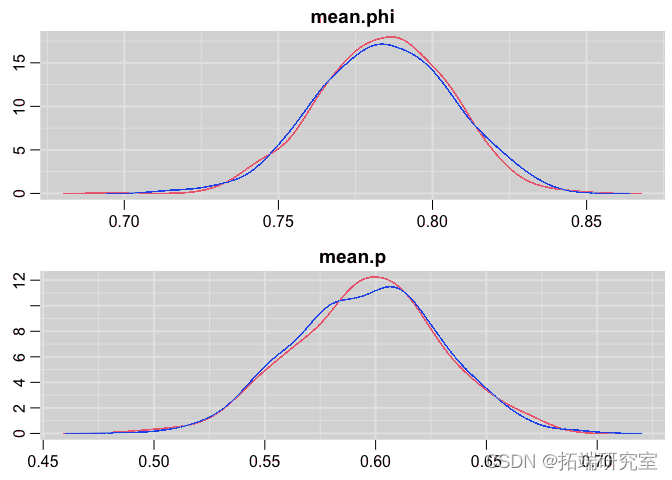
denplot

最受欢迎的见解
4.R语言中的block Gibbs吉布斯采样贝叶斯多元线性回归



 浙公网安备 33010602011771号
浙公网安备 33010602011771号