拓端数据tecdat|R语言基于Bagging分类的逻辑回归(Logistic Regression)、决策树、森林分析心脏病患者
原文链接:http://tecdat.cn/?p=22448
原文出处:拓端数据部落公众号
今天,我们将看下bagging 技术里面的启发式算法。
通常,bagging 与树有关,用于生成森林。但实际上,任何类型的模型都有可能使用bagging 。回顾一下,bagging意味着 "boostrap聚合"。因此,考虑一个模型m:X→Y。让 ![]() 表示从样本中得到的m的估计
表示从样本中得到的m的估计
![]()
现在考虑一些boostrap样本,![]() ,i是从{1,⋯,n}中随机抽取的。 基于该样本,估计
,i是从{1,⋯,n}中随机抽取的。 基于该样本,估计![]() 。然后抽出许多样本,考虑获得的估计值的一致性,使用多数规则,或使用概率的平均值(如果考虑概率主义模型)。因此
。然后抽出许多样本,考虑获得的估计值的一致性,使用多数规则,或使用概率的平均值(如果考虑概率主义模型)。因此
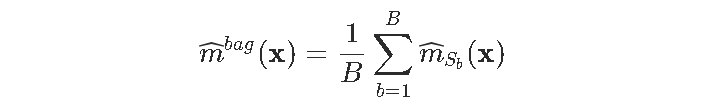
Bagging逻辑回归
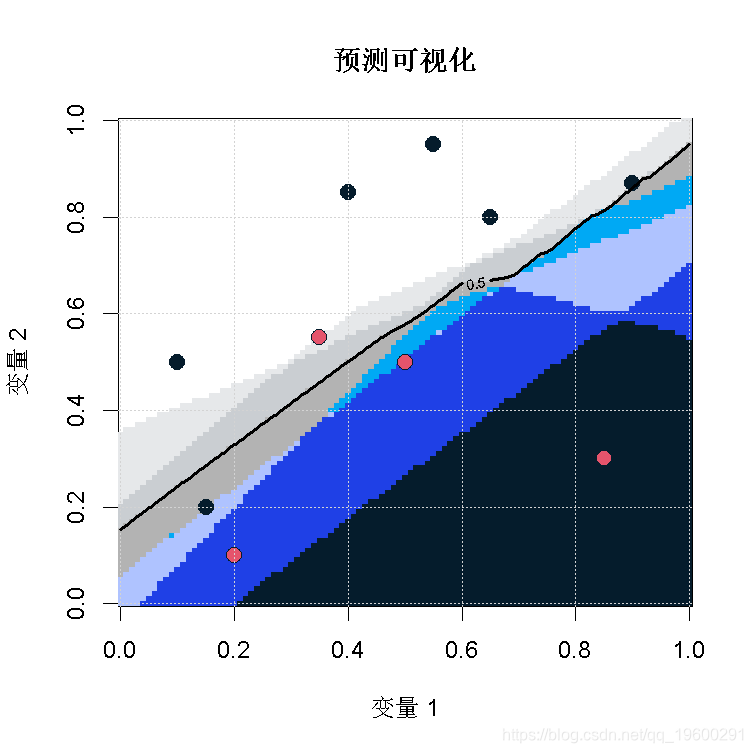
考虑一下逻辑回归的情况。为了产生一个bootstrap样本,自然要使用上面描述的技术。即随机抽取一对(yi,xi),均匀地(概率为![]() )替换。这里考虑一下小数据集。对于bagging部分,使用以下代码
)替换。这里考虑一下小数据集。对于bagging部分,使用以下代码
-
-
for(s in 1:1000){
-
df_s = df[sample(1:n,size=n,replace=TRUE)
-
logit[s]= glm(y~., df_s, family=binomial
然后,我们应该在这1000个模型上进行汇总,获得bagging的部分。
-
-
unlist(lapply(1:1000,function(z) predict(logit[z],nnd))}
我们现在对任何新的观察都有一个预测
-
vv = outer(vu,vu,(function(x,y) mean(pre(c(x,y)))
-
contour(vu,vu,vv,levels = .5,add=TRUE)
Bagging逻辑回归
另一种可用于生成bootstrap样本的技术是保留所有的xi,但对其中的每一个,都(随机地)抽取一个y的值,其中有
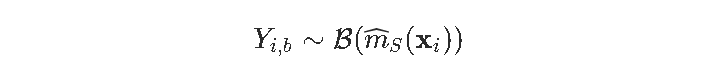
因此
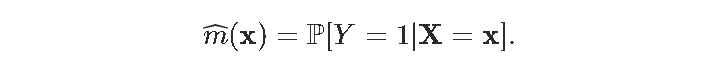
因此,现在Bagging算法的代码是
-
glm(y~x1+x2, df, family=binomial)
-
for(s in 1:100)
-
y = rbinom(size=1,prob=predict(reg,type="response")
-
L_logit[s] = glm(y~., df_s, family=binomial)
bagging算法的agg部分保持不变。在这里我们获得
-
vv = outer(vu,vu,(function(x,y) mean(pre(c(x,y)))))
-
contour(vu,vu,vv,levels = .5,add=TRUE)
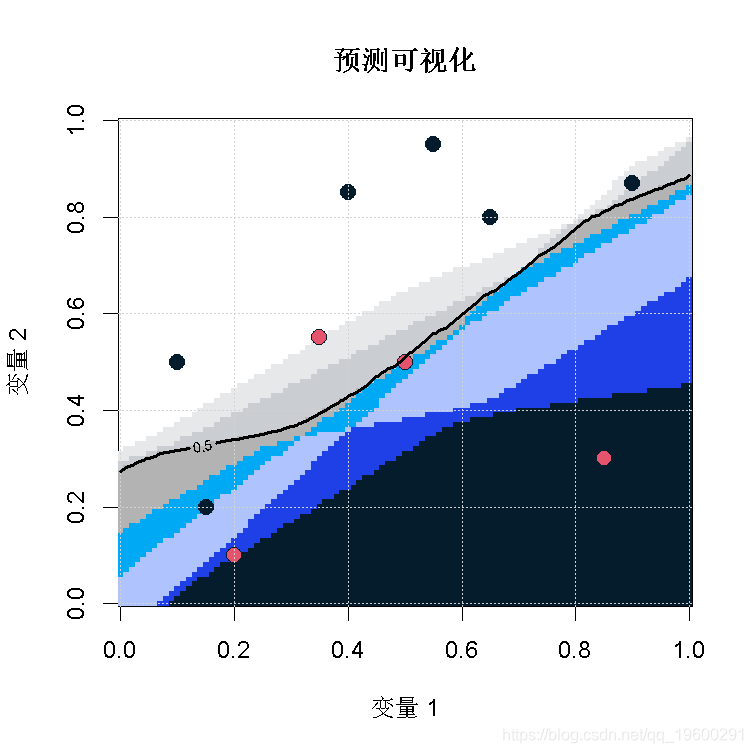
当然,我们可以使用该代码,检查预测获得我们的样本中的观察。
在这里考虑心肌梗塞数据。
数据
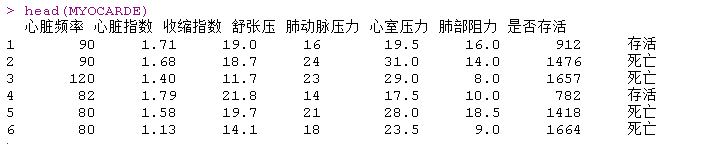
我们使用心脏病数据,预测急诊病人的心肌梗死,包含变量:
- 心脏指数
- 心搏量指数
- 舒张压
- 肺动脉压
- 心室压力
- 肺阻力
- 是否存活
其中我们有急诊室的观察结果,对于心肌梗塞,我们想了解谁存活下来了,得到一个预测模型
-
reg = glm(as.factor(PRO)~., carde, family=binomial)
-
for(s in 1:1000){
-
L_logit[s] = glm(as.factor(PRO)~., my_s, family=binomial)
-
}
-
-
unlist(lapply(1:100,predict(L_logit[z],newdata=d,type="response")}
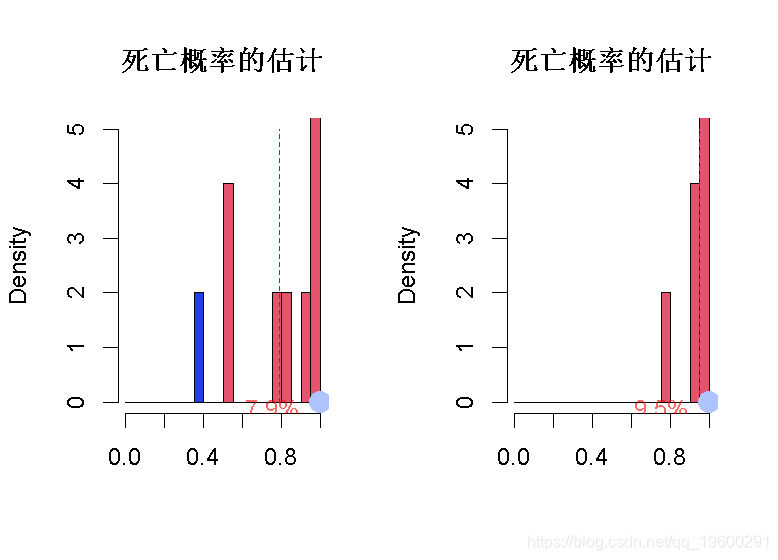
对于第一个观察,通过我们的1000个模拟数据集,以及我们的1000个模型,我们得到了以下死亡概率的估计。
-
v_x = p(x)
-
hist(v_x,proba=TRUE,breaks=seq(,by.05),=",="",
-
segments(mean(v_x),0,mean(v_x,5="=2)
-
因此,对于第一个观察,在78.8%的模型中,预测的概率高于50%,平均概率实际上接近75%。
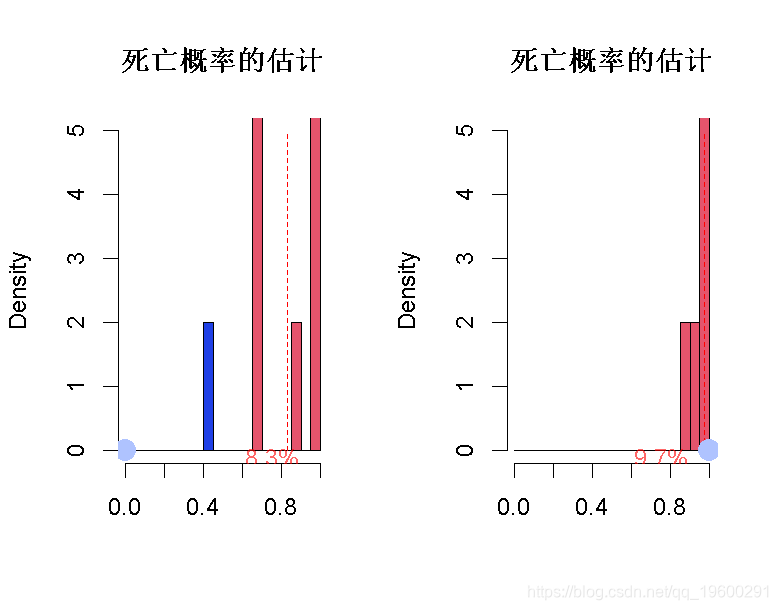
或者,对于样本22,预测与第一个非常接近。
-
histo(23)
-
histo(11)
我们在此观察到
Bagging决策树
Bagging是由Leo Breiman于1994年在Bagging Predictors中介绍的。如果说第一节描述了这个程序,那么第二节则介绍了 "Bagging分类树"。树对于解释来说是不错的,但大多数时候,它们是相当差的预测模型。Bagging的想法是为了提高分类树的准确性。
bagging 的想法是为了生成大量的树
-
-
for(i in 1:12)
-
set.seed(sed[i])
-
idx = sample(1:n, size=n, replace=TRUE)
-
cart = rpart(PR~., md[idx,])
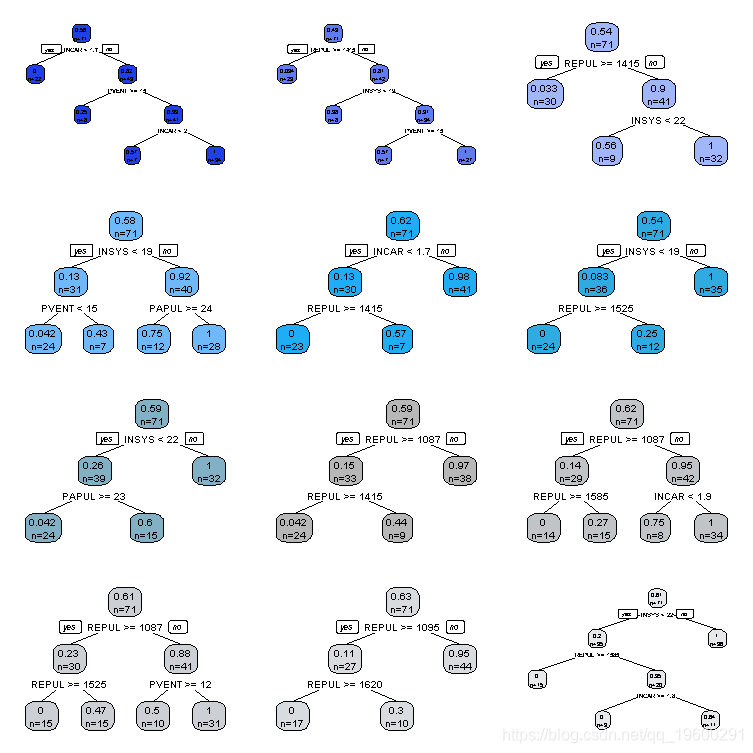
这个策略其实和以前一样。对于bootstrap部分,将树存储在一个列表中
-
for(s in 1:1000)
-
idx = sample(1:n, size=n, replace=TRUE)
-
L_tree[[s]] = rpart(as.(PR)~.)
而对于汇总部分,只需取预测概率的平均值即可
-
p = function(x){
-
unlist(lapply(1:1000,function(z) predict(L_tree[z],newdata,)[,2])
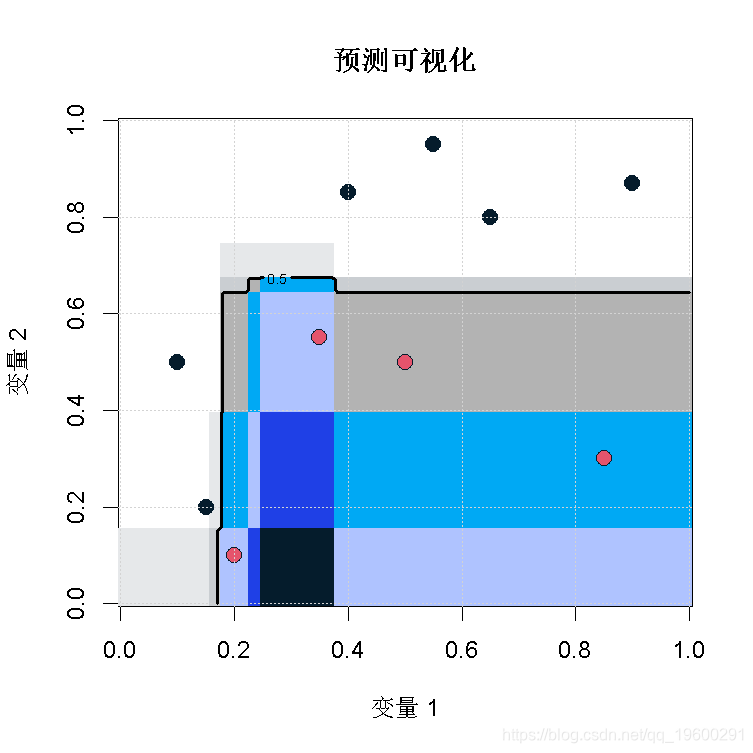
因为在这个例子中,我们无法实现预测的可视化,让我们在较小的数据集上运行同样的代码。
-
-
for(s in 1:1000){
-
idx = sample(1:n, size=n, replace=TRUE)
-
L_tree[s] = rpart(y~x1+x2,
-
}
-
unlist(lapply(1:1000,function(z) predict(L_tree[[z]])
-
outer(vu,vu,Vectorize(function(x,y) mean(p(c(x,y)))
从bagging到森林
在这里,我们生成了很多树,但它并不是严格意义上的随机森林算法,正如1995年在《随机决策森林》中介绍的那样。实际上,区别在于决策树的创建。当我们有一个节点时,看一下可能的分割:我们考虑所有可能的变量,以及所有可能的阈值。这里的策略是在p中随机抽取k个变量(当然k<p,例如k=sqrt{p})。这在高维度上是有趣的,因为在每次分割时,我们应该寻找所有的变量和所有的阈值,而这可能需要相当长的时间(尤其是在bootstrap 程序中,目标是长出1000棵树)。

最受欢迎的见解
1.用机器学习识别不断变化的股市状况—隐马尔科夫模型(HMM)的应用





