拓端数据tecdat|R语言贝叶斯推断与MCMC:实现Metropolis-Hastings 采样算法示例
原文链接:http://tecdat.cn/?p=21545
示例1:使用MCMC的指数分布采样
任何MCMC方案的目标都是从“目标”分布产生样本。在这种情况下,我们将使用平均值为1的指数分布作为我们的目标分布。所以我们从定义目标密度开始:
-
target = function(x){
-
if(x<0){
-
return(0)}
-
else {
-
return( exp(-x))
-
}
-
}
定义了函数之后,我们现在可以用它来计算几个值(只是为了说明函数的概念):
target(1)[1] 0.3678794target(-1)[1] 0接下来,我们将规划一个Metropolis-Hastings方案,从与目标成比例的分布中进行抽样
-
x[1] = 3 #这只是一个起始值,我设置为3
-
for(i in 2:1000){
-
A = target(proposedx)/target(currentx)
-
if(runif(1)<A){
-
x[i] = proposedx # 接受概率min(1,a)
-
} else {
-
x[i] = currentx #否则“拒绝”行动,保持原样
-
}
注意,x是马尔可夫链的实现。我们可以画几个x的图:

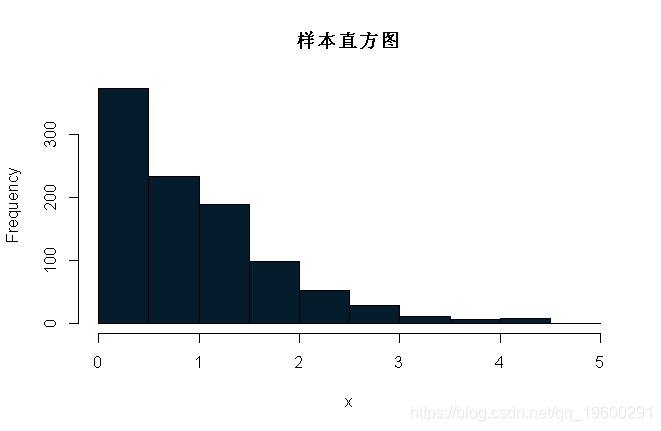
我们可以将其封装在一个mcmc函数中,以使代码更整洁,这样更改起始值和提议分布更容易
-
-
for(i in 2:niter){
-
currentx = x[i-1]
-
proposedx = rnorm(1,mean=currentx,sd=proposalsd)
-
A = target(proposedx)/target(currentx)
-
if(runif(1)<A){
-
x[i] = proposedx # 接受概率min(1,a)
-
} else {
-
x[i] = currentx # 否则“拒绝”行动,保持原样
-
}
现在我们将运行MCMC方案3次,看看结果有多相似:
-
z1=MCMC(1000,3,1)
-
z2=MCMC(1000,3,1)
-
z3=MCMC(1000,3,1)
-
-
plot(z1,type="l")

-
par(mfcol=c(3,1)) #告诉R将3个图形放在一个页面上
-
-
hist(z1,breaks=seq(0,maxz,length=20))
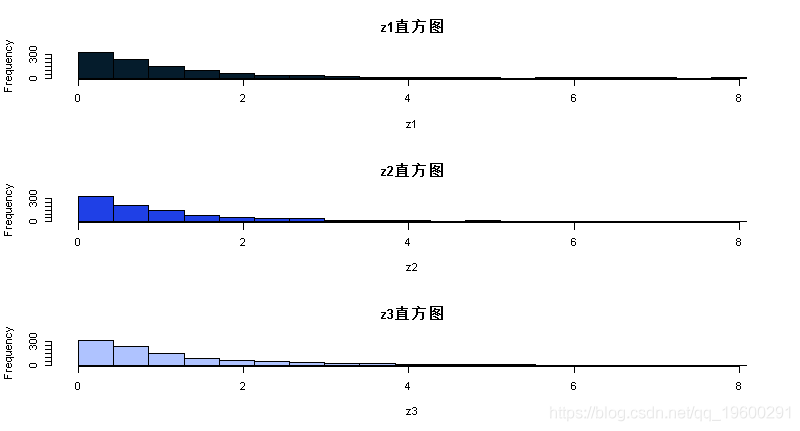
练习
使用函数easyMCMC了解以下内容:
- 不同的起始值如何影响MCMC方案?
- 较大/较小的提案标准差有什么影响?
- 尝试将目标函数更改为
-
target = function(x){
-
-
return((x>0 & x <1) + (x>2 & x<3))
-
}
这个目标看起来像什么?如果提议sd太小怎么办?(例如,尝试1和0.1)
例2:估计等位基因频率
在对双等位基因座的基因型(如具有AA和AA等位基因的基因座)进行建模时,一个标准的假设是群体是“随机”的。这意味着如果p是等位基因AA的频率,那么基因型![]() 和
和![]() 将分别具有频率
将分别具有频率![]() 和
和![]() 。
。
p一个简单的先验是假设它在[0,1]上是均匀的。 假设我们抽样n个个体,观察![]() 基因型
基因型![]() 、
、![]() 基因型
基因型![]() 和
和![]() 基因型
基因型![]() 。
。
下面的R代码给出了一个简短的MCMC例程,可以从p的后验分布中进行采样。请尝试遍历该代码,看看它是如何工作的。
-
prior = function(p){
-
if((p<0) || (p>1)){ # || 这里意思是“或”
-
return(0)}
-
else{
-
return(1)}
-
}
-
-
likelihood = function(p, nAA, nAa, naa){
-
return(p^(2*nAA) * (2*p*(1-p))^nAa * (1-p)^(2*naa))
-
}
-
-
psampler = function(nAA, nAa, naa){
-
-
for(i in 2:niter){
-
-
if(runif(1)<A){
-
p[i] = newp # 接受概率min(1,a)
-
} else {
-
p[i] = currentp # 否则“拒绝”行动,保持原样
-
}
对![]() 运行此样本。
运行此样本。
现在用一些R代码来比较后验样本和理论后验样本(在这种情况下可以通过分析获得;因为我们观察到121个As和79个as,在200个样本中,p的后验样本是β(121+1,79+1)。
-
-
hist(z,prob=T)
-
lines(x,dbeta(x,122, 80)) # 在直方图上叠加β密度
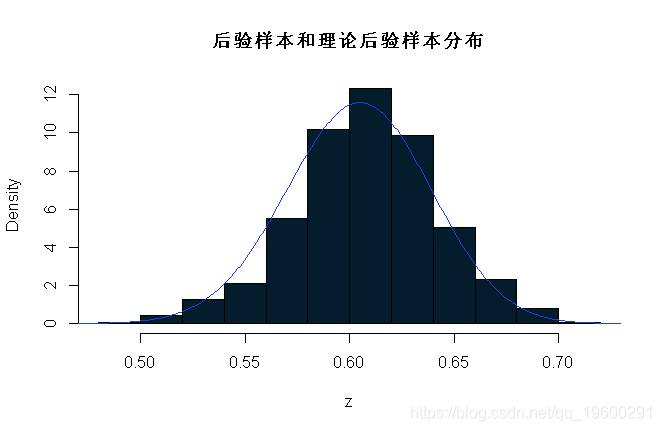
您也可能希望将前5000 z的值丢弃为“burnin”(预烧期)。这里有一种方法,在R中仅选择最后5000 z
hist(z[5001:10000])
练习
研究起点和提案标准偏差如何影响算法的收敛性。
例3:估计等位基因频率和近交系数
一个复杂一点的替代方法是假设人们有一种倾向,即人们会与比“随机”关系更密切的其他人近交(例如,在地理结构上的人口中可能会发生这种情况)。一个简单的方法是引入一个额外的参数,即“近亲繁殖系数”f,并假设![]() 和
和![]() 基因型有频率
基因型有频率
![]() 和
和![]() 。
。
在大多数情况下,将f作为种群特征来对待是很自然的,因此假设f在各个位点上是恒定的。
请注意,f和p都被约束为介于0和1之间(包括0和1)。对于这两个参数中的每一个,一个简单的先验是假设它们在[0,1]上是独立的。 假设我们抽样n个个体,观察![]() 基因型
基因型![]() 、
、![]() 基因型
基因型![]() 和
和![]() 基因型
基因型![]() 。
。
练习:
- 编写一个短的MCMC程序,从f和p的联合分布中取样。
-
sampler = function(){
-
-
f[1] = fstartval
-
p[1] = pstartval
-
for(i in 2:niter){
-
currentf = f[i-1]
-
currentp = p[i-1]
-
newf = currentf +
-
newp = currentp +
-
-
}
-
return(list(f=f,p=p)) # 返回一个包含两个名为f和p的元素的“list”
-
}
- 使用此样本获得f和p的点估计(例如,使用后验平均数)和f和p的区间估计(例如,90%后验置信区间),数据:
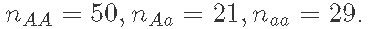
附录:GIBBS采样
您也可以用Gibbs采样器解决这个问题
为此,您将想要使用以下“潜在变量”表示模型:
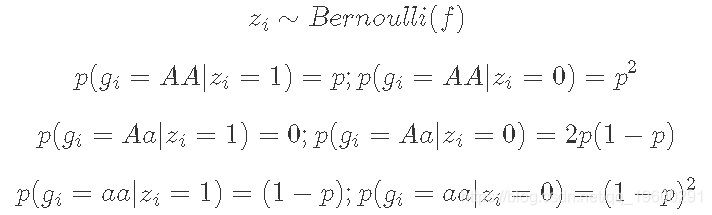
将zi相加得到与上述相同的模型:
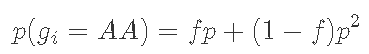

最受欢迎的见解
1.使用R语言进行METROPLIS-IN-GIBBS采样和MCMC运行
3.R语言实现MCMC中的Metropolis–Hastings算法与吉布斯采样
4.R语言BUGS JAGS贝叶斯分析 马尔科夫链蒙特卡洛方法(MCMC)采样
5.R语言中的block Gibbs吉布斯采样贝叶斯多元线性回归
7.R语言用Rcpp加速Metropolis-Hastings抽样估计贝叶斯逻辑回归模型的参数





