拓端数据tecdat|R语言RStan贝叶斯示例:重复试验模型和种群竞争模型Lotka Volterra
原文链接:http://tecdat.cn/?p=19737
Stan是一种用于指定统计模型的概率编程语言。Stan通过马尔可夫链蒙特卡罗方法(例如No-U-Turn采样器,一种汉密尔顿蒙特卡洛采样的自适应形式)为连续变量模型提供了完整的贝叶斯推断。
可以通过R使用rstan 包来调用Stan,也可以 通过Python使用 pystan 包。这两个接口都支持基于采样和基于优化的推断,并带有诊断和后验分析。
在本文中,简要展示了Stan的主要特性。还显示了两个示例:第一个示例与简单的伯努利模型相关,第二个示例与基于常微分方程的Lotka-Volterra模型有关。
什么是Stan?
- Stan是命令式概率编程语言。
- Stan程序定义了概率模型。
- 它声明数据和(受约束的)参数变量。
- 它定义了对数后验。
- Stan推理:使模型拟合数据并做出预测。
- 它可以使用马尔可夫链蒙特卡罗(MCMC)进行完整的贝叶斯推断。
- 使用变分贝叶斯(VB)进行近似贝叶斯推断。
- 最大似然估计(MLE)用于惩罚最大似然估计。
Stan计算什么?
- 得出后验分布 。
- MCMC采样。
- 绘制
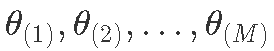 ,其中每个绘制
,其中每个绘制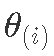 都按后验概率
都按后验概率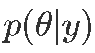 的边缘分布。
的边缘分布。 - 使用直方图,核密度估计等进行绘图
安装 rstan
要在R中运行Stan,必须安装 rstan C ++编译器。在Windows上, Rtools 是必需的。
最后,安装 rstan:
install.packages(rstan)Stan中的基本语法
定义模型
Stan模型由六个程序块定义 :
- 数据(必填)。
- 转换后的数据。
- 参数(必填)。
- 转换后的参数。
- 模型(必填)。
- 生成的数量。
数据块读出的外部信息。
-
data {
-
int N;
-
int x[N];
-
int offset;
-
}
变换后的数据 块允许数据的预处理。
-
transformed data {
-
int y[N];
-
for (n in 1:N)
-
y[n] = x[n] - offset;
-
}
参数 块定义了采样的空间。
-
parameters {
-
real<lower=0> lambda1;
-
real<lower=0> lambda2;
-
}
变换参数 块定义计算后验之前的参数处理。
-
transformed parameters {
-
-
lambda = lambda1 + lambda2;
-
}
在 模型 块中,我们定义后验分布。
-
model {
-
y ~ poisson(lambda);
-
lambda1 ~ cauchy(0, 2.5);
-
lambda2 ~ cauchy(0, 2.5);
-
}
最后, 生成的数量 块允许进行后处理。
-
generated quantities {
-
int x_predict;
-
x_predict = poisson_rng(lambda) + offset;
-
}
类型
Stan有两种原始数据类型, 并且两者都是有界的。
- int 是整数类型。
- real 是浮点类型。
-
int<lower=1> N;
-
-
real<upper=5> alpha;
-
real<lower=-1,upper=1> beta;
-
-
real gamma;
-
real<upper=gamma> zeta;
实数扩展到线性代数类型。
-
vector[10] a; // 列向量
-
matrix[10, 1] b;
-
-
row_vector[10] c; // 行向量
-
matrix[1, 10] d;
整数,实数,向量和矩阵的数组均可用。
-
real a[10];
-
-
vector[10] b;
-
-
matrix[10, 10] c;
Stan还实现了各种 约束 类型。
-
simplex[5] theta; // sum(theta) = 1
-
-
ordered[5] o; // o[1] < ... < o[5]
-
positive_ordered[5] p;
-
-
corr_matrix[5] C; // 对称和
-
cov_matrix[5] Sigma; // 正定的
关于Stan的更多信息
所有典型的判断和循环语句也都可用。
-
if/then/else
-
-
for (i in 1:I)
-
-
while (i < I)
有两种修改 后验的方法。
-
y ~ normal(0, 1);
-
-
target += normal_lpdf(y | 0, 1);
-
-
# 新版本的Stan中已弃用:
-
increment_log_posterior(log_normal(y, 0, 1))
而且许多采样语句都是 矢量化的。
-
parameters {
-
real mu[N];
-
real<lower=0> sigma[N];
-
}
-
-
model {
-
// for (n in 1:N)
-
// y[n] ~ normal(mu[n], sigma[n]);
-
-
y ~ normal(mu, sigma); // 向量化版本
-
}
贝叶斯方法
概率是 认知的。例如, 约翰·斯图亚特·米尔 (John Stuart Mill)说:
事件的概率不是事件本身,而是我们或其他人期望发生的情况的程度。每个事件本身都是确定的,不是可能的;如果我们全部了解,我们应该或者肯定地知道它会发生,或者它不会。
对我们来说,概率表示对它发生的期望程度。
概率可以量化不确定性。
Stan的贝叶斯示例:重复试验模型
我们解决一个小例子,其中的目标是给定从伯努利分布中抽取的随机样本,以估计缺失参数的后验分布 ![]() (成功的机会)。
(成功的机会)。
步骤1:问题定义
在此示例中,我们将考虑以下结构:
-
数据:
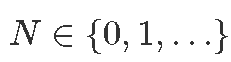 ,试用次数。
,试用次数。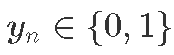 ,即试验n的结果 (已知的建模数据)。
,即试验n的结果 (已知的建模数据)。
-
参数:
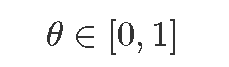
- 先验分布
![]()
- 概率
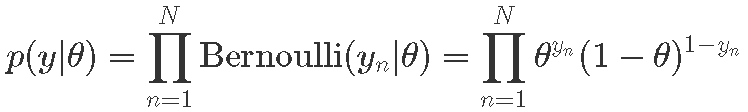
- 后验分布
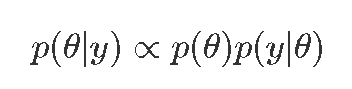
步骤2:Stan
我们创建Stan程序,我们将从R中调用它。
-
-
data {
-
int<lower=0> N; // 试验次数
-
int<lower=0, upper=1> y[N]; // 试验成功
-
}
-
-
-
model {
-
theta ~ uniform(0, 1); // 先验
-
y ~ bernoulli(theta); // 似然
-
}
步骤3:数据
在这种情况下,我们将使用示例随机模拟一个随机样本,而不是使用给定的数据集。
-
# 生成数据
-
-
y = rbinom(N, 1, 0.3)
-
y
## [1] 0 0 0 1 1 0 0 0 0 0 1 0 0 1 0 0 0 0 0 1根据数据计算 MLE作为样本均值:
## [1] 0.25步骤4:rstan使用贝叶斯后验估计
最后一步是使用R中的Stan获得我们的估算值。
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
...
-
-
## SAMPLING FOR MODEL '6dcfbccbf2f063595ccc9b93f383e221' NOW (CHAIN 4).
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
-
# 提取后验抽样
-
# 计算后均值(估计)
-
mean(theta_draws)
## [1] 0.2715866# 计算后验区间
-
ggplot(theta_draws_df, aes(x=theta)) +
-
geom_histogram(bins=20, color="gray")
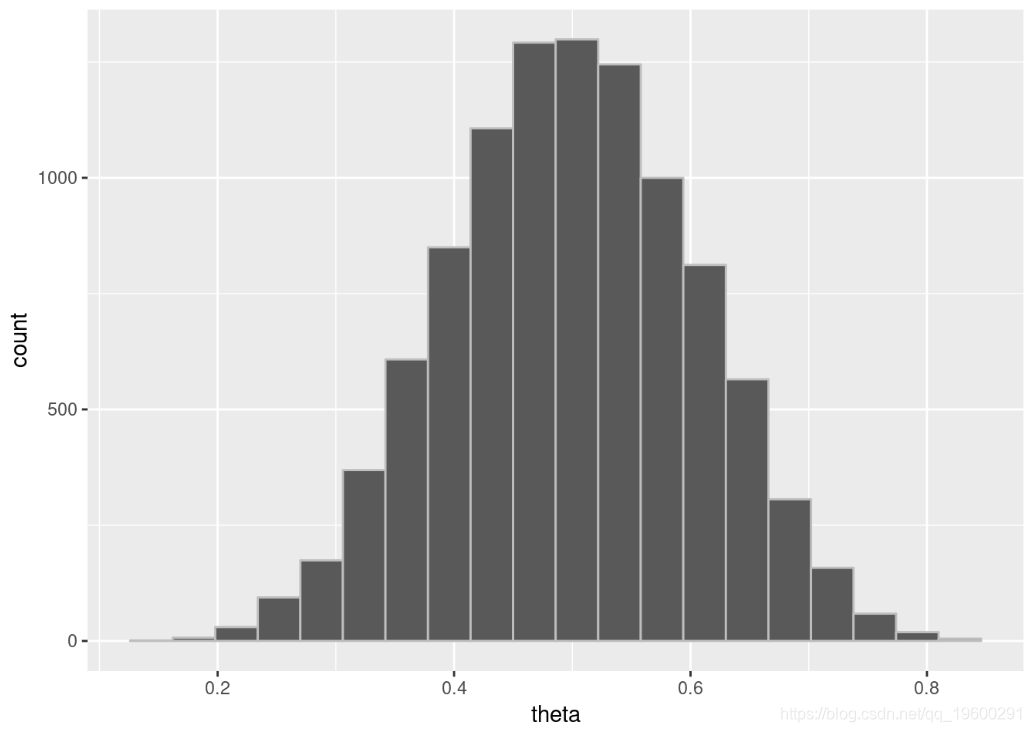
RStan:MAP,MLE
Stan的估算优化;两种观点:
- 最大后验估计(MAP)。
- 最大似然估计(MLE)。
optimizing(model, data=c("N", "y"))
种群竞争模型 ---Lotka-Volterra模型
- 洛特卡(Lotka,1925)和沃尔泰拉(Volterra,1926)制定了参数化微分方程,描述了食肉动物和猎物的竞争种群。
- 完整的贝叶斯推断可用于估计未来(或过去)的种群数量。
- Stan用于对统计模型进行编码并执行完整的贝叶斯推理,以解决从噪声数据中推断参数的逆问题。
在此示例中,我们希望根据公司每年收集的毛皮数量,将模型拟合到1900年至1920年之间各自种群的加拿大猫科食肉动物和野兔猎物。
数学模型
我们表示U(t)和V(t)作为猎物和捕食者种群数量 分别。与它们相关的微分方程为:
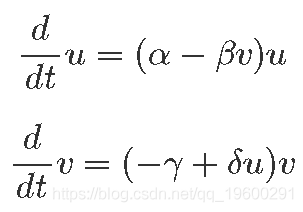
这里:
- α:猎物增长速度。
- β:捕食引起的猎物减少速度。
- γ:自然的捕食者减少速度。
- δ:捕食者从捕食中增长速度。
stan中的Lotka-Volterra
-
real[] dz_dt(data real t, // 时间
-
real[] z, // 系统状态
-
real[] theta, // 参数
-
data real[] x_r, // 数值数据
-
data int[] x_i) // 整数数据
-
{
-
real u = z[1]; // 提取状态
-
real v = z[2];
观察到已知变量:
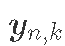 :表示在时间
:表示在时间 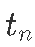 的
的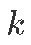 物种数量
物种数量
必须推断未知变量):
- 初始状态:
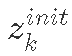 :k的初始物种数量。
:k的初始物种数量。 - 后续状态
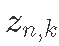 :在时间t的物种数量k。
:在时间t的物种数量k。 - 参量
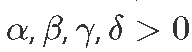 。
。
假设误差是成比例的(而不是相加的):

等效:
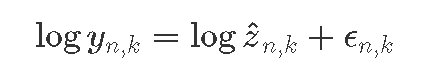
与
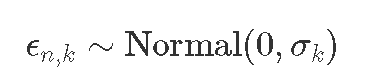
建立模型
已知常数和观测数据的变量。
-
data {
-
int<lower = 0> N; // 数量测量
-
real ts[N]; // 测量次数>0
-
real y0[2]; // 初始数量
-
real<lower=0> y[N,2]; // 后续数量
-
}
未知参数的变量。
-
parameters {
-
real<lower=0> theta[4]; // alpha, beta, gamma, delta
-
real<lower=0> z0[2]; // 原始种群
-
real<lower=0> sigma[2]; // 预测误差
-
}
先验分布和概率。
-
model {
-
// 先验
-
sigma ~ lognormal(0, 0.5);
-
theta[{1, 3}] ~ normal(1, 0.5);
-
-
// 似然(对数正态)
-
for (k in 1:2) {
-
y0[k] ~ lognormal(log(z0[k]), sigma[k]);
我们必须为预测的总体定义变量 :
- 初始种群(
z0)。 - 初始时间(
0.0),时间(ts)。 - 参数(
theta)。 - 最大迭代次数(
1e3)。
Lotka-Volterra参数估计
print(fit, c("theta", "sigma"), probs=c(0.1, 0.5, 0.9))获得结果:
-
mean se_mean sd 10% 50% 90% n_eff Rhat
-
## theta[1] 0.55 0 0.07 0.46 0.54 0.64 1168 1
-
## theta[2] 0.03 0 0.00 0.02 0.03 0.03 1305 1
-
## theta[3] 0.80 0 0.10 0.68 0.80 0.94 1117 1
-
## theta[4] 0.02 0 0.00 0.02 0.02 0.03 1230 1
-
## sigma[1] 0.29 0 0.05 0.23 0.28 0.36 2673 1
-
## sigma[2] 0.29 0 0.06 0.23 0.29 0.37 2821 1
分析所得结果:
- Rhat接近1表示收敛;n_eff是有效样本大小。
- 10%,后验分位数;例如
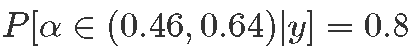 。
。 - 后验均值是贝叶斯点估计:α=0.55。
- 后验平均估计的标准误为0。
- α的后验标准偏差为0.07。

最受欢迎的见解
4.R语言中的block Gibbs吉布斯采样贝叶斯多元线性回归



