拓端tecdat|R语言代写绘制生存曲线估计|生存分析|如何R作生存曲线图
原文链接:http://tecdat.cn/?p=6087
根据生存曲线的估计,可以推断出相比组之间存活时间的差异,因此生存曲线非常有用,几乎可以在每个生存分析中看到。
例
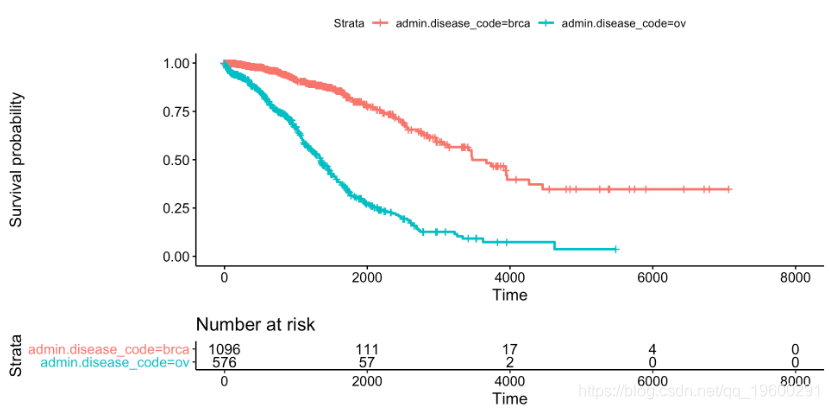
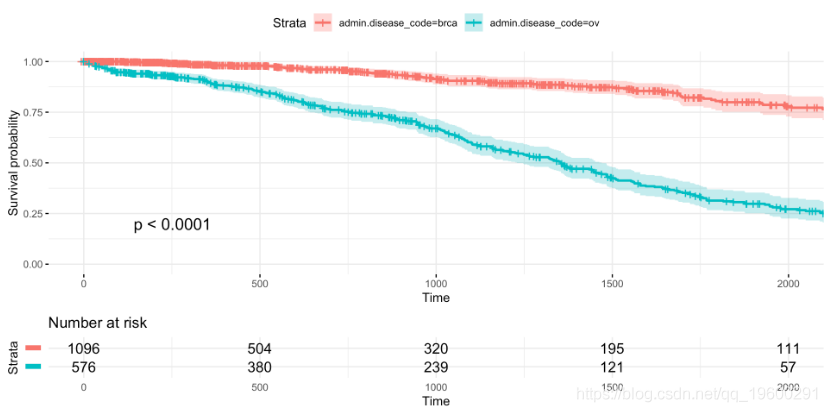
我们可以创建简单的生存曲线估计。让我们来看看患有卵巢癌(卵巢浆液性囊腺癌)和患有乳腺癌(乳腺浸润癌)的患者之间存活时间的差异 。
这个简单的图表以优雅的方式呈现了生存概率的估计值,该估计值取决于根据癌症类型分组的癌症诊断天数和信息风险集表,其中显示了在特定时间段内观察的患者数量。生存分析是一个特定的数据分析领域,因为事件数据的审查时间,因此风险集大小是视觉推理的必要条件。
每个参数都在相应的注释中描述,但我想强调xlim控制X轴限制但不影响生存曲线的参数,这些参数考虑了所有可能的时间。
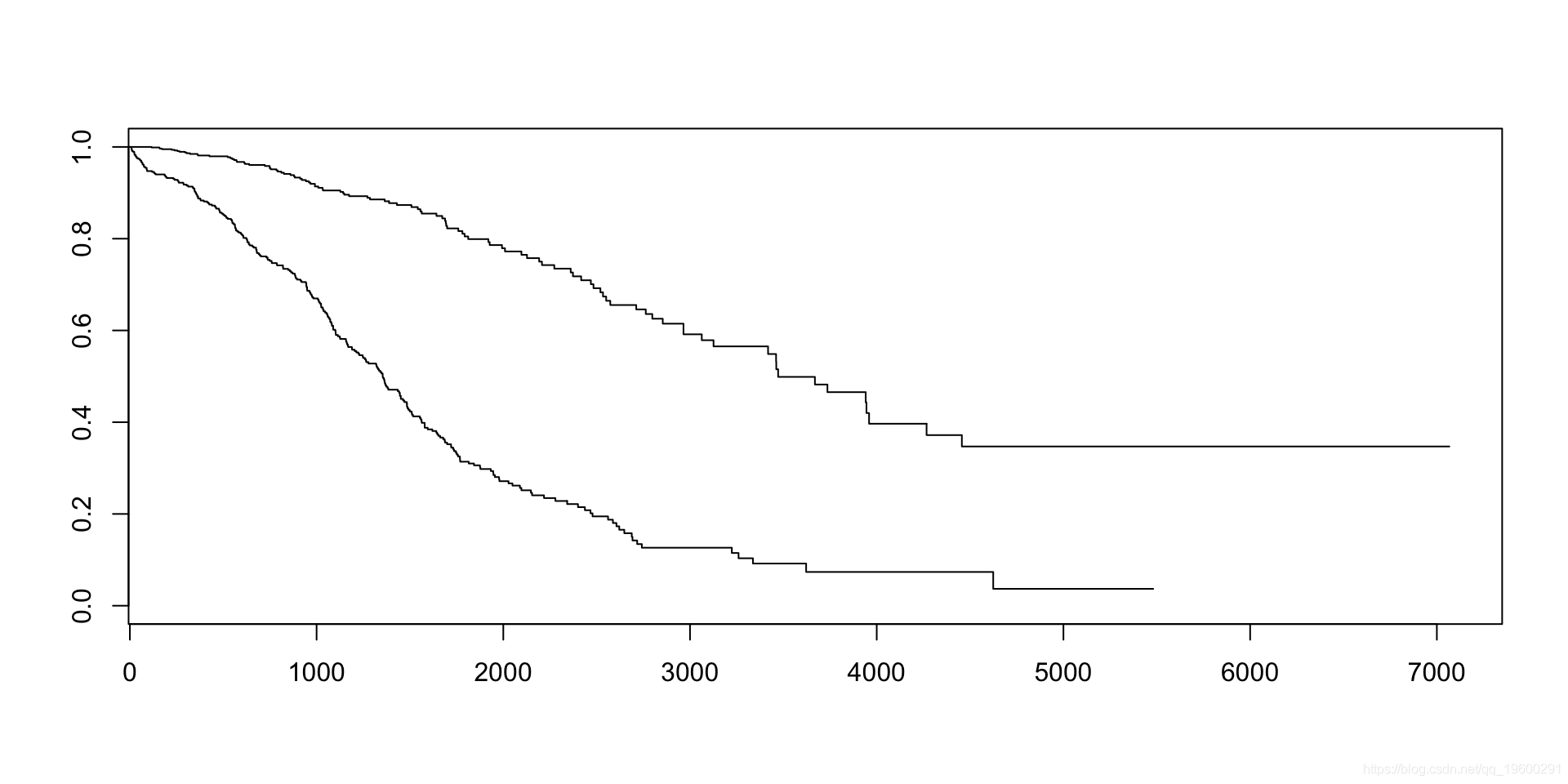
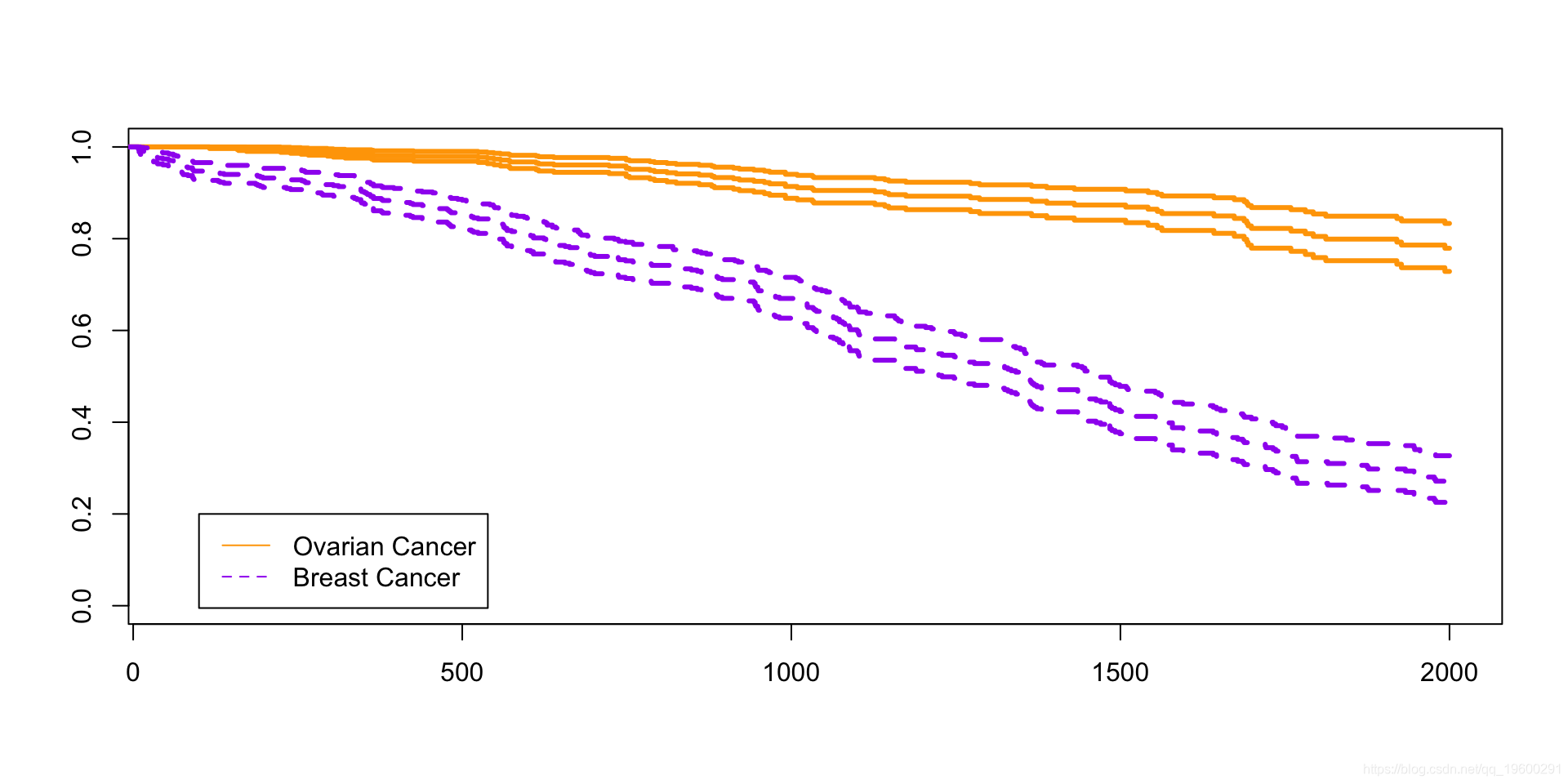
比较
基础包
看起来很漂亮.....
相关文章
3.R语言如何在生存分析与Cox回归中计算IDI,NRI指标
8.R语言如何找到患者数据中具有差异的指标?(PLS—DA分析)
9.R语言中的生存分析Survival analysis晚期肺癌患者4例
▍关注我们
【大数据部落】第三方数据服务提供商,提供全面的统计分析与数据挖掘咨询服务,为客户定制个性化的数据解决方案与行业报告等。
▍咨询链接:http://y0.cn/teradat
▍联系邮箱:3025393450@qq.com