全网最好看的单细胞umap图绘制教程
全网最好看的单细胞umap图绘制教程
作者按
大家或许都曾被Nature, Science上的单细胞umap图吸引过,不免心生崇拜。在这里,我们将介绍一种简单方便的顶刊级umap图可视化
全文字数|预计阅读时间: 2000|5min
——Starlitnightly(星夜)
环境加载
我们先导入一些必须的依赖包
import omicverse as ov
import scanpy as sc
import matplotlib.pyplot as plt
ov.ov_plot_set()
____ _ _ __
/ __ \____ ___ (_)___| | / /__ _____________
/ / / / __ `__ \/ / ___/ | / / _ \/ ___/ ___/ _ \
/ /_/ / / / / / / / /__ | |/ / __/ / (__ ) __/
\____/_/ /_/ /_/_/\___/ |___/\___/_/ /____/\___/
Version: 1.6.3, Tutorials: https://omicverse.readthedocs.io/
读取数据
我们选取了一个结直肠癌的数据作为演示。
adata=ov.read('crc_50000.h5ad')
adata
AnnData object with n_obs × n_vars = 50000 × 25121
obs: 'n_genes', 'doublet_score', 'predicted_doublet', 'n_genes_by_counts', 'total_counts', 'total_counts_mt', 'pct_counts_mt', 'leiden', 'scsa_celltype', 'major_celltype', 'scsa_true_celltype'
var: 'gene_ids', 'n_cells', 'mt', 'n_cells_by_counts', 'mean_counts', 'pct_dropout_by_counts', 'total_counts', 'highly_variable', 'means', 'dispersions', 'dispersions_norm'
uns: 'dendrogram_leiden', 'hvg', 'leiden', 'leiden_colors', 'leiden_sizes', 'log1p', 'major_celltype_colors', 'neighbors', 'paga', 'pca', 'rank_genes_groups', 'scrublet', 'scsa_celltype_colors', 'umap'
obsm: 'X_pca', 'X_umap'
obsp: 'connectivities', 'distances'
首先我们先尝试一下最基础的umap图绘制可视化
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],
title='',
show=False,
size=10,
frameon='small',
ax=ax
)
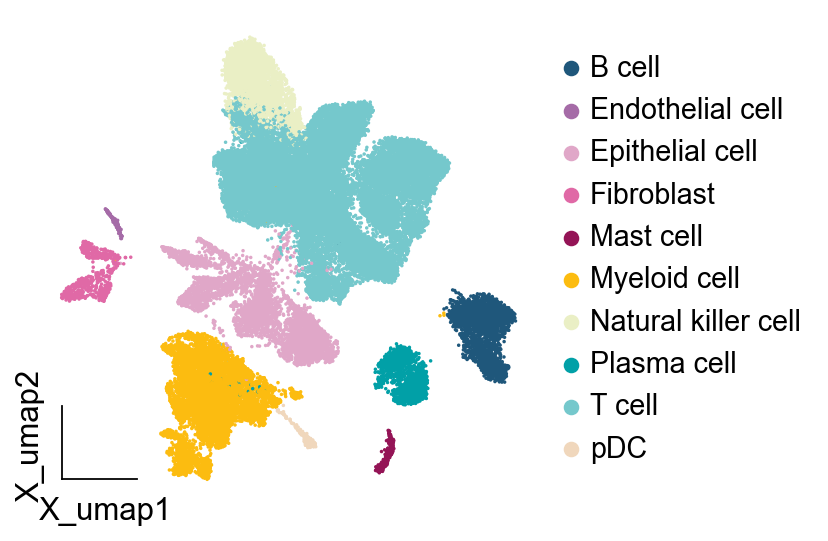
细胞可视化顺序
我们发现效果平平无奇对吧,接下来,我们首先需要修改的是,细胞可视化的顺序,将免疫细胞放到一块,将其他细胞放到另一块
adata.obs['major_celltype'].cat.categories
Index(['B cell', 'Endothelial cell', 'Epithelial cell', 'Fibroblast',
'Mast cell', 'Myeloid cell', 'Natural killer cell', 'Plasma cell',
'T cell', 'pDC'],
dtype='object')
new_order=['B cell', 'Plasma cell','T cell', 'Natural killer cell','Myeloid cell','pDC','Mast cell',
'Endothelial cell', 'Epithelial cell', 'Fibroblast',
]
adata.obs['major_celltype']=adata.obs['major_celltype'].cat.reorder_categories(new_order)
此时我们再可视化,会发现细胞的排序变成了我们指定的顺序
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False,
frameon='small',
size=10,
ax=ax
)
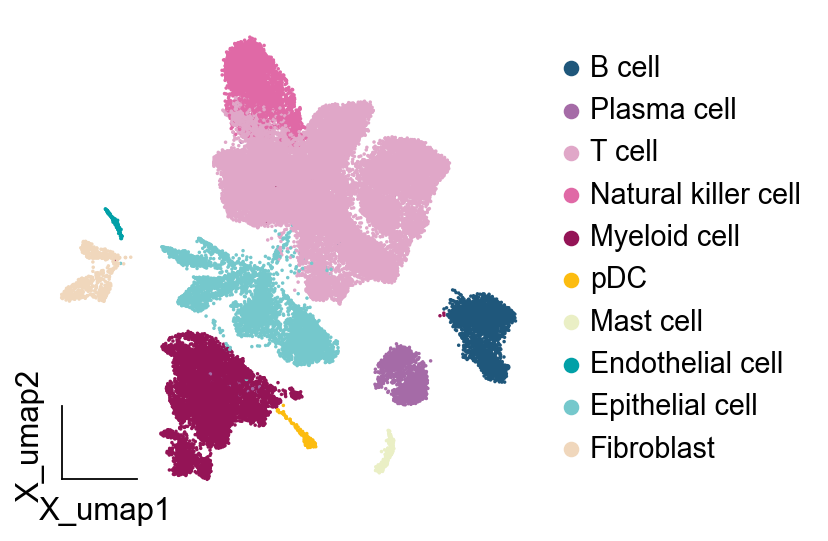
接下来我们再指定细胞类型的颜色,这里有两个方法:
- 第一个方法是我们使用颜色条顺序来指定
- 第二个方法是直接指定细胞类型的颜色
我们先尝试第一个方法,我们希望B细胞是蓝色,T细胞是红色,髓系细胞是绿色,其他细胞是黄色,那么我们根据数量指定颜色条。
需要注意的是,我们在omicverse中提供了5种顶刊级别的颜色条调用
- 红色:
ov.pl.red_color - 蓝色:
ov.pl.blue_color - 橙色:
ov.pl.orange_color - 绿色:
ov.pl.green_color - 紫色:
ov.pl.purple_color
如果你有其他的颜色需求也可以自行指定,颜色条是list格式,例如palette=['#000000','#FFFFFF']
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False,
size=10,
frameon='small',
palette=ov.pl.blue_color[4:6]+ov.pl.purple_color[1:3]+ov.pl.green_color[:3]+ov.pl.orange_color[:],
ax=ax
)
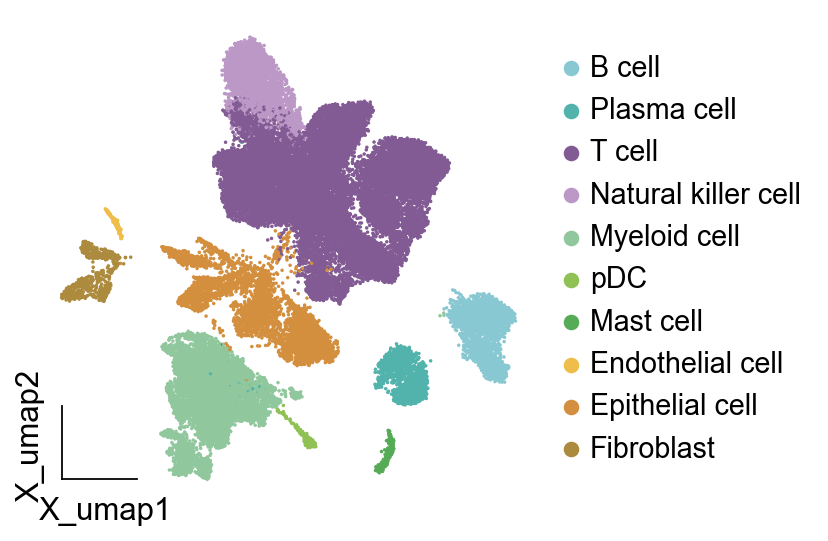
我们发现颜色变成了我们指定的颜色类型
第二种方法也很简单,我们指定每一种细胞类型的颜色然后进行可视化就好了
color_dict={
'B cell':ov.pl.blue_color[4],
'Plasma cell':ov.pl.blue_color[5],
'T cell':ov.pl.purple_color[0],
'Natural killer cell':ov.pl.purple_color[1],
'Myeloid cell':ov.pl.green_color[0],
'pDC':ov.pl.green_color[1],
'Mast cell':ov.pl.green_color[2],
'Endothelial cell':ov.pl.red_color[0],
'Epithelial cell':ov.pl.red_color[1],
'Fibroblast':ov.pl.red_color[2]
}
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False,
frameon='small',
size=10,
palette=color_dict,
ax=ax
)
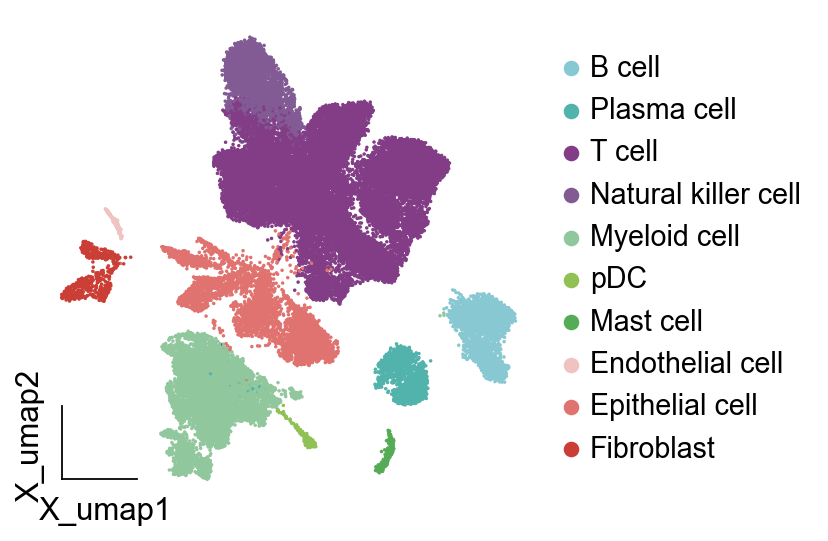
细胞标签优化
我们接下来需要优化的是细胞类型的展示,我们上面绘制的细胞的标签在右边,对于读者而言,需要一个一个去颜色对应的。在Nature最新的出版需求中,希望作者们考虑到色弱等人士的需求,因此我们可以直接在细胞umap图上绘制细胞类型。
需要注意的是,scanpy默认的细胞类型在umap图上的可视化可能出现遮挡,我们在omicverse中提供了新的函数ov.pl.embedding_adjust
from matplotlib import patheffects
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False, legend_loc=None, add_outline=False,
frameon='small',legend_fontoutline=2,ax=ax
)
ov.pl.embedding_adjust(
adata,
basis="X_umap",
groupby='major_celltype',
ax=ax,
adjust_kwargs=dict(arrowprops=dict(arrowstyle='-', color='black')),
text_kwargs=dict(fontsize=12 ,weight='bold',
path_effects=[patheffects.withStroke(linewidth=2, foreground='w')] ),
)
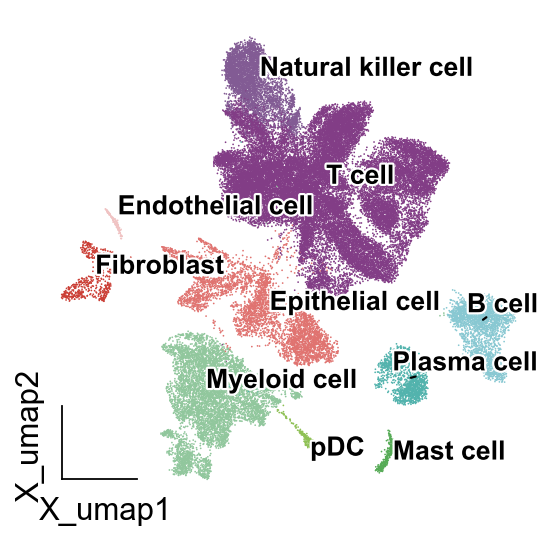
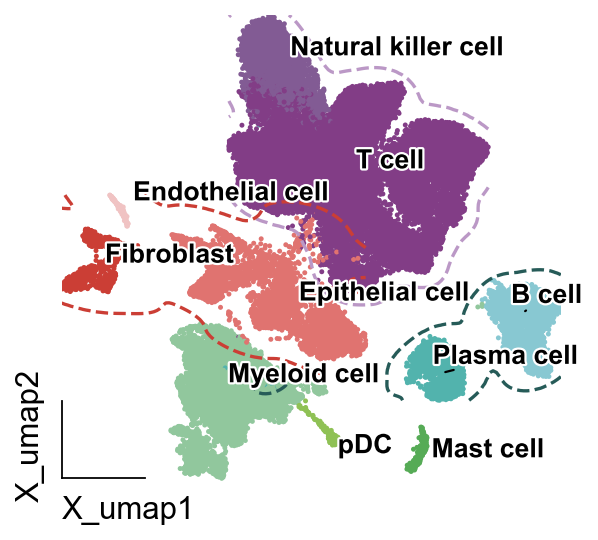
同类细胞轮廓
我们在很多顶刊中会发现,同类细胞往往作者喜欢用轮廓线圈起来,因此,我们在omicverse中也提供了类似的实现。
from matplotlib import patheffects
fig, ax = plt.subplots(figsize=(4,4))
ov.pl.embedding(adata,
basis="X_umap",
color=['major_celltype'],title='',
show=False, legend_loc=None, add_outline=False,
frameon='small',legend_fontoutline=2,ax=ax
)
ov.pl.embedding_adjust(
adata,
basis="X_umap",
groupby='major_celltype',
ax=ax,
adjust_kwargs=dict(arrowprops=dict(arrowstyle='-', color='black')),
text_kwargs=dict(fontsize=12 ,weight='bold',
path_effects=[patheffects.withStroke(linewidth=2, foreground='w')] ),
)
ov.pl.contour(ax=ax,adata=adata,
basis="X_umap",
groupby='major_celltype',clusters=['T cell','Natural killer cell'],
contour_threshold=0.02,colors=ov.pl.red_color[2],linestyles='dashed')
ov.pl.contour(ax=ax,adata=adata,
basis="X_umap",
groupby='major_celltype',clusters=['Plasma cell','B cell'],
contour_threshold=0.01,colors=ov.pl.blue_color[6],linestyles='dashed')
ov.pl.contour(ax=ax,adata=adata,
basis="X_umap",
groupby='major_celltype',clusters=['Endothelial cell', 'Epithelial cell', 'Fibroblast'],
contour_threshold=0.002,colors=ov.pl.orange_color[2],linestyles='dashed')
fig.savefig('umap-ct_major.png',dpi=300,bbox_inches='tight')
fig.savefig('umap-ct_major.pdf',dpi=300,bbox_inches='tight')
以上就是本期教程的全部内容了,如果觉得OmicVerse对你的论文有帮助,不要忘记应用哦,更多单细胞的可视化方法与教程请参考: https://starlitnightly.github.io/omicverse/Tutorials-plotting/t_visualize_single/
本文来自博客园,作者:Starlitnightly,转载请注明原文链接:https://www.cnblogs.com/starlitnightly/p/18262565



