使用国产大模型完成单细胞自动注释
作者按
我们在Python的scverse生态中,重新实现了GPTCelltype的函数,并加入了更多大模型的扩展,同时我们并将其封装进OmicVerse框架中
全文字数|预计阅读时间: 2000|5min
——Starlitnightly(星夜)
GPT-4 是一种专为语音理解和生成而设计的大型语言模型。
哥伦比亚大学梅尔曼公共卫生学院(Columbia University Mailman School of Public Health)的 Wenpin Hou 和杜克大学医学院(Duke University School of Medicine)的 Zhicheng Ji 证明,大语言模型 GPT-4 可以在单细胞 RNA 测序分析中使用标记基因信息准确注释细胞类型。
该研究以「Assessing GPT-4 for cell type annotation in single-cell RNA-seq analysis」为题,于 2024 年 3 月 25 日发布在《Nature Methods》。

在将 GPTCelltype 算法集成到 OmicVerse 中时,我们做了两项改进:
- 原生支持 Python: 由于 GPTCelltype 是一个 R 语言包,为了使其符合 scverse 的 anndata 生态系统,我们重写了整个函数,使其能够在 Python 下完美运行。
- 更多模型支持: 我们在 Openai 之外提供了更多大型模型供用户选择,例如 Qwen(通义千问)、Kimi,而且还通过参数 base_url 提供了更多模型支持。
import omicverse as ov
print(f'omicverse version:{ov.__version__}')
import scanpy as sc
print(f'scanpy version:{sc.__version__}')
ov.ov_plot_set()
____ _ _ __
/ __ \____ ___ (_)___| | / /__ _____________
/ / / / __ `__ \/ / ___/ | / / _ \/ ___/ ___/ _ \
/ /_/ / / / / / / / /__ | |/ / __/ / (__ ) __/
\____/_/ /_/ /_/_/\___/ |___/\___/_/ /____/\___/
Version: 1.6.0, Tutorials: https://omicverse.readthedocs.io/
omicverse version:1.6.0
scanpy version:1.7.2
加载数据
数据包括来自健康供体的 3k PBMCs,可从 10x Genomics 免费获取(这里 和这个 网页)。在 Unix 系统上,您可以取消注释并运行以下命令来下载和解压数据。最后一行会创建一个目录用于写入处理过的数据。
# !mkdir data
# !wget http://cf.10xgenomics.com/samples/cell-exp/1.1.0/pbmc3k/pbmc3k_filtered_gene_bc_matrices.tar.gz -O data/pbmc3k_filtered_gene_bc_matrices.tar.gz
# !cd data; tar -xzf pbmc3k_filtered_gene_bc_matrices.tar.gz
# !mkdir write
将计数矩阵读入 AnnData 对象,该对象包含许多用于注释和不同数据表示的插槽。它还具有自己的基于 HDF5 的文件格式:.h5ad。
adata = sc.read_10x_mtx(
'data/filtered_gene_bc_matrices/hg19/', # 含 `.mtx` 文件的目录
var_names='gene_symbols', # 使用基因符号作为变量名称(变量轴索引)
cache=True) # 写入缓存文件以加快后续读取速度
... 正在从缓存文件 cache/data-filtered_gene_bc_matrices-hg19-matrix.h5ad 读取
数据预处理
这里我们使用 ov.single.scanpy_lazy 预处理 scRNA-seq 原始数据,包括过滤双细胞、每细胞归一化、对数转换、提取高度可变基因、计算细胞聚类等。
但如果您想逐步体验预处理过程,我们也提供了更详细的预处理步骤,请参考我们的 预处理章节 获取详细说明。
我们将原始计数存储在 count 层中,并将原始数据存储在 adata.raw.to_adata() 中。
#adata=ov.single.scanpy_lazy(adata)
#质量控制
adata=ov.pp.qc(adata,
tresh={'mito_perc': 0.05, 'nUMIs': 500, 'detected_genes': 250})
#归一化和高可变基因(HVGs)计算
adata=ov.pp.preprocess(adata,mode='shiftlog|pearson',n_HVGs=2000,)
#保存所有基因并过滤非HVGs
adata.raw = adata
adata = adata[:, adata.var.highly_variable_features]
#对 adata.X 进行缩放
ov.pp.scale(adata)
#降维
ov.pp.pca(adata,layer='scaled',n_pcs=50)
#邻域图构建
sc.pp.neighbors(adata, n_neighbors=15, n_pcs=50,
use_rep='scaled|original|X_pca')
#聚类
sc.tl.leiden(adata)
#寻找标志物
sc.tl.dendrogram(adata,'leiden',use_rep='scaled|original|X_pca')
sc.tl.rank_genes_groups(adata, 'leiden', use_rep='scaled|original|X_pca',
method='wilcoxon',use_raw=False,)
#用于可视化的降维(X_mde=X_umap+GPU)
adata.obsm["X_mde"] = ov.utils.mde(adata.obsm["scaled|original|X_pca"])
adata
ov.pl.embedding(adata,
basis='X_mde',
color=['leiden'],
legend_loc='on data',
frameon='small',
legend_fontoutline=2,
palette=ov.utils.palette()[14:],
)
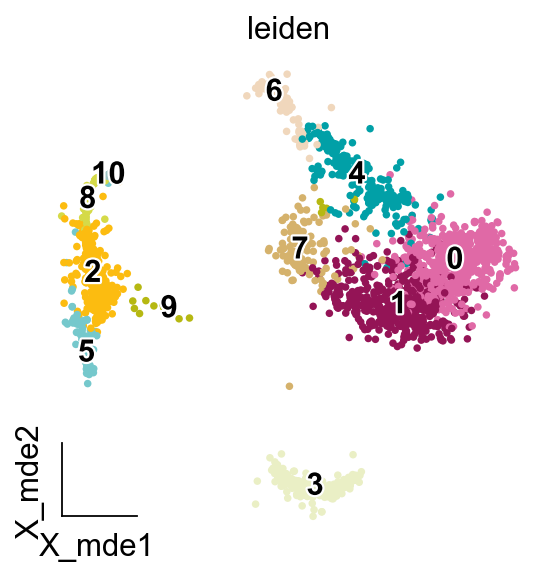
GPT Celltype
gptcelltype 支持字典格式输入,我们提供 omicverse.single.get_celltype_marker 来获取每种细胞类型的标志基因作为字典。
使用手动基因
我们可以手动定义一个字典来确定输出的准确性
import os
all_markers={'cluster1':['CD3D','CD3E'],
'cluster2':['MS4A1']}
os.environ['AGI_API_KEY'] = 'sk-**' # 替换为您的实际 API 密钥
result = ov.single.gptcelltype(all_markers, tissuename='PBMC', speciename='human',
model='qwen-plus', provider='qwen',
topgenenumber=5)
result
注意: 找到 AGI API 密钥: 返回细胞类型注释。
注意: 在进行下游分析之前,建议检查 GPT-4 返回的结果以防 AI 幻觉。
{'cluster1': '1. T cells (CD3D, CD3E)', 'cluster2': '2. B cells (MS4A1)'}
自动获取聚类基因
all_markers=ov.single.get_celltype_marker(adata,clustertype='leiden',rank=True,
key='rank_genes_groups',
foldchange=2,topgenenumber=5)
all_markers
...获取细胞类型标志物
{'0': ['LTB', 'LDHB', 'IL32', 'CD3D', 'IL7R'],
'1': ['LDHB', 'CD3D', 'NOSIP', 'CD3E', 'C6orf48'],
'10': ['SPARC', 'PPBP', 'GNG11', 'PF4', 'CD9'],
'2': ['LYZ', 'S100A9', 'S100A8', 'FCN1', 'TYROBP'],
'3': ['CD74', 'CD79A', 'HLA-DRA', 'CD79B', 'HLA-DPB1'],
'4': ['CCL5', 'NKG7', 'CST7', 'GZMA', 'IL32'],
'5': ['LST1', 'FCER1G', 'COTL1', 'AIF1', 'IFITM3'],
'6': ['NKG7', 'GZMB', 'GNLY', 'CTSW', 'PRF1'],
'7': ['CCL5', 'GZMK'],
'8': ['FTL', 'FTH1', 'S100A8', 'S100A9', 'TYROBP'],
'9': ['HLA-DRB1', 'HLA-DPA1', 'HLA-DPB1', 'HLA-DRA', 'HLA-DRB5']}
import os
os.environ['
AGI_API_KEY'] = 'sk-**' # 替换为您的实际 API 密钥
result = ov.single.gptcelltype(all_markers, tissuename='PBMC', speciename='human',
model='qwen-plus', provider='qwen',
topgenenumber=5)
result
注意: 找到 AGI API 密钥: 返回细胞类型注释。
注意: 在进行下游分析之前,建议检查 GPT-4 返回的结果以防 AI 幻觉。
{'0': '1. T cells (LTB, IL7R)',
'1': '1. T cells (CD3D, CD3E)',
'10': '2. Megakaryocytes (PPBP, PF4)',
'2': '3. Monocytes (LYZ, S100A9)',
'3': '4. B cells (CD74, CD79A)',
'4': '1. T cells (CCL5, GZMA)',
'5': '3. Monocytes (FCER1G, AIF1)',
'6': '5. Natural Killer (NK) cells (NKG7, GZMB)',
'7': '5. Natural Killer (NK) cells (CCL5, GZMK)',
'8': '3. Monocytes (S100A8, S100A9)',
'9': '4. B cells (HLA-DRB1, HLA-DRA)'}
将 GPT Celltype 结果映射到 Scanpy
我们可以仅保留输出的细胞类型,并删除其他无关信息。
new_result = {}
for key in result.keys():
new_result[key] = result[key].split(': ')[-1].split(' (')[0].split('. ')[1]
new_result
{'0': 'T cells',
'1': 'T cells',
'10': 'Platelets or Megakaryocytes',
'2': 'Neutrophils',
'3': 'B cells',
'4': 'Cytotoxic T cells or NK cells',
'5': 'Monocytes or Macrophages',
'6': 'Cytotoxic T cells or NK cells',
'7': 'Cytotoxic T cells',
'8': 'Neutrophils',
'9': 'Antigen presentation'}
adata.obs['gpt_celltype'] = adata.obs['leiden'].map(new_result).astype('category')
ov.pl.embedding(adata,
basis='X_mde',
color=['leiden', 'gpt_celltype'],
legend_loc='on data',
frameon='small',
legend_fontoutline=2,
palette=ov.utils.palette()[14:],
)
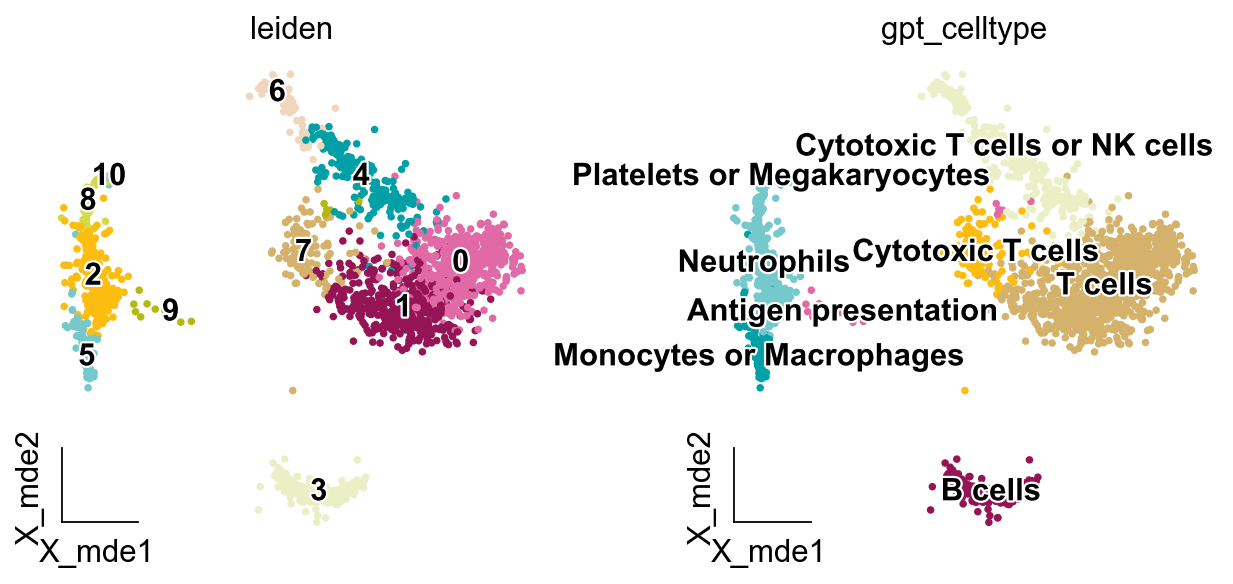
更多模型
在 omicverse 中实现的 gptcelltype 支持几乎所有支持 openai API 格式的大模型。
all_markers = {'cluster1': ['CD3D', 'CD3E'],
'cluster2': ['MS4A1']}
OpenAI
OpenAI API 使用 API 密钥进行身份验证。您可以在用户或服务账户级别创建 API 密钥。服务账户绑定到一个“机器人”个体,应该用于为生产系统提供访问权限。每个 API 密钥可以被范围化到以下之一:
-
用户密钥 - 我们的传统密钥。提供对用户已添加的所有组织和项目的访问权限;访问 API 密钥以查看您的可用密钥。我们强烈建议转换到项目密钥以获得最佳的安全实践,尽管目前仍支持通过此方法进行访问。
-
请选择您需要使用的模型:支持的模型列表。
os.environ['AGI_API_KEY'] = 'sk-**' # 替换为您的实际 API 密钥
result = ov.single.gptcelltype(all_markers, tissuename='PBMC', speciename='human',
model='gpt-4o', provider='openai',
topgenenumber=5)
result
注意: 找到 AGI API 密钥: 返回细胞类型注释。
注意: 在进行下游分析之前,建议检查 GPT-4 返回的结果以防 AI 幻觉。
{'cluster1': '1. T cell', 'cluster2': '2. B cell'}
Qwen (通义千问)
-
已开通灵积模型服务并获得 API-KEY:开通 DashScope 并创建 API-KEY。
-
我们推荐您将 API-KEY 配置到环境变量中以降低 API-KEY 的泄漏风险,配置方法可参考通过环境变量配置 API-KEY。您也可以在代码中配置 API-KEY,但泄漏风险会提高。
-
请选择您需要使用的模型:支持的模型列表。
os.environ['AGI_API_KEY'] = 'sk-**' # 替换为您的实际 API 密钥
result = ov.single.gptcelltype(all_markers, tissuename='PBMC', speciename='human',
model='qwen-plus', provider='qwen',
topgenenumber=5)
result
注意: 找到 AGI API 密钥: 返回细胞类型注释。
注意: 在进行下游分析之前,建议检查 GPT-4 返回的结果以防 AI 幻觉。
{'cluster1': 'CD3D, CD3E: T cells', 'cluster2': 'MS4A1: B cells'}
Kimi (月之暗面)
os.environ['AGI_API_KEY'] = 'sk-**' # 替换为您的实际 API 密钥
result = ov.single.gptcelltype(all_markers, tissuename='PBMC', speciename='human',
model='moonshot-v1-8k', provider='kimi',
topgenenumber=5)
result
注意: 找到 AGI API 密钥: 返回细胞类型注释。
注意: 在进行下游分析之前,建议检查 GPT-4 返回的结果以防 AI 幻觉。
{'cluster1': '1. T cell', 'cluster2': '2. B cell'}
其他模型
您可以手动设置 base_url 参数来指定需要使用的其他模型,注意模型需要支持 Openai 的参数。这里提供了三个例子(当您指定 base_url 参数时,provider 参数将无效):
if provider == 'openai':
base_url = "https://api.openai.com/v1/"
elif provider == 'kimi':
base_url = "https://api.moonshot.cn/v1"
elif provider == 'qwen':
base_url = "https://dashscope.aliyuncs.com/compatible-mode/v1"
os.environ['AGI_API_KEY'] = 'sk-**' # 替换为您的实际 API 密钥
result = ov.single.gptcelltype(all_markers, tissuename='PBMC', speciename='human',
model='moonshot-v1-8k', base_url="https://api.moonshot.cn/v1",
topgenenumber=5)
result
注意: 找到 AGI API 密钥: 返回细胞类型注释。
注意: 在进行下游分析之前,建议检查 GPT-4 返回的结果以防 AI 幻觉。
{'cluster1': '1. T cell', 'cluster2': '2. B cell'}
本文来自博客园,作者:Starlitnightly,转载请注明原文链接:https://www.cnblogs.com/starlitnightly/p/18260551



