遗传算法(白话文版)
遗传算法是一种全局最优化算法,其很好地借鉴了自然界中生物进化的规律,通过模拟这种优胜劣汰的规律以寻找极值点。本文将从其原理,实现过程以及可行性的角度通俗地解剖这种算法!
一、算法基本思想
遗传算法是进化算法中的一种,初见其字面其实就可以大致推知出其的思想了。遗传算法就是建立在达尔文的生物进化论基础上的,通过模拟自然界不断筛选适应度比较好的个体,并通过使它们发生交叉(生物学中的专用名词,简单地理解就是生儿育女),变异,产生的新的个体会组成新的种群,继续进行新的一轮进化,最终留下的个体都有很高的适应度,这些个体中往往包含我们需要寻找的极值点。
太抽象不理解?没关系,笔者还为你准备了精美的演示图片:
- 很久之前,有一群快乐小猪生活在山区里,它们生活的位置都是不同的。

- 然而,有一天突然从山脚下升起了一股毒烟,并慢慢向山上蔓延,此时,一些生活在低海拔的小猪就被毒死了,而生活在较高海拔的小猪侥幸活了下来,但它们知道毒烟不断向上蔓延,所以它们会向高处迁居。

- 迁居的过程是漫长的,在这个过程中,有些小猪会相爱?,并生下后代,然后,它们自身就会死去,它们的子孙从它们父母已达到的高度继续向上迁居。这样,一代又一代,有一天,这些小猪发现自己达到了最高点附近,此时,毒烟渐渐散去,小猪们在最高点继续快乐地生活下去。

以上就是遗传算法的基本思想了。
二、实现过程
接下来,我们根据上面的图片讲解遗传算法怎么实现。
首先,我们注意,小猪的初始生活点是随机的,也就是,有的小猪生活的海拔高,有的就比较低,对应到算法中就是,算法开始,我们会随机生成一组点(当然这些点需要在可行域内)。
然后,毒气的出现,使得一些海拔低的小猪被毒死了,而海拔高的小猪活了下来,对应到算法就是选择(Selection)的过程(有?轮盘赌算法?和锦标赛算法),在这个过程中,我们会把适应度高的点保留下来,而遗弃适应度低的点(所谓适应度可以通俗地理解为这个点对应的函数值的大小)。
接着,活下来的小猪会生育后代,其后代会在父母已达到的高度继续向上迁居,对应算法的过程是交叉(Crossover)过程,这个过程中,我们随机抽取几对点,每对点会进行类似“中和”的操作,适应度高的点和适应度低的点“中和”生成平均适应度更高的点。这个地方我们通过一个具体的例子来进行理解:我们假设抽取的一个点的坐标为(1,1),其适应度为8,而另一个点的坐标为(2,2),适应度为2,此时,这对点的平均适应度为5,接着,我们随机生成一个随机数a(0~1之间),然后,a(1,1)+(1-a)(2,2)和(1-a)(1,1)+a(2,2)这两个点(也就是(1,1)和(2,2)连线上的两点)就是之前两个点“中和”的结果,我们会发现这两个新生成的点平均适应度往往更好,最后我们用新生成的点来取代原先的两个点。当然,交叉的操作的实现方式有很多种,上面的只是其中一种实现方式。
然而,我们知道,生物在进化过程中可能会发生变异(上面的漫画中未体现),变异后的个体适应度可能更好,当然也可能更差,所以我们会回到选择的过程,将适应度好的保留下来,将差的剔除掉,接着继续交叉,变异,这样就开始了新的一轮循环。当迭代到一定次数时,这些点渐渐都来到了最高点附近。在上面的迭代过程中,我们会记录下适应度最好的那个体,如果,新的迭代中产生更好的个体,则将该个体记录为最好的,算法结束后,我们返回这个适应度最好的个体即可。
三、可行性评估
遗传算法既然流传了几十年,解决了很多问题,从实践角度看,其肯定是可行的,在接下来的程序运行中我们也会看到。我们还需要从理论角度评估其可行性。
首先,选择过程中,我们默认采用了?轮盘赌算法?,这个算法可以使得适应度高的个体有更大的概率保留下来,而适应度低的个体保留下来的概率则小,当然,也有可能,适应度低的个体保留下了而抛弃了适应度高的,但从概率角度看,随着测试次数的增加,往往适应度高的个体能被保留下来。
我们再来看交叉的过程,这个过程中,当有一对个体发生交叉时,则产生的后代的适应度是居于其父母的适应度的之间的,这样看起来把适应度高的个体“拖累”了,但这样的结果是产生的后代的平均适应度往往增大了,也就是种群的平均水平提高了。
上面讲到交叉操作使得种群的平均适应度提高了而“拖累”了适应度高的点,这样不就实现不了找最高的目标了呀。所以,我们还需要变异操作,使其中一些点发生“变异”,变异的点可能适应度更高,在下一轮的选择过程中,这些适应度高的点更有可能保留下来,组成新的种群,整个过程下来,整个种群的平均适应度基本上一直在提高(除非都到了最高点附近),而变异这种碰运气的操作使得我们可能瞎猫拖到一个最高点。
可以说,整个遗传算法的各个阶段都是碰运气的过程,但碰到适应度高的点的可能性大,所以,在多次迭代和多次测试中,这些点往往能收敛于最高点。
四、具体代码
激动人心的时候来了,有了上述基础后,我们就可以阅读具体代码了(这段代码笔者在anaconda3中多次测试通过,但并不保证代码完全正确,如果你发现问题的话,可以在评论中留言,谢谢)。
# -*- coding: utf-8 -*-
"""
If you find bugs,please contact us :sj2050@vip.163.com or
zzzain46@gmail.com or
ruanyipu@gmail.com
Thank you!
@author: SJ2050
"""
import numpy as np
import matplotlib.pyplot as plt
# Solve Chinese garbled problem
plt.rcParams['font.sans-serif']=['SimHei']
plt.rcParams['axes.unicode_minus']=False
def ga_real(population_size,fit_func,feasible_range,**options):
"""
input:
popolation_size = population_size (int)
fit_func = name of fitness value function (function)
feasible_range = feasible restrain region (two dimensional array)
options : is_print(plot,default = True) ,
max_iter(iteration number,default = 3*population_size),
p_c(crossover possibility, default = 0.75),
p_m(mutation_possibility, default = p_c/20)
return:
best_value_so_far,
best_chromosome_so_far,
search_points,
upper,
lower,
average
"""
# initialize input
is_print = options.get('is_print')
if (is_print==None or is_print!=False):
is_print = True
# selection = options.get('selection')
# if (selection==None or type(selection)!=int):
# selection = 1
max_iter = options.get('max_iter')
if(max_iter==None or type(max_iter)!= int or max_iter <= 0):
max_iter = 3*population_size
p_c = options.get('p_c')
if(p_c==None or type(p_c) not in [int,float] or p_c <= 0 or p_c > 1 ):
p_c = 0.75
p_m = options.get('p_m')
if (p_m==None or type(p_m) not in [int, float] or p_m<=0 or p_m > 1):
p_m = p_c/20
lower_boundary = np.array([feasible_range[i][0] for i in range(len(feasible_range))])
upper_boundary = np.array([feasible_range[i][1] for i in range(len(feasible_range))])
upper = np.array(np.zeros(max_iter+1))
average = np.array(np.zeros(max_iter+1))
lower = np.array(np.zeros(max_iter+1))
search_points = np.array([[]])
P = np.array(np.zeros((population_size,len(feasible_range))))
fitness = np.array(np.zeros(population_size))
# produce groups of chromosomes randomly
for i in range(population_size):
P[i,:] = lower_boundary + np.array([np.random.rand()*(upper_boundary[0] - lower_boundary[0]),\
np.random.rand()*(upper_boundary[1] - lower_boundary[1])])
best_value_so_far = 0
for i in range(population_size):
fitness[i] = fit_func(P[i,:])
max_value_index = np.argmax(fitness)
max_value = fitness[max_value_index]
best_value_so_far = max_value
best_chromosome_so_far = P[max_value_index,:]
upper[0] = max_value
average[0] = np.average(fitness)
lower[0] = np.min(fitness)
search_points = np.append(search_points,[best_chromosome_so_far],axis = 1)
# start iteration
for i in range(max_iter):
# when all chromosomes become same
if(not check_same(fitness)):
# Selection
mating_pool = roulette_wheel(P[:],fitness[:]) # deliver a copy of fitness
# Crossover
new_mating_pool = mating_pool.copy()
for j in range(population_size//2):
ind1 = int(np.floor(np.random.rand()*population_size))
ind2 = int(np.floor(np.random.rand()*population_size))
while(ind1 == ind2):
ind2 = int(np.floor(np.random.rand()*population_size))
parent1 = mating_pool[ind1,:]
parent2 = mating_pool[ind2,:]
if (np.random.rand()<p_c):
alpha = np.random.rand()
offspring1 = alpha*parent1 + (1-alpha)*parent2
offspring2 = alpha*parent2 + (1-alpha)*parent1
new_mating_pool[ind1,:] = offspring1
new_mating_pool[ind2,:] = offspring2
# Mutation
for j in range(population_size):
if (np.random.rand() < p_m):
alpha = np.random.rand()
new_mating_pool[j,:] = alpha*new_mating_pool[j,:] + \
(1-alpha)*(lower_boundary + np.array([np.random.rand()*(upper_boundary[0] - lower_boundary[0]),\
np.random.rand()*(upper_boundary[1] - lower_boundary[1])]))
P = new_mating_pool
# Evaluation
for j in range(population_size):
fitness[j] = fit_func(P[j,:])
max_value_index = np.argmax(fitness)
max_value = fitness[max_value_index]
if (max_value > best_value_so_far):
best_value_so_far = max_value
best_chromosome_so_far = P[max_value_index,:]
upper[i+1] = max_value
average[i+1] = np.average(fitness)
lower[i+1] = np.min(fitness)
search_points = np.concatenate((search_points,[P[max_value_index,:]]))
# Plotting
if is_print:
plt.plot(range(0,max_iter+1),upper,'o:',range(0,max_iter+1),average,'x-',range(0,max_iter+1),lower,'*--')
plt.title('遗传算法(real_num)迭代变化图')
plt.legend(('最好','平均','最差'))
plt.xlabel('迭代次数')
plt.ylabel('适应度')
plt.show()
return best_value_so_far,best_chromosome_so_far,search_points,upper,lower,average
# one way to achieve crossover process
def roulette_wheel(chromosomes,fitness):
fitness = fitness - min(fitness) # to keep fitness positive
each_fitness_proportion = fitness/np.sum(fitness)
cumulation_proportion = 0
mating_pool = np.array(np.zeros(chromosomes.shape))
for i in range(len(fitness)):
random_num = np.random.rand()
for j in range(len(fitness)):
if (cumulation_proportion <= random_num and random_num < cumulation_proportion + each_fitness_proportion[j]):
mating_pool[i,:] = chromosomes[j,:]
break
cumulation_proportion += each_fitness_proportion[j]
cumulation_proportion = 0
return mating_pool
# check whether all the chromosomes become same
def check_same(fitness):
fitness = fitness - min(fitness) # to keep fitness positive
# to check whether all the chromosomes are same
for i in range(len(fitness)):
if (fitness[i]!=0):
return False
return True
if __name__ == '__main__':
def fit_func(chromosome):
x,y = chromosome[0],chromosome[1]
return 3*(1-x)**2*np.e**(-x**2-(y+1)**2) - 10*(x/5-x**3-y**5)*np.e**(-x**2-y**2) - (np.e**(-(x+1)**2-y**2))/3
best_value,best_index,search_points,upper,lower,average = ga_real(20,fit_func,[[-3,3],[-3,3]],is_print = True)
print(best_value)
print(best_index)
这个代码是实数版的遗传代码实现(我们这里也附上二进制版的遗传代码),选择过程选择了?轮盘赌算法?,交叉过程之前已提到过。我在这个代码的必要部分已经做好了注释,如果读者按照之前讲的那些思想来阅读代码的相应片段的,我相信,这些并不能难倒聪明的你。
五、运行结果
在笔者多次调试和修改后,这个代码的稳定性很好,收敛速度也很快,基本可以找到可行域中的最高点,偏差2%左右,如果多运行几次,所得结果基本可和真值媲美。
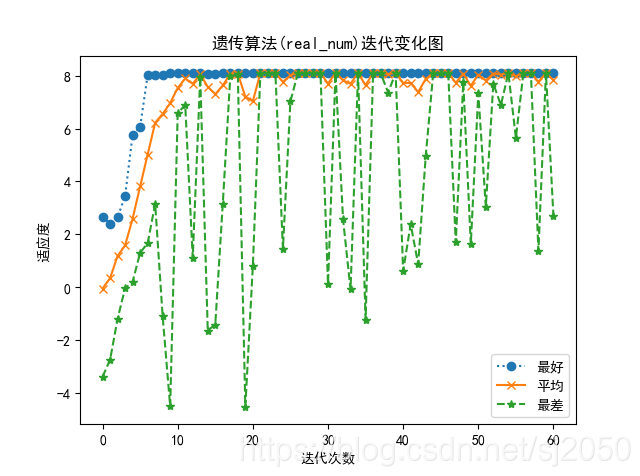
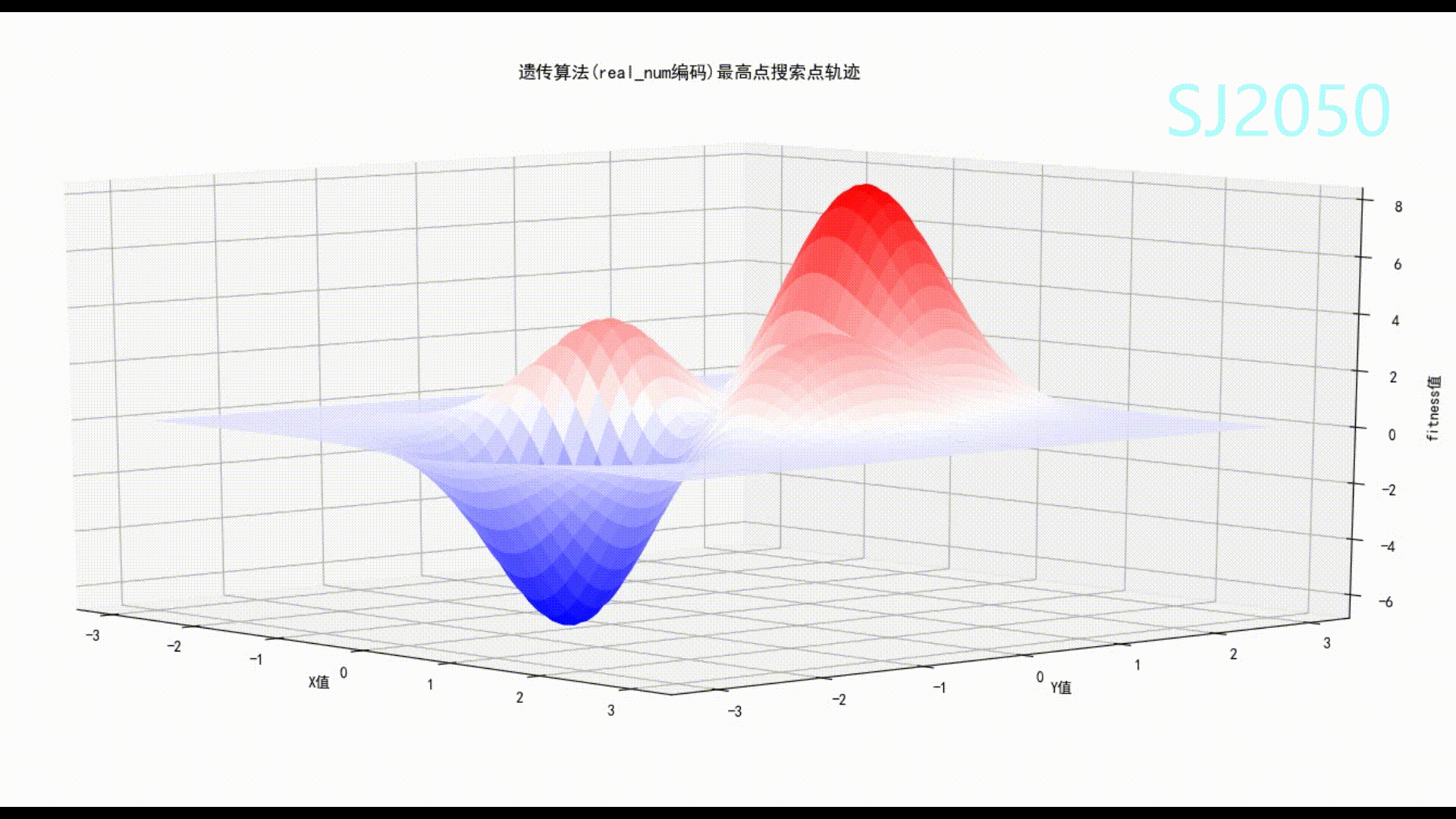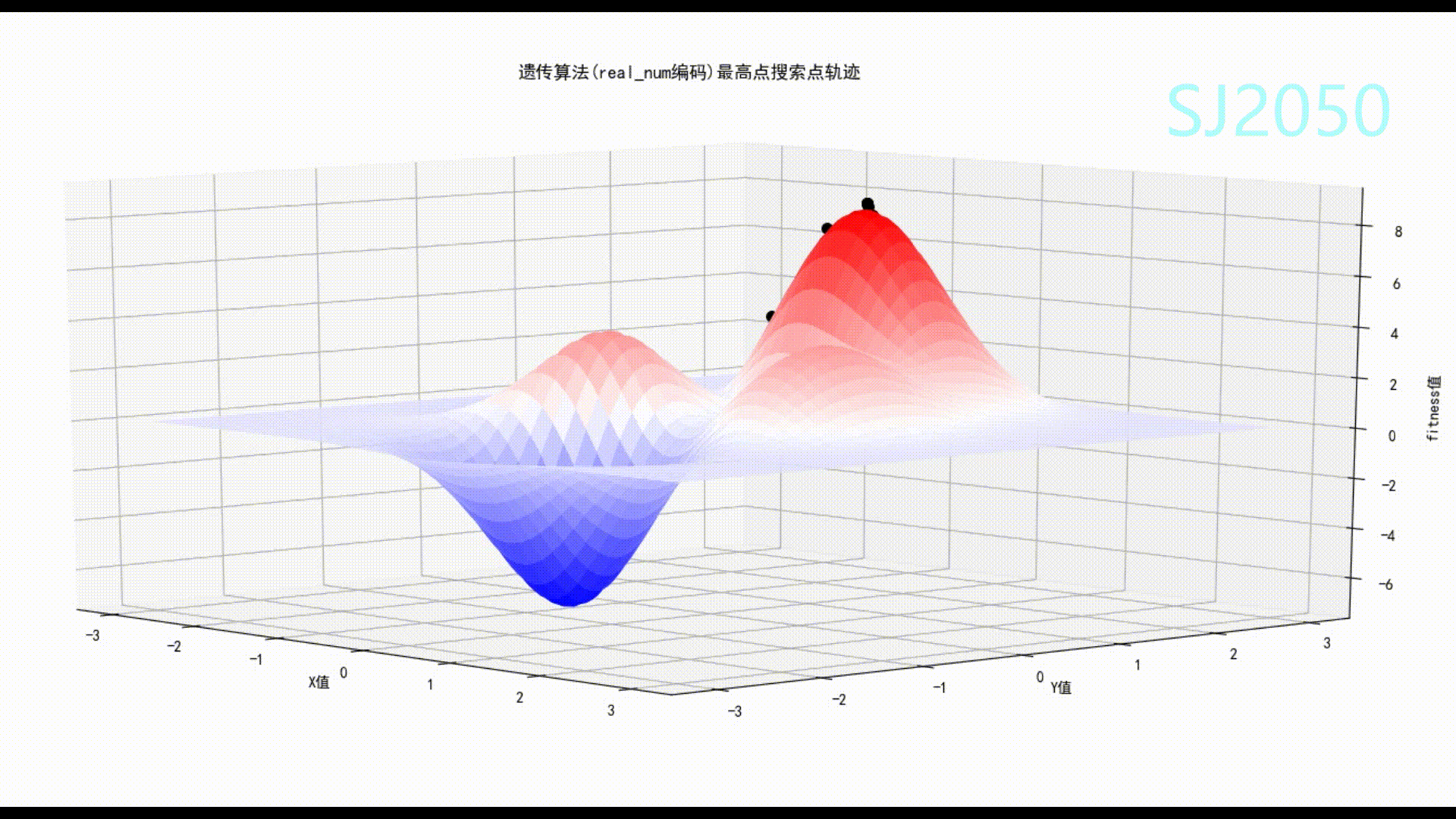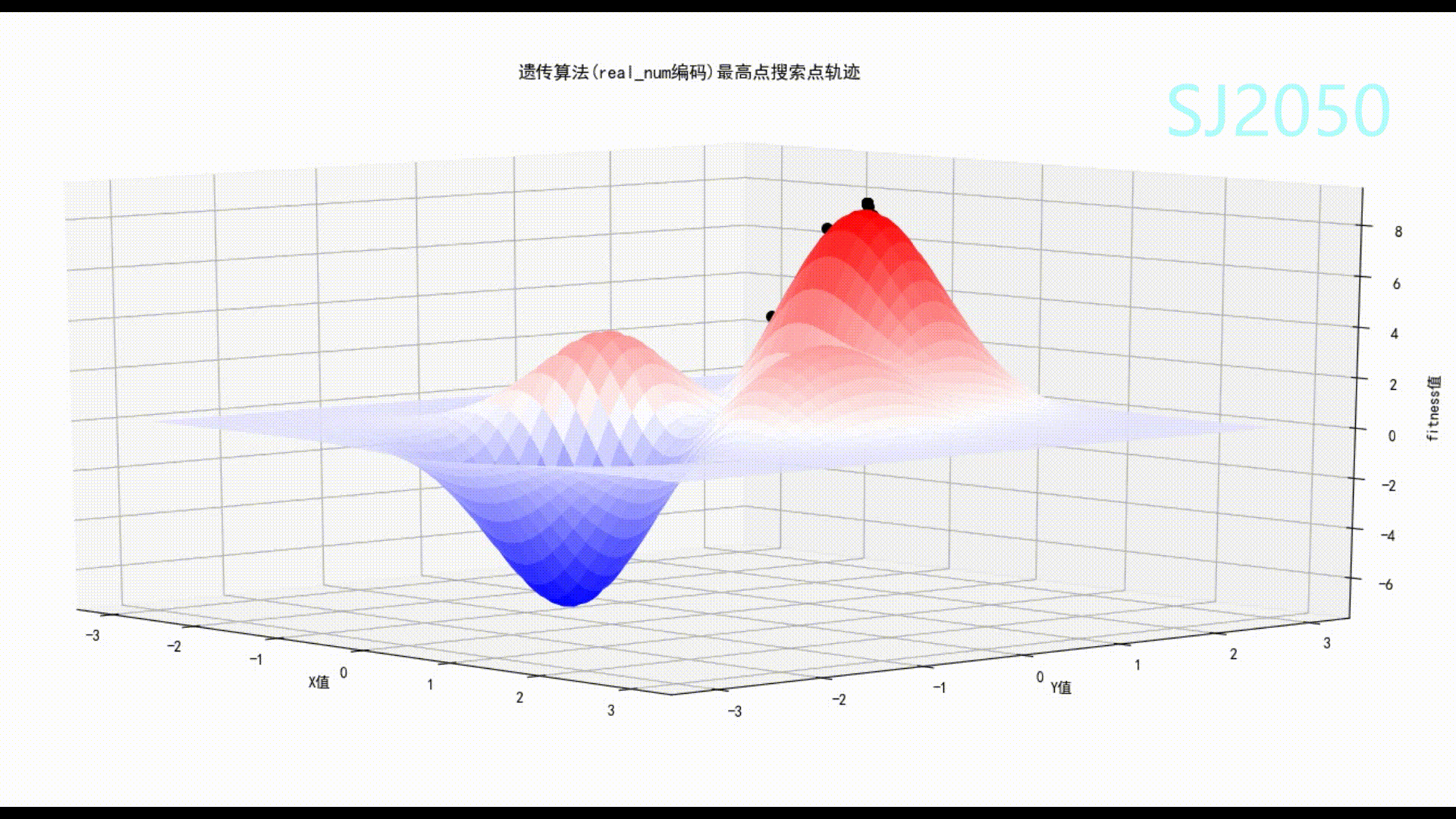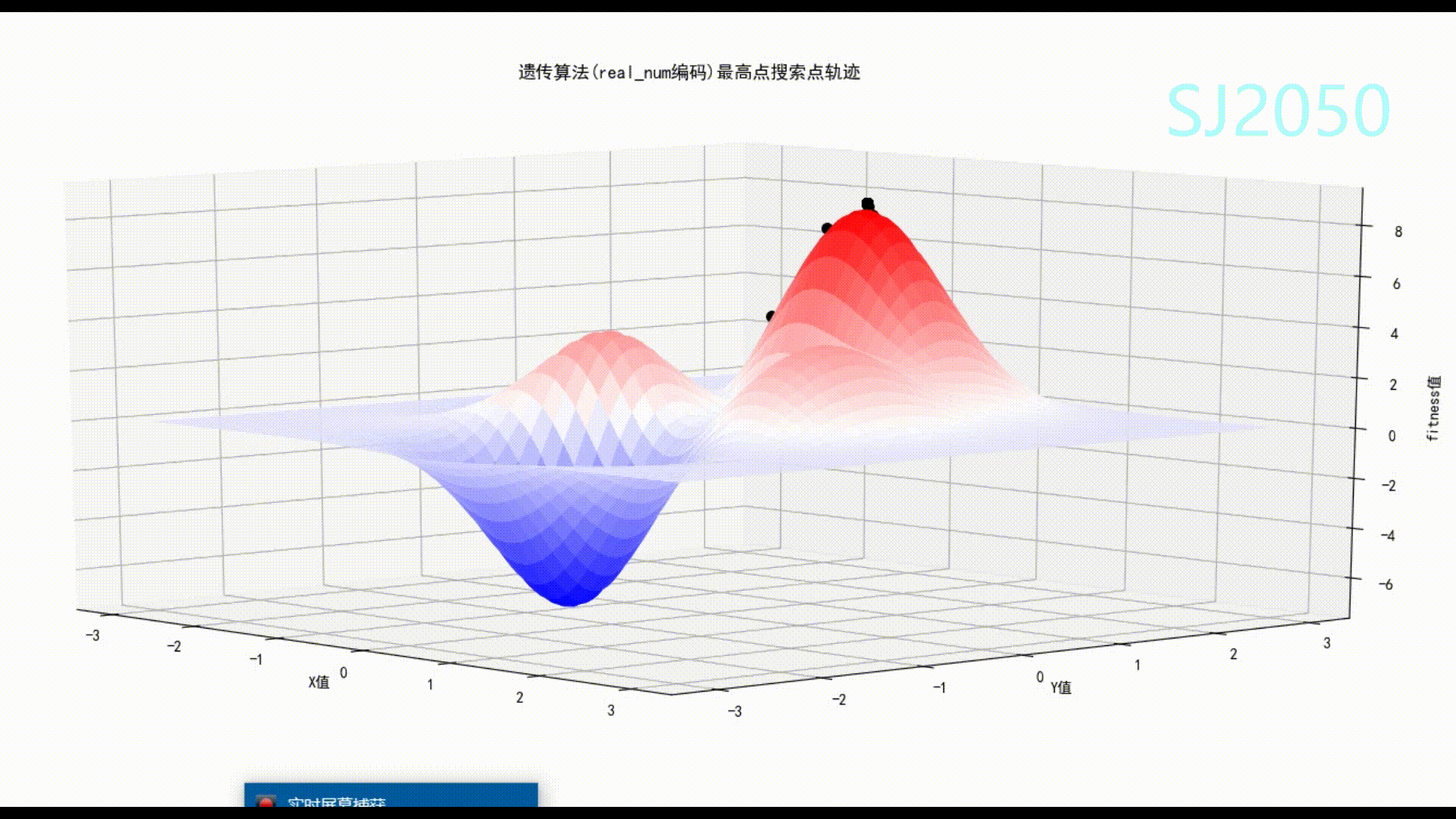
通过这个算法,我们也可以体会到大自然的美,顺自然者兴,逆自然者亡。
最后,感谢阅读!如果你觉得可以的话,也可以为我点赞?哦!
- 参考资料
1、《An introduction to optimization 4th edition》
2、某高校最优化课件
3、轮盘赌算法


