R 数据可视化 : 热图
本文作者蒋刘一琦,自嘲是一个有艺术追求的生信狗,毕业于浙江大学生物信息学专业,目前在复旦大学就读研究生,研究方向为宏基因组。
在生物信息领域我们常常使用 R 语言对数据可视化。在对数据可视化的时候,我们需要明确想要展示的信息,从而选择最为合适的图突出该信息。本系列文章将介绍多种基于不同 R 包的作图方法,希望能够帮助到各位读者。
什么是热图(Heatmap)
热图是一个以颜色变化来显示数据的矩阵。Toussaint Loua 在1873年就曾使用过热图来绘制对巴黎各区的社会学统计。

生物学中热图经常用于展示多个基因在不同样本中的表达水平。然后可以通过聚类等方式查看不同组(如疾病组和对照组)特有的 pattern。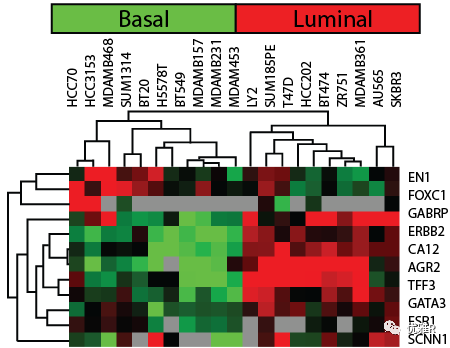 如上图每一列代表一个样本(左侧的样本是 Basal,右侧的样本是 Luminal),每一行代表一个基因,颜色代表了表达量(这张图没有显示图例,不知道是偏绿还是偏红代表高表达量)。可以看到这些挑选出的基因在两组的表达有较大的差异,EN1、FOXC1 这几个基因在 Basal 组总体呈现红色,在 Luminal 呈现绿色。而后面的几个基因在 Luminal 总体呈现红色,在Basal总体呈现绿色。
如上图每一列代表一个样本(左侧的样本是 Basal,右侧的样本是 Luminal),每一行代表一个基因,颜色代表了表达量(这张图没有显示图例,不知道是偏绿还是偏红代表高表达量)。可以看到这些挑选出的基因在两组的表达有较大的差异,EN1、FOXC1 这几个基因在 Basal 组总体呈现红色,在 Luminal 呈现绿色。而后面的几个基因在 Luminal 总体呈现红色,在Basal总体呈现绿色。
外面的树状图形是对基因和样本的聚类,通常聚类的结果把基因的表达量相似的聚在一起,把基因表达的 pattern 相似的样本聚在一起。所以在该图中可以看到 Basal 的样本都聚在了一起,Luminal 的样本也都聚在了一起。通常如果两组的差异较明显,组内的 pattern 较为相似,就能有这样的聚类结果——一个组的样本聚类在一起。相反如果是差异较小的两组样本,就很可能混在一起。
热图还可以用于展示其他物质的丰度比如微生物的相对丰度、代谢组不同物质的含量等等。当然,另一个热图的重要用处就是展现不同指标、不同样本等之间的相关性。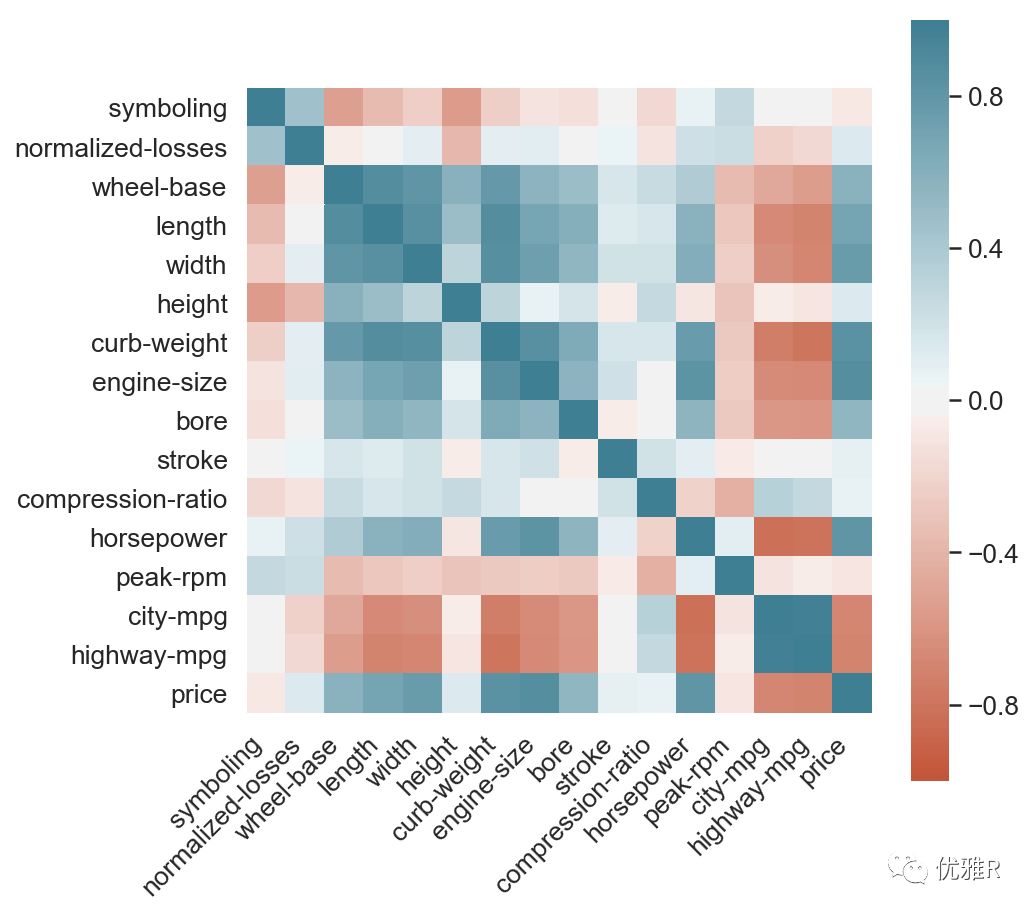 此时颜色代表的就是相关系数的大小。所以可以看到自己和自己的相关系数是1,也就是最深的蓝色。约接近白色说明相关性越弱,偏蓝(正相关)或者偏红(负相关)则代表相关性强。
此时颜色代表的就是相关系数的大小。所以可以看到自己和自己的相关系数是1,也就是最深的蓝色。约接近白色说明相关性越弱,偏蓝(正相关)或者偏红(负相关)则代表相关性强。
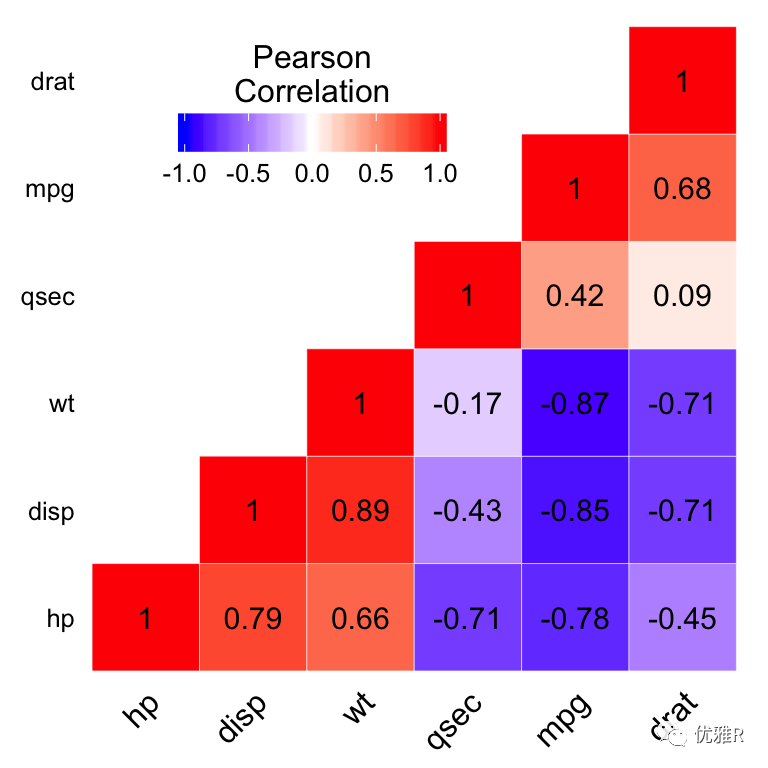
当然在相关性的计算中除了相关系数以外,我们还会看 pvalue 是否显著。如果我们想要把 pvalue 表示在图中,可以在格子上添加*号或者具体的数值。同时因为这里可以看到其实不同的两个指标之间的关系是被重复展现了2次,比如 symboling 与 normalized-losses(最上面一行的第二个格子,和从上往下的第二行的第一个格子),因此有时候我们只展现一半即对角线以上或以下的一半图形。
怎么做热图Heatmap
1)需要什么格式的数据
有很多的软件都可以做 heatmap。我们要介绍的当然是 R,R 默认中提供了 heatmap 函数。当然,R 中也有很多具有 heatmap 功能的包,比如 ggplot2, gplots。今天我们介绍含有 heatmap.2 功能的 gplots 包。heatmap.2 函数和我们之前要求的数据类型不太一样,这个函数输入数据要求是个矩阵(matrix)。
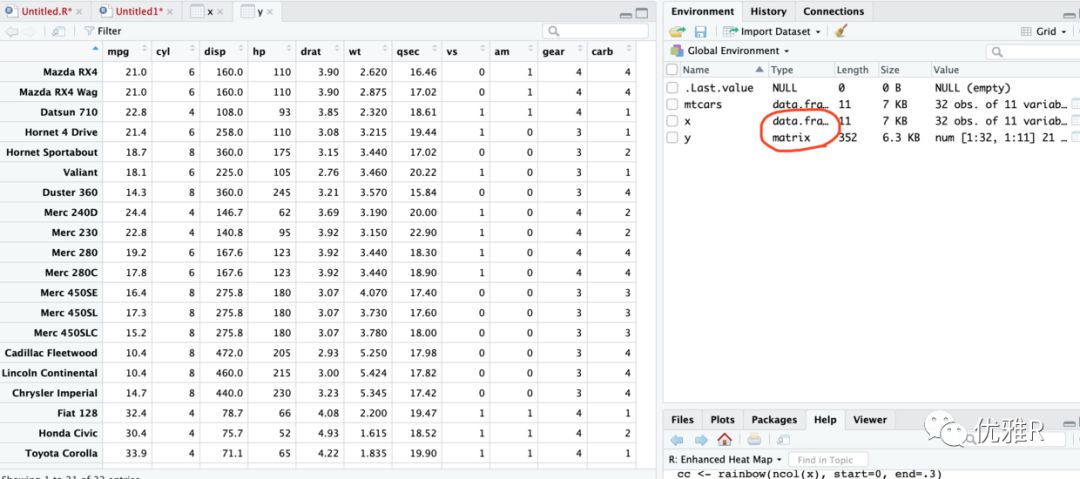
data(mtcars)x<-mtcarsy<-as.matrix(mtcars)
在 R Studio 中我们可以清楚的看到 x 和 y 的区别(虽然如果点开你也许会觉得 x 与 y 难道不是一模一样吗),x 的 type 是 dataframe 的格式,而 y 是 matrix 也就是矩阵格式。这两种数据类型有什么差别呢?matrix 中的值只能是一个格式,比如都是字符型。而 dataframe 可以同时支持不同的类型比如数值型和字符型。
2)如何做图
本节用一个不是那么生物的数据集来展示一下如何做热图。
data("attitude")Ca <- cor(attitude)
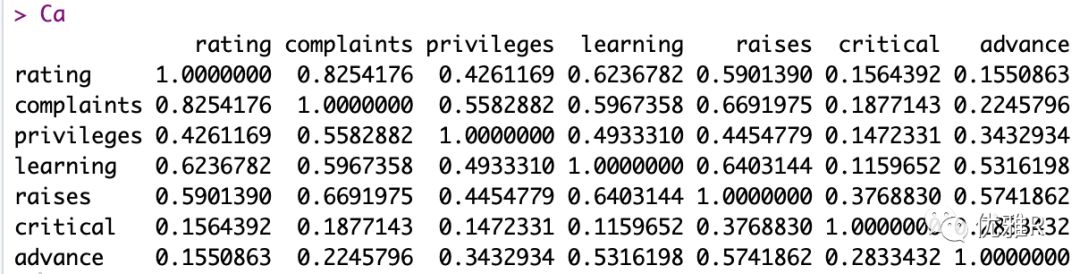
如果直接使用默认的 heatmap.2 功能我们可以看到: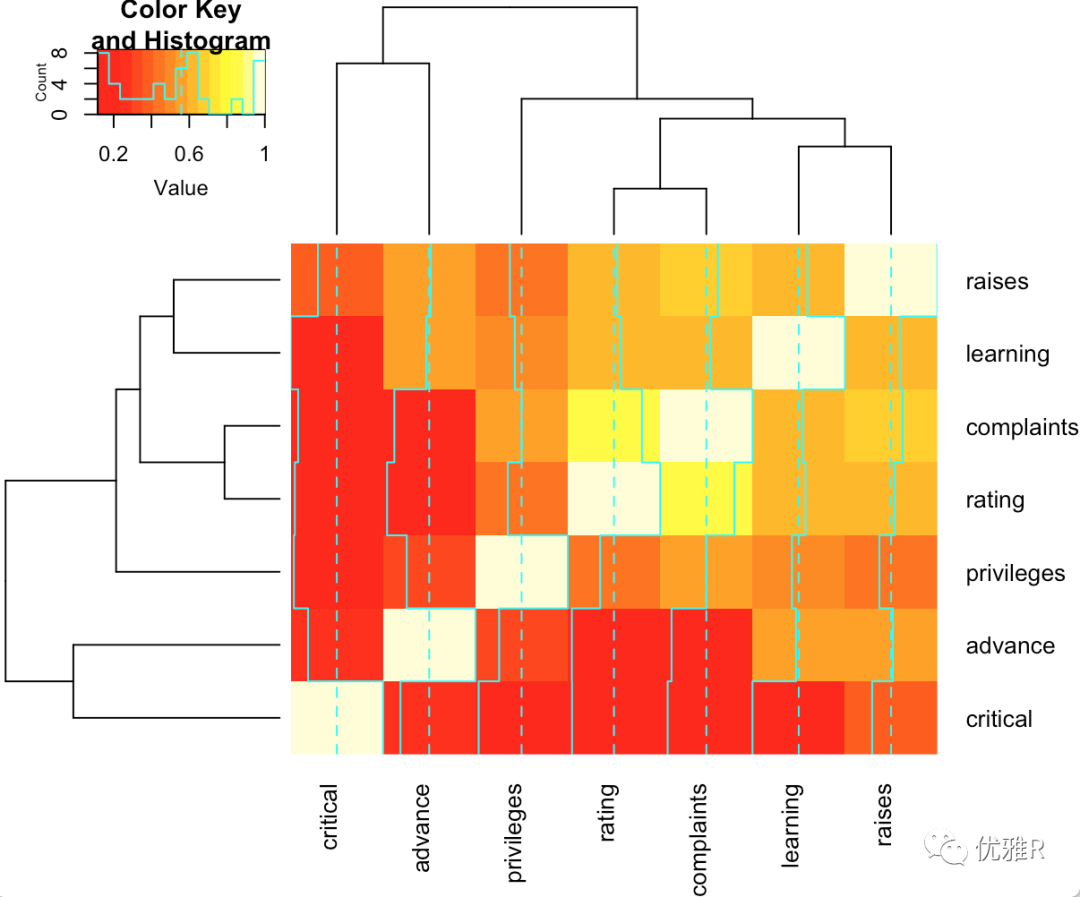
和平时看到的 heatmap 有些不一样,中间的这些蓝色的线我们称作 “trace”:虚线表示这一列平均值,实线表示与平均值的偏离程度。默认是按照列计算平均等,也可以改为行。但是我们这里的数据是做相关性,所以这些线的意义就不是那么大。图例中也类似,展示了不同颜色对应的值大小,而蓝色的实线是根据数据分布做的密度曲线,虚线是平均值。黑色的线之前我们已经提到过是对数据进行了聚类。
然后我们对图进行一些修改,红色太扎眼换个颜色,把一些不需要的功能去掉。比如聚类比如这些蓝色的线。
data("attitude")Ca <- cor(attitude) #cor 的结果就是矩阵library(gplots)library(RColorBrewer)coul <- colorRampPalette(brewer.pal(8, "PiYG"))(25) #换个好看的颜色hM <- format(round(Ca, 2)) #对数据保留2位小数heatmap.2(Ca,trace="none", #不显示tracecol=coul, #修改热图颜色density.info = "none", #图例取消densitykey.xlab ='Correlation',key.title = "",cexRow = 1,cexCol = 1, #修改横纵坐标字体Rowv = F,Colv = F, #去除聚类margins = c(6, 6),cellnote = hM,notecol='black' #添加相关系数的值及修改字体颜色)
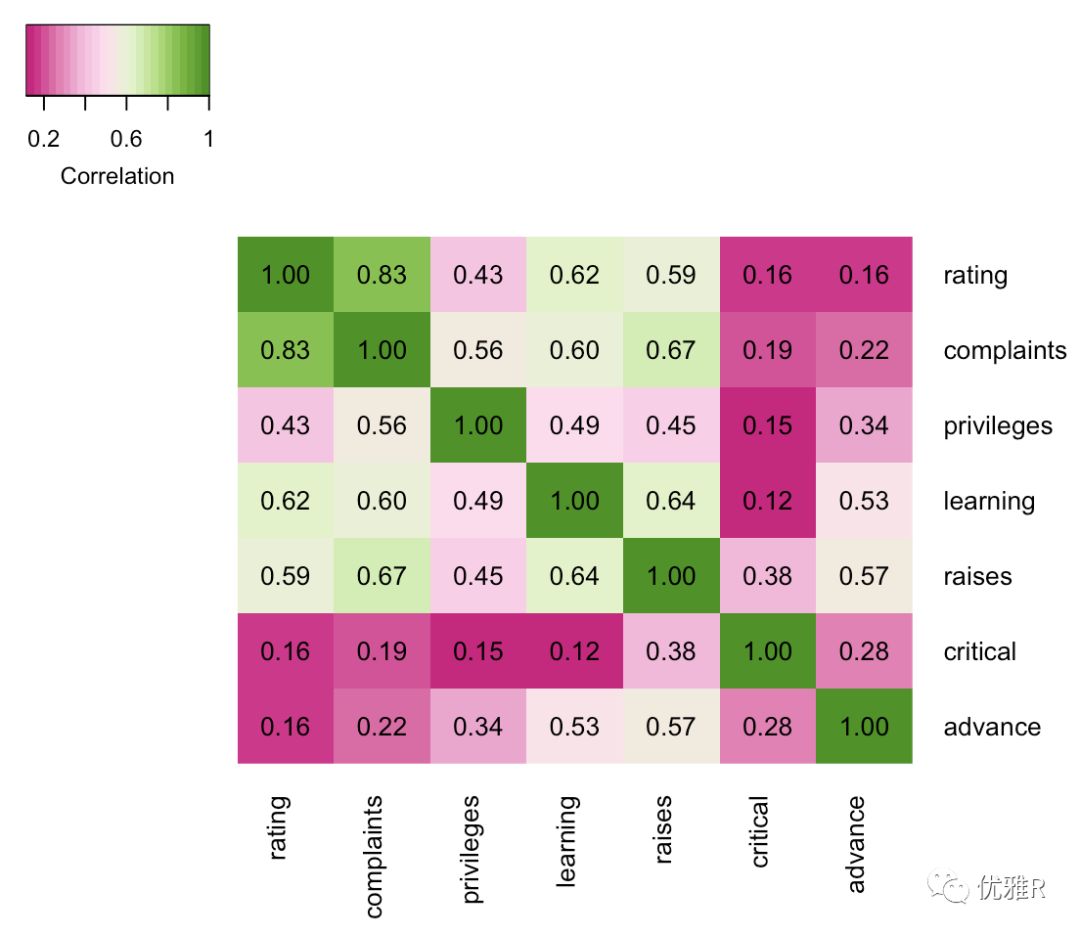
当然也可以按照相同顺序把相关性系数换成 pvalue。颜色也可以根据情况进行修改。其他的也可以进一步调整。

本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。


