在 RedHat 使用 gdc-client 下载 TCGA 数据
今天,只聊一下 RedHat/CentOS 下 gdc-client 安装的那些事。
gdc-client,官网地址:https://gdc.cancer.gov/access-data/gdc-data-transfer-tool,是由 GDC 官方提供的一个可以在命令行下批量下载 TCGA 数据的客户端工具。
在 gdc-client 官网可以看到 Mac、Windows 和 Ubuntu 的二进制版本下载,却唯独没看到 CentOS/RedHat 版本的!而且还给了我们一个提示说,如果你想要安装 RedHat Enterprise Release 6 版本的 gdc-client 需要跟 GDC 进行联系!!
如果你用 "gdc-client" + "centos6" 的关键字去谷歌,会发现大部分的答案都是教你用 Python2 的虚拟环境去安装 gdc-client。
其实,这些大部分都存在误导的成分,虽然 gdc-client 官网虽然没有提供 CentOS 6 的二进制程序包,但它托管在 GitHub 的源码我们是可以直接安装的,而且是只支持 Python 3!!
坑一:Python 2 引发 parse 模块异常
使用 Python 2 安装:
conda create -n Python2 python=2.7
source activate Python2
git clone https://github.com/NCI-GDC/gdc-client
cd gdc-client
python setup.py install 2>&1 | tee -a install.log
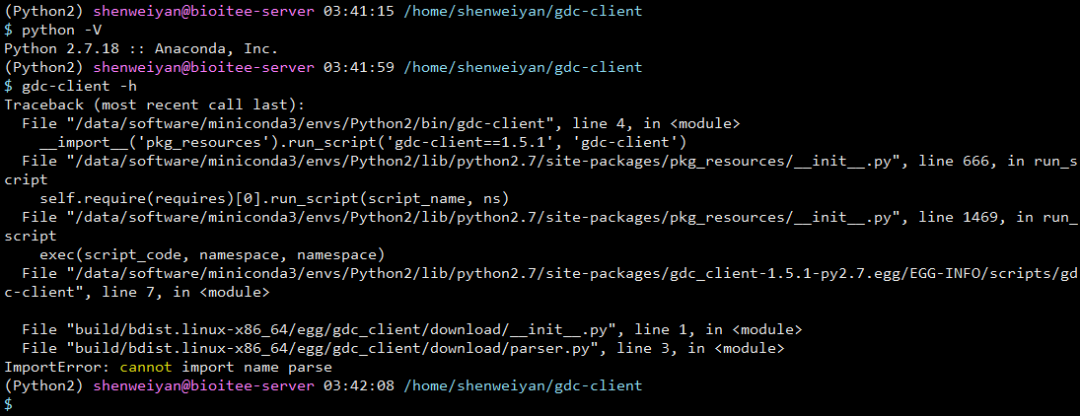
这种方法虽然看起来没什么问题,却会执行 gdc-client -h 提示parse模块异常。其原因是 build/bdist.linux-x86_64/egg/gdc_client/download/parser.py 的第三行 from urllib import parse as urlparse 是 py3 的语法:在 python 2.x 中的 urlparse 模块在 Python 3 中已经重命名为 urllib.parse 。
# Python 2 正确语法
from urlparse import urlparse
# Python 3 正确语法
from urllib import parse as urlparse
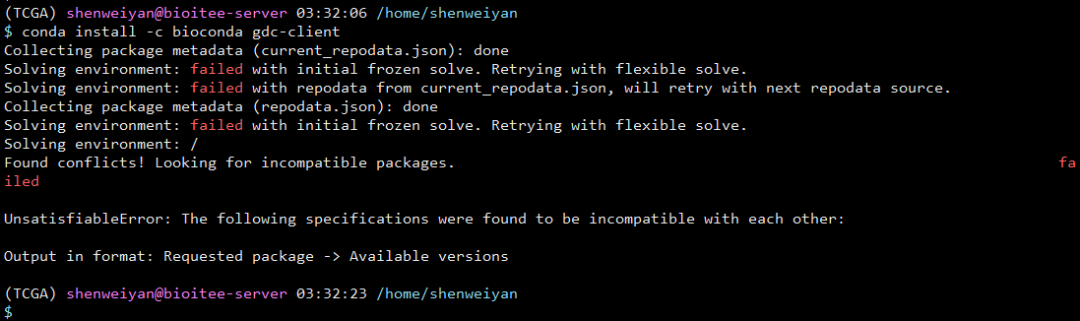
坑二:conda 安装无法响应
bioconda 虽然也提供了 gdc-client,但是本人 一直没法安装成功,可能是我的运气不太好!
最后,CentOS 6 的正确解锁姿势
在 CentOS 6 中使用源码方式安装:
conda create -n gdc python=3.7
source activate gdc
git clone https://github.com/NCI-GDC/gdc-client
cd gdc-client
pip install -r requirements.txt
python setup.py install 2>&1 | tee -a install.log
最后,打开 GDC 的官方《Data Transfer Tool Command Line Documentation》文档,查看在命令下怎么使用 gdc-client 下载你想要的 TCGA 数据吧!
2. 如何卸载 python setup.py install 安装的包?
6. Anaconda 环境下 R 包 ggraph_1.0.2 安装小记
7. 拒绝conda, 用virtualenv构建多版本的python开发环境
8. 癌症中克隆种群结构统计推断分析软件PyClone安装小记

本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。

