Galaxy Release_20.09 发布,新增多个数据上传组件
Galaxy Project(https://galaxyproject.org/)是在云计算背景下诞生的一个生物信息学可视化分析开源项目。
该项目由美国国家科学基金会(NSF)、美国国家人类基因组研究所(NHGRI)、哈克生命科学研究所(The Huck Institutes of the Life Sciences)、宾州州立大学网络科学研究所(The Institute for CyberScience at Penn State),以及约翰霍普金斯大学(Johns Hopkins University)提供支持,是目前生物医学研究领域最受欢迎的在线生物信息分析工具之一。
2020年10月左右,Galaxy Project 正式发布了 release-20.09 的版本;11月18日,发布这一版本的 announcement 文档。这里总结一下该版本一些主要的更新内容,为关注和从事 Galaxy 相关工作的中文用户提供参考。
1. 重点更新
1.1 可插拔的上传组件
@jmchilton为上传源编写了一个新的可插入接口。他最初添加了两个新的后端,现在您可以从 Dropbox 和您可能有权访问的任何 WebDav 服务器导入数据。这项工作在将来,会为开发人员更轻松地添加对其他新的个人数据源提供支持。
一个 Dropbox 帐户的示例:
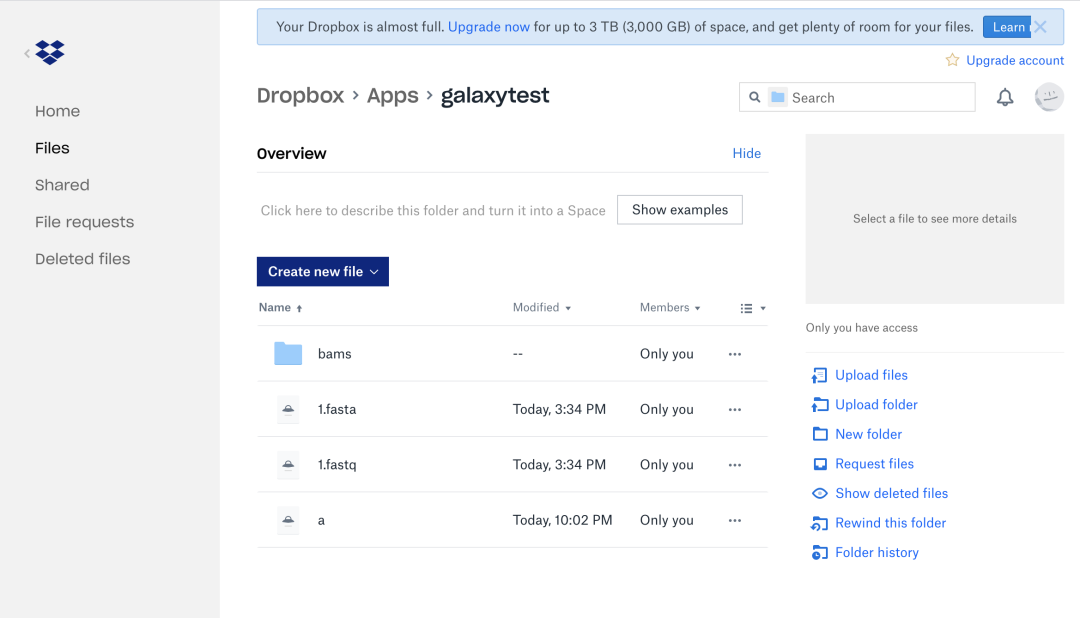
配置源后,将有一个用于 “选择远程文件(Choose Remote files)”的新按钮,然后您将能够查看和搜索您可能有权访问的所有远程数据位置。
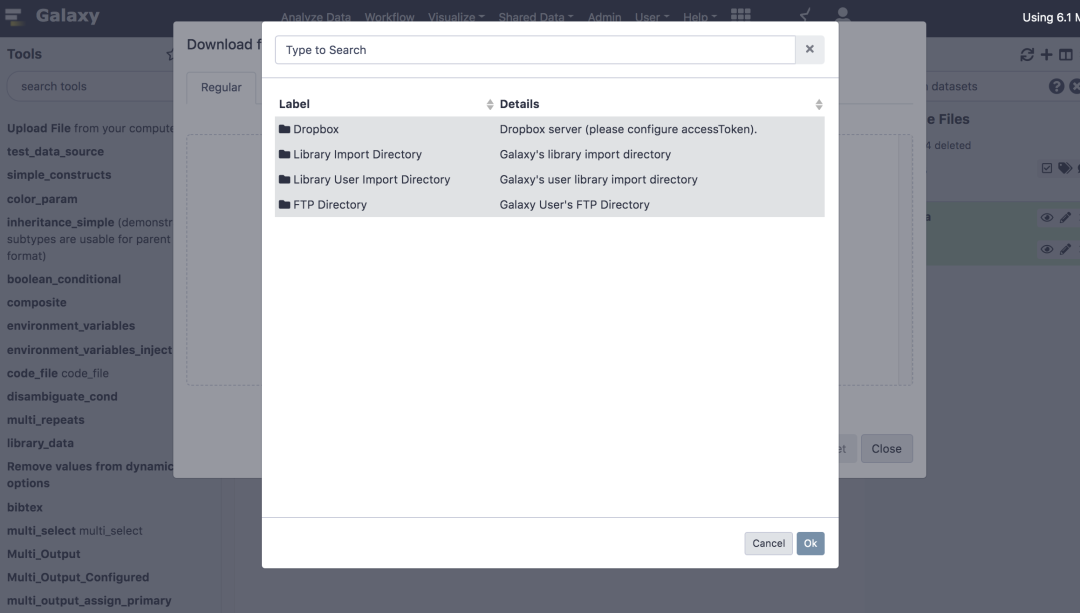
您将能够在 Galaxy 中轻松查看和加载文件!
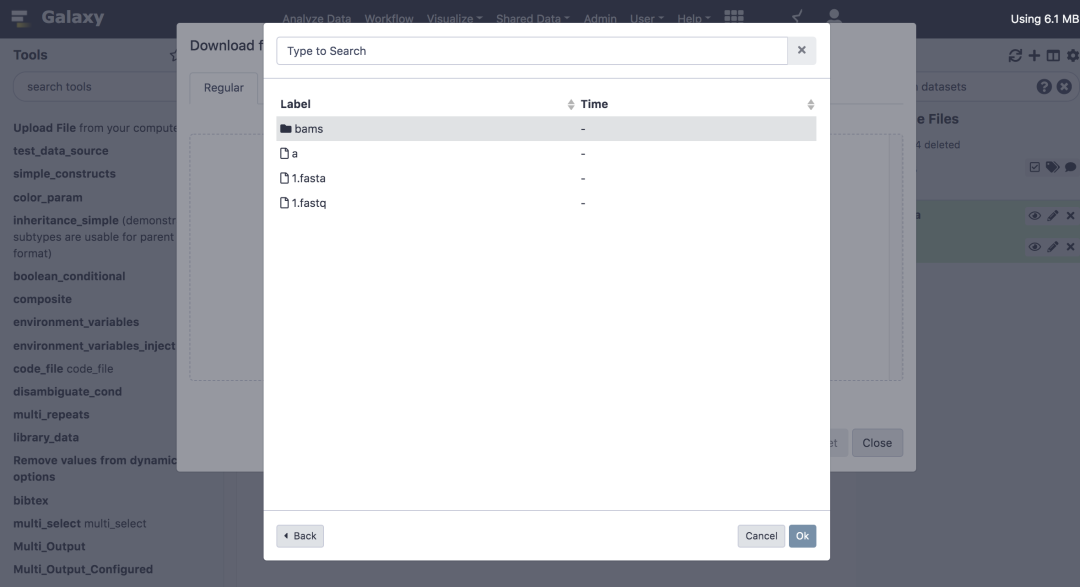
如果要使用这些服务,管理员首先需要配置这些功能,然后,需要在 Galaxy 的用户首选项中提供这些服务的凭据。
1.2 在 Galaxy 中接入 GTN
在 BCC2020 CoFest 期间,@hexylena和@shiltemann共同增加了对从 Galaxy 内部访问 Galaxy 培训网络教程的支持。点击导航栏的刻度帽图标将激活 GTN 界面。
对于更新的教程,工具将突出显示为蓝色按钮。单击后,这些按钮将隐藏 GTN,并直接将您带到 Galaxy 界面中正确工具的正确版本。用户将无需再花时间弄清楚教程中作者意味的是哪一个种工具!准备就绪后,您又可以轻松地跳回到培训材料。
Thanks @pvanheus for the video of it in action!
1.3 从工具表单直接上传数据
如果你曾经试过点击一个工具后,在工具页面开始进行参数配置,但忘记了先上传数据集,以至于你在上传数据后不得不重新开始之前的参数配置操作。参考 @jmchilton. Pull Request 9809,您可以直接在工具表单中上传数据集。1.4 改进的流程布局算法
旧的自动化流程布局算法喜欢在流程中隐藏数据流的信息。
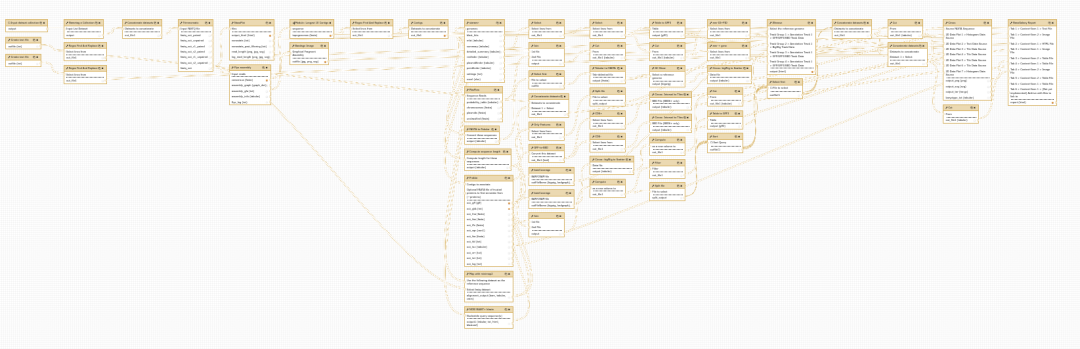
因此,20.09 版本用一种较新的布局算法代替了该算法,这应该使流程中的数据流更容易理解。您可能仍希望在流程自动布局后进行手动调整,但是希望这样可以节省一些时间。
1.4 改进的流程报告
提交一个或多个工作流程后,这些工作流程及其报告的状态页面报告已得到改进!
另外,20.09 在工作流报告语法中还添加了几个新功能,您可以在报告中使用这些功能。参考@OlegZharkov. Pull Request 9938
invocation-time -The iso time of the workflow invocation time.generate-time -The iso time of the time of generation of the Markdown.generate-galaxy-version -The Galaxy MAJOR_VERSION at the time of generation of the Markdown.dataset-type -The ext/format/datatype of the target dataset.dataset-name -The name of the target dataset.
1.5 可以自行删除帐户
在(希望的)极少数情况下,您可能想要关闭帐户并删除关联的数据。@astrovsky01 已实现用户自删除,因此您不再需要请求管理员为您执行此操作并等待他们的响应。参考Pull Request 10107
2. 新的可视化功能
GalaxyP MVP 可视化插件(感谢 @tmcgowan)。参考Pull Request 10136
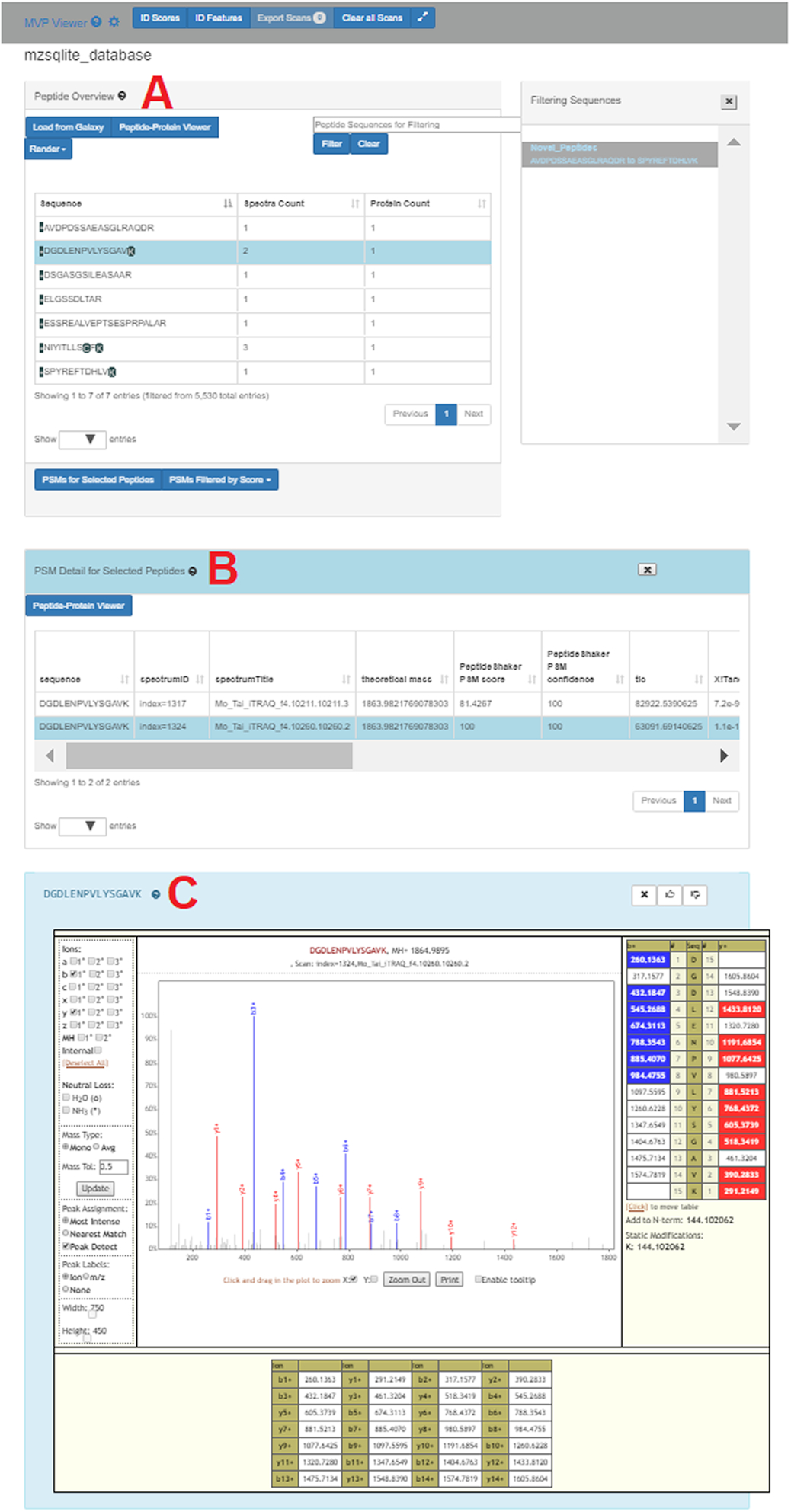
从他们的相关论文中:
[The MVP App] displays this example data where the list of variant peptides are shown in the Peptide Overview window (A). One of these peptides (sequence DGDLENPVLYSGAVK) has been selected in this list, and the button “PSMs for Selected Peptides” clicked to display the 2 PSMs that matched to this sequence, along with associated scoring metrics (B). Double-clicking on one of these PSMs opens the Lorikeet MS/MS viewer (C). Lorikeet [29] renders MS/MS spectra, providing a visualization of the annotated spectra that led to a PSM using the upstream sequence database searching software. C shows an example PSM, where the blue- and red-colored m/z peak values correspond to amino acid fragments that would be predicted to derive from the peptide sequence identified by this PSM.
3. 新的数据类型
从 2017 年开始,几个社区成员和提交者(@stevecassidy,@hexylena,@anuprulez,@bgruening,@mvdbeek)一起工作,以添加音频和视频相关的数据类型。现在,此 PR 已合并并可以在 Galaxy 中使用。另外,这些文件还包括针对这些文件的特定可视化效果,使您可以直接在 Galaxy 中查看音频或视频媒体。
增加 VCF_BGZIP 到 VCF 转换(感谢@almahmoud).Pull Request 7926
添加 NcbiTaxonomySQlite 类来描述 NCBI 分类标准 SQLite 数据库(感谢@pvanheus).Pull Request 9712
增加 last 工具的 lastdb 的数据类型(PR#2749)(感谢@RomainDallet).Pull Request 9872
增加 BLASTDBv5 数据类型(for blast >=2.8.1)(感谢@abretaud).Pull Request 9939
使用 compression_utils 对压缩数据集进行安全浏览(感谢@pvanheus).Pull Request 9990
添加 Anndata 监视功能(感谢@mtekman).Pull Request 10013,Pull Request 10555
修复 fastq_to_fqtoc 格式转换中的 OSError(感谢@bgruening).Pull Request 10042
不显示可选的不存在的元数据文件的下载链接.Pull Request 10068
将 "tgz" 数据类型添加到配置示例文件中,以用于 toolfactory 和任何其他生成 gzipped tar 文件的工具(感谢@fubar2).Pull Request 10080
修复在 Tabular 格式文件中 'int', ‘float' 列类型的检测(感谢@selten).Pull Request 10155
Decrease pysam verbosityPull Request 10225
从示例配置文件中删除对丢失类型的引用.Pull Request 10269
Fix tuple assignment (fixes anndata, scanpy tool tests)Pull Request 10293
修复
Wav数据类型以从Audio继承(感谢@nsoranzo).Pull Request 10312Fix wiggle estimated_display_viewportPull Request 10333
4. 更新内置工具
修复并简化内置修剪工具(感谢@wm75).Pull Request 10145
Allow unary plus and minus in filtering(感谢@wm75).Pull Request 10154
修复 filter.py 失败的所有表达式(感谢@almahmoud).Pull Request 10266
后话
从个人而言,Galaxy Project 是一个极好的开源学习项目,它包含了从生信到数据库、系统,以及服务器、集群,前端等等的知识;从公司而言,也是从传统命令行生信分析向可视化界面处理的一个很好开源案例,值得借鉴与参考。
本文章的所有链接,以及可用于 Galaxy 管理员、开发人员更加详细的更新文档,可以点击文章底部"阅读原文"进行查阅。
本文由“壹伴编辑器”提供技术支持
如果您觉得这篇文章对您有点用的话,麻烦您为本文来个四连:转发分享、点赞、点在看、留言,因为这将是我写作与分享更多优质文章的最强动力!
本文由“壹伴编辑器”提供技术支

本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。


