使用 Docker 分析高通量测序数据
端午节假期,先祝各位 Bio IT 的爱好者们,节日快乐!
做生信的童鞋想要学习 Docker,或者使用 Docker+Pipeline 封装自己的一套数据分析流程,相信一定不能错过胡博强老师在2017年写这篇《[Docker]使用阿里云 + Docker 分析高通量测序数据——RNA-Seq 与 ChIP-Seq. - Boqiang Hu》教程,这个教程同时也以推文的方式发布在了 2017-03-21 生信技能树公众号上,感兴趣的同学可以自己去翻一下。
根据教程+tangEpiNGSInstall 仓库提供的原始测试数据,本人这两天测试跑了一下,发现了一点点小问题。
$ git clone https://github.com/shenweiyan/tangEpiNGSInstall.git
$ tree
.
└── tangEpiNGSInstall
├── Dockerfile
├── README.md
├── settings
│ ├── run_chipseq.py
│ ├── run_chipseq.sh
│ ├── run_mRNA.py
│ ├── run_mRNA.sh
│ ├── scripts_chipseq.py
│ └── scripts_mRNA.py
├── src
│ └── run_sample.sh
├── test_fq
│ ├── H3K4me3
│ │ ├── test.1.fq.gz
│ │ └── test.2.fq.gz
│ ├── Input
│ │ ├── test.1.fq.gz
│ │ └── test.2.fq.gz
│ └── sample.tab.xls
└── test_fq_RNA
├── SampleA1
│ ├── test.1.fastq.gz
│ └── test.2.fastq.gz
└── sample.tab.xls
8 directories, 17 files
$ mkdir -p results database_ChIP/mm10
$ chmod 777 results database_ChIP/mm10 # avoiding Permission issue
$ tree
.
├── database_ChIP
│ └── mm10
├── results
└── tangEpiNGSInstall
├── Dockerfile
├── README.md
├── settings
│ ├── run_chipseq.py
│ ├── run_chipseq.sh
│ ├── run_mRNA.py
│ ├── run_mRNA.sh
│ ├── scripts_chipseq.py
│ └── scripts_mRNA.py
├── src
│ └── run_sample.sh
├── test_fq
│ ├── H3K4me3
│ │ ├── test.1.fq.gz
│ │ └── test.2.fq.gz
│ ├── Input
│ │ ├── test.1.fq.gz
│ │ └── test.2.fq.gz
│ └── sample.tab.xls
└── test_fq_RNA
├── SampleA1
│ ├── test.1.fastq.gz
│ └── test.2.fastq.gz
└── sample.tab.xls
11 directories, 17 files
$ docker pull hubq/tanginstall:latest
$ docker run -v /data/docker/train/tangEpiNGSInstall/test_fq:/fastq -v /data/docker/train/results:/home/analyzer/project -v /data/docker/train/database_ChIP/mm10:/home/analyzer/database_ChIP/mm10 -v /data/docker/train/tangEpiNGSInstall/settings/:/settings/ --env ref=mm10 --env type=ChIP hubq/tanginstall:latest
INFO @ 2021-06-10 03:29:48,154: Begin checking input files.
INFO @ 2021-06-10 03:29:48,154: Input database files were all put in /home/analyzer/database_ChIP/mm10.
INFO @ 2021-06-10 03:29:48,154: Input fasta /home/analyzer/database_ChIP/mm10/mm10.fa not find. Now download from UCSC
INFO @ 2021-06-10 03:45:01,604: /home/analyzer/database_ChIP/mm10/mm10.fa generation done!
INFO @ 2021-06-10 03:45:01,605: Fasta were not indexed.
INFO @ 2021-06-10 03:45:02,105: Now build index using bwa.
INFO @ 2021-06-10 03:48:20,683: Building index done!
INFO @ 2021-06-10 03:48:20,683: Genome GTF file were not found.
INFO @ 2021-06-10 03:48:21,184: Now download refGene file from UCSC.
INFO @ 2021-06-10 05:03:38,768: Generate refGene done!
INFO @ 2021-06-10 05:03:38,768: RepeatMask file were not found.
INFO @ 2021-06-10 05:03:39,269: Now download rmsk file from UCSC.
INFO @ 2021-06-10 05:06:03,592: Generate RepeatMask done!
Traceback (most recent call last):
File "/home/analyzer/module/ChIP/run_chipseq.py", line 148, in <module>
main()
File "/home/analyzer/module/ChIP/run_chipseq.py", line 126, in main
samp_peak.get_idr_stat()
File "/home/analyzer/module/ChIP/frame/module02_call_peaks.py", line 244, in get_idr_stat
mod_Stat.IDR_Stat()
File "/home/analyzer/module/ChIP/frame/module00_StatInfo.py", line 113, in IDR_Stat
f_idr_out = open(file_idr_out,"w")
IOError: [Errno 2] No such file or directory: '/home/analyzer/project/ChIP_test/StatInfo/IDR_result./home/analyzer/project/ChIP_test/sample.tab.xls'
cp: cannot stat `03.2.Peak_mrg/*/*_treat_minus_control.sort.norm.bw': No such file or directory
cp: cannot stat `03.3.Peak_idr/*/*.conservative.regionPeak.gz*': No such file or directory
cp: cannot stat `StatInfo/*': No such file or directory

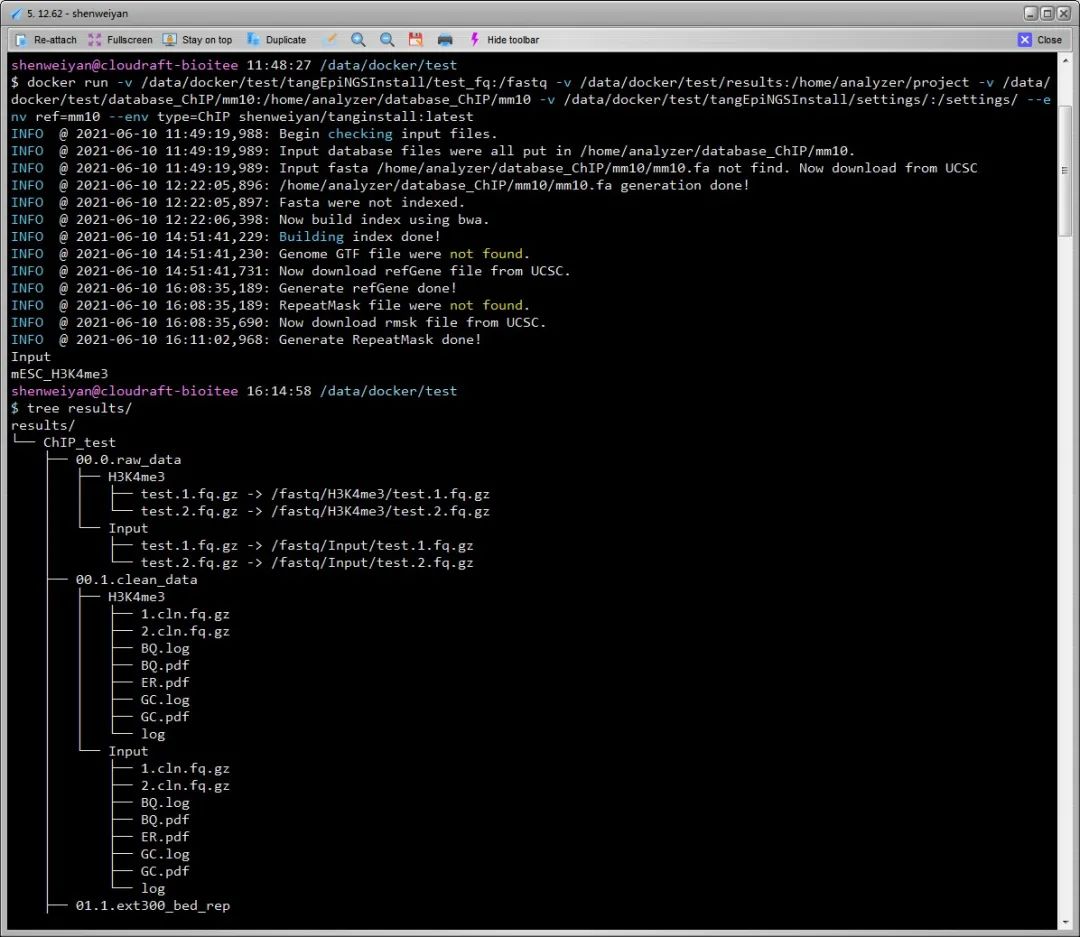
简单说一下这个镜像的几点细节。
整个镜像体积比较大,总共约 7.37GB,pull 下来可能比较慢。
- 如果没有 ref(hg19/hg38 or mm9/mm10),镜像执行过程中会首先执行下载,然后拆分合并,建立 index。
db01.DownloadRef.sh
$ cat db01.DownloadRef.sh
ref=$1
dir_database=/home/analyzer/database_ChIP/$ref
dir_path=/home/analyzer/module/ChIP
cd $dir_database
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/bigZips/chromFa.tar.gz
tar -zxvf $dir_database/chromFa.tar.gz
for i in {1..22} X Y M
do
cat $dir_database/chr$i.fa
done >$dir_database/${ref}.fa && rm $dir_database/chr*fa
db02.RefIndex.sh
$ cat db02.RefIndex.sh
ref=$1
dir_database=/home/analyzer/database_ChIP/$ref
bwa_exe=/software/install_packages/bwa-0.7.5a/bwa
samtools_exe=/software/install_packages/samtools-0.1.18/samtools
div_bins_exe=/home/analyzer/module/ChIP/bin/div_bins/bed_read
$samtools_exe faidx $dir_database/${ref}.fa
$bwa_exe index $dir_database/${ref}.fa
$dix_bins_exe -b 100 $dir_database/${ref}.fa.fai $dir_database/columns.100.bed
$dix_bins_exe -b 1000 $dir_database/${ref}.fa.fai $dir_database/columns.1kb.bed
cut -f 1-2 $dir_database/${ref}.fa.fai >$dir_database/${ref}.fa.len
db03.RefGene.sh
$ cat db03.RefGene.sh
ref=$1
dir_database=/home/analyzer/database_ChIP/$ref
bedtools_exe=/software/install_packages/bedtools2/bin/bedtools
ucsc_dir=/software/install_packages/UCSC
bin=/home/analyzer/module/ChIP/bin
dir_path=/home/analyzer/module/ChIP
cd $dir_database
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/database/refGene.txt.gz
### remove chromosome fragments(unassembled).
for i in {1..22} X Y M
do
zcat $dir_database/refGene.txt.gz | grep -w chr$i
done >$dir_database/tmp
mv $dir_database/tmp $dir_database/refGene.txt
# refGene.bed
cat $dir_database/refGene.txt |\
awk '{
tag="noncoding";
if($4~/^NM/){tag="protein_coding"};
OFS="\t";
print $3,$5,$6,$2,$4,$10,$11,tag,$13
}' /dev/stdin |\
python $bin/s03_genePred2bed.py /dev/stdin |\
$bedtools_exe sort -i /dev/stdin >$dir_database/refGene.bed &&\
# region.Intragenic.bed
# For novo lncRNA detection
$bin/find_ExonIntronIntergenic/find_ExonIntronIntergenic \
$dir_database/refGene.bed \
$dir_database/${ref}.fa.fai >$dir_database/pos.bed &&\
grep -v "Intergenic" $dir_database/pos.bed |\
awk '{OFS=" ";print $1,$2,$3,"Intragenic"}' /dev/stdin \
>$dir_database/region.Intragenic.bed &&\
# refGene.gtf
# For mapping
zcat $dir_database/refGene.txt.gz |\
cut -f 2- |\
$ucsc_dir/genePredToGtf file stdin /dev/stdout |\
grep -w exon |\
$bedtools_exe sort -i /dev/stdin >$dir_database/refGene.gtf &&\
cat $dir_path/database/ERCC.gtf >>$dir_database/refGene.gtf
db04.rmsk.sh
$ cat db04.rmsk.sh
ref=$1
dir_database=/home/analyzer/database_ChIP/$ref
bedtools_exe=/software/install_packages/bedtools2/bin/bedtools
ucsc_dir=/software/install_packages/UCSC
bin=/home/analyzer/module/ChIP/bin
dir_path=/home/analyzer/module/ChIP
cd $dir_database
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/database/rmsk.txt.gz
zcat $dir_database/rmsk.txt.gz |\
awk '{
OFS="\t";
print $6,$7,$8,$2,".",".",".","("$9")",$10,$11,$12 "/" $13,$14,$15,$16,$17
}' /dev/stdin |\
tail -n +2 /dev/stdin >$dir_database/chrom.bed
for i in {1..22} X Y M
do
grep -w chr$i $dir_database/chrom.bed
done >$dir_database/tmp
mv $dir_database/tmp $dir_database/chrom.bed
$bedtools_exe sort -i $dir_database/chrom.bed >$dir_database/chrom.sort.bed
为节省下载时间,建议事先准备好 ${ref}.fa,如果没有,也可以先下载好以下文件。
# db01.DownloadRef.sh:
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/bigZips/chromFa.tar.gz
# db03.RefGene.sh:
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/database/refGene.txt.gz
#db04.rmsk.sh:
wget http://hgdownload.soe.ucsc.edu/goldenPath/${ref}/database/rmsk.txt.gz
bwa index(db02.RefIndex.sh)非常耗时,个人一个4核16G配置的服务器也跑了大约2.5小时。


生信服务器 | 更改 CentOS/RHEL 6/7 中的时区





本文分享自微信公众号 - 生信科技爱好者(bioitee)。
如有侵权,请联系 support@oschina.cn 删除。
本文参与“OSC源创计划”,欢迎正在阅读的你也加入,一起分享。


