R语言--数据框
创建数据框
因为数据框的本质是由一堆向量或者因子构成的列表,其中的每一个向量或者因子代表了一列。因此,数据框可以包含不同类型的数据(数值型、布尔型或字符型),但是每一列的数据类型必须相同。
data.frame
我们可以通过data.frame()函数将相同长度的向量数据,构建一个数据框
name <- c("Jane","Maria","kangkang","Micheal",'Yukio',"sansa")
age <- c(15,14,15,15,16,14)
country <- c("Canada","Cuba","China","American","Japan","American")
info <- data.frame(name,age,country)
as.data.frame
可以通过as.data.frame将列表数据转化成数据框
info <- list(name = name,age = age,country = country)
info <- as.data.frame(info)
数据输入
通过read.csv()函数、read.table()函数读入的表格文件,以及从数据库读入的数据都会返回一个数据框
访问元素
通过行列的下标访问
# 查看第一列
info[,1]
# 查看第一行
info[1,]
# 查看第二行第三列
info[2,3]
通过列名或列下标查看列
info[1:2] # 查看第一至第二列
info[c("name","age")] # 查看列名为name,age的列
[[]]和$
若只查看数据的某一列还可以通过 [[]] 和 $
info[[2]] # 查看数据的第二列,这和上面的列的下标查看很相似,但是返回的数据类型不同,上面返回的是数据框,这返回的是向量
info[["name"]] # 查看数据中中列名为name的列,和上面列名查看相似,但返回的数据类型不同
info$name # 查看数据中的name列
增加和删除列
通过 $ 增加列
上面提到我们可以通过 $ 访问数据框某列,我们也可以通过 $ 向数据框添加某列
info$gender <- c("female","female","male","male","male","female") # 向info数据框中添加名为gender列,列的数据必须和其它列的长度一致,如果不一致将自动循环补齐,而循环过程中,长度必须为整数倍
info$grade <- c("class 1","class 2","class 3") # 其他列的长度为6,是该向量的2倍,向量循环2次。等价于info$class <- c("class 1","class 2","class 3","class 1","class 2","class 3")
info$grade <- c("class 1","class 2","class 3","class 4") # 非整数倍将出错
可以通过下标删除行或列
info[-1,] # 删除第一行
info[,-1] # 删除第一列
还可以通过将某一列赋值为NULL,来删除该列
info$name <- NULL
另外还可以按照条件索引、匹配等来删除数据框的某行或者某列这部分见数据框匹配
重命名数据框列名
names()
names()函数可以获得/命名一个对象的名称。返回/设置的为一个字符向量
names(info) <- c("newname","newage","newcountry","newgender","newgrade")
因为是字符操作,所以可以运用字符穿的匹配,替换等操作结合向量元素替换操作来重命名
names(info)[names(info) == "name"] <- c("rename") # 以及names(info) == "name",获取列名并判断列名是否等同于name,然后names函数将与条件匹配的元素重新赋值为"rename"
names(info)[grep("name",names(info))] <- c("newname")
names(info)[grepl("name",names(info))] <- c("newname")
...
数据框列重新排序
通过列的下标排序。其实这个就是和数据框通过列的提取,然后将提取的列重新构成一个数据框而已。所以,最好细心,因为不小心就会把数据只得到某几列。如下面info数据框有5列,想调整第二列和第三轮的顺序
info <- info[c(1,3,2)] # 此时相当于提取了原info数据框的第一列、第三列、第二列然后赋值给info。而第四列、第五列就删除了
info <- info[c(1,3,2,4,5)] #应该将整个数据都提取,只是顺序重新排列。这样比较麻烦
另外可以把下标替换为列名,然后和下标一样的用法
info <- info[c("name","country","age","gender","grade")]
匹配、提取
subset()
subset()函数根据条件返回向量或矩阵或数据框
## S3 method for class 'data.frame'
subset(x, subset, select, drop = FALSE, ...)
- x:带匹配的数据框
- subset:表明要保留的行或者元素的逻辑表达式。(行)
- select:表明要从数据框中挑选的列(列)
- drop:布尔值,表示返回的是数据框还是向量。默认FALSE,即数据框
male <- subset(info,gender == "male") # gender == "male",因为subset必须是逻辑值,所以行的提取还可以通过返回为逻辑的字符匹配操作,来筛选
male <- subset(info,grepl("a",info$name)) # 提取info数据框中name列中元素带字母a的行
male <- subset(info,gender == "male",select = c(gender,name,age)) # select 提取列,所以提取info 数据框中gender为male的行,然后提取了gender,name,age这三列
newInfo <- subset(info,select = grep("a",names(info))) # 提取列名中含有字母a的列
male <- subset(info,gender == "male"|country == "china") #另外还可以通过|(或)&(且)等进行跳进筛选
newInfo <- subset(info,age >= 15,select = grep("a",names(info))) #提取age中大于等于15的行,列名中含有字母a的列
跟据下标匹配、提取或删除
subset是根据返回的逻辑值来匹配、提取。另外还可以根据字符串下标来提取,我们通过返回匹配值为下标,然后根据该下标来提取或删除。而我们知道匹配字符串返回下标的函数有which、grep、str_which
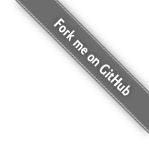
newInfo <- info[which(info$name == "Jane"),] #提取info数据框name列中为Jane的所有行。删除的话是直接就是直接加删除符号(-),行列都是一样以用的
newInfo <- info[grep("Maria",info$name),3] # 提取info数据框name列中为Maria的行,第三列。当然列也是可以根据条件筛选的,只需要匹配函数返回的是下标
newInfo <- info[str_which(info$name,"sansa"),grep("gender",names(info)] # 提取info数据框name列中为sansa的行,列名为gender的列
newInfo <- info[grep(".*?(a).*?",info$name),] # 提取name中包含字符 a 的行
match
当然这是很小的数据,但是,假如我们手里有几千个基因的数据,老板甩给你几百个基因,找出这几百个基因或者找出除了这几百个基因外其它基因就可以用这个啦
转化变量
分类变量转化为另一个分类变量
比如说数据框中有一个分类变量"female","male"。我们根据这两个变量,创建一个变量(drink),"female"用来表示喝的饮料("juice"),"male”表示喝的啤酒("beer")
oldvals <- c("female","male")
newvals <- factor(c("juice","beer")) # 此处顺序一一对应,即female对应juice,male对应beer。所以这一个顺序自己仔细点
info$drink <- newvals[match(info$gender,oldvals)] #match将info中gender列元素与oldvals匹配,元素与oldvas中第一个匹配返回1,与第二个匹配返回2。然后其实就是c("juice","beer")[c(1,1,2,2,2,1)]
另外还有一种就是使用向量索引的方式
info$drink[info$gender == "female"] <- "juice"
info$drink[info$gender == "male"] <- "beer"
info$drink <- factor(info$drink)
#还可以通过|(或)、&(与)操作符,多个条件定义。如我们现在在性别为男性、年纪大于等于16的定义为喝白酒(liquor),将小于等于15的定义为喝啤酒
info$drink[info$gender == "male" &info$age >= 16] <- "liquor"
info$drink[info$gender == "male" & info$age <= 15] <- "beer"
连续变量转化为分类变量
比如我们现在有一个变量年纪,其中包括3岁到80岁的数据记录,我们将年龄小于18岁的定义为未成年,将大于60岁的定义为老年,将大于18岁小于60岁的定义为中年。我们可以通过cut函数,先创建边界值,0到18岁,18到60岁,60岁到无穷大,这三个区间。同时cut函数会将每个年纪进行匹配,看这个年纪在什么区间。
cut(x, breaks, labels = NULL,
include.lowest = FALSE, right = TRUE, dig.lab = 3,
ordered_result = FALSE, ...)
- x:待分区的连续数据
- breaks:边界值,定义区间
- labels:各个区间(类别)的结果标签
- right:区间闭合的位置,right = TRUE,表示区间右边闭合,即(18,60]
# 因为之前的年龄设置都太小,重新赋值
info$age <- c(13,15,25,35,60,65)
info$ageclass <- cut(info$age,breaks = c(0,18,60,Inf),labels = c("teenager","adult","aged")) # 定义年龄区间(0,18],标签teenager。(18,60],标签adult。(60,inf],标签aged。然后一一定位,然后创建标签变量,即分类变量
单个变量转换
单个变量转换即对数据框中的某一个变量进行公式计算,并重新赋值一个变量,这有三个方法,运用 $ 引用列、使用transform()函数、使用plyr包中的mutate()函数
transform(`_data`, ...)
- _data:待转换的对象
- ...:生成标签 = 值 的参数
mutate(.data, ...)
比如说我们现在他们有一门考试分数,满分一百的,我们将它转化为满分150。
info$score <- c(100,98,95,96,99,93) # 添加成绩
info$newscore <- info$score * 1.5 # 第一种通过 $ 引用列
info <- transform(info,newscore = score * 1.5) # 通过transform函数转化
library(plyr)
info <- mutate(info,newscore = score * 1.5) # 通过plyr包中的mutate函数转化
分组转换
分组转换就是数据框中有分组变量(一个或多个),然后我们对各个分组进行统计分析,并将统计结果生成新列看可以使用plyr包中的ddply函数。
ddply(.data, .variables, .fun = NULL, ..., .progress = "none",
.inform = FALSE, .drop = TRUE, .parallel = FALSE, .paropts = NULL)
举个例子,比如测脉搏,所有的脉搏数据在一列,有两个分组变量,性别和有没有服用某种药,这样我们就可以统计不同性别的脉搏情况,服用和不服用药的脉搏情况,以及不同性别在有服用和不用药情况下的脉搏情况。比如我们要分析男性各自脉搏和所有男性脉搏均数之间的差异,以及女性各自脉搏和所有女性脉搏均数之间的差异。
info$pulse <- c(58,60,59,56,61,63) # 添加脉搏数据
info <- ddply(info,"gender",transform,mpulse = pulse - mean(pulse)) # 首先会根据"gender"将数据分组分成两个数据框,然后对两个数据框调用transform函数,而transform函数定义为mpulse = pulse - mean(pulse),最后赋值给info
分类汇总数据
其实这个和上面原理是完全一样的,只是应用的函数不同而已,以及生成的数据框不同。另外这个其实我觉得应该放在统计篇。。
举例子,统计服药和不服药情况下的,男性和女性脉搏平均数,也就是男性服药脉搏,男性不服药脉搏,女性服药脉搏,女性不服药脉搏
ddply(info,c("gender","pill"),summarise,mean_pulse = mean(pulse)) # 根据gender(性别)和pill(是否服药),分组,然后计算各组pulse的平均数,并将该数赋值给变量mean_pulse。ddply返回的是一个数据框

另外还可以通过aggregate()函数完成分组情况的统计,以及lapply、apply、tapply等
aggregate(x, by, FUN, ..., simplify = TRUE, drop = TRUE)
- x:一个R的对象,要应用公式的数据,即要进行计算的公式。
- by:一个分组元素列表,其中每个元素都是x数据框中的变量,也就是用分组变量组合成列表
- FUN:一个函数,这个函数应用于所有分组子数据框,进行数据统计
- simplify:逻辑值,表示结果是否简化为一个向量或者列表
所以上面的还可以用aggregate()
aggregate(info$pulse,by = list(info$gender,info$pill),mean)
分组转换之split
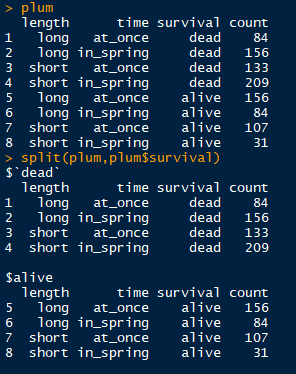
split函数可以将一个数据框根据分组变量转化为列表,列表中的元素为根据变量分组的数据框。有时候分组汇总用起来有点麻烦的时候可以用这个
维度转换
长变宽之dcast
dcast(data, formula, fun.aggregate = NULL, ..., margins = NULL,
subset = NULL, fill = NULL, drop = TRUE,
value.var = guess_value(data))
- data:要变化的数据框
- formula:变化公式,一般包括标识变量(需要保留的列)和可变变量(转化生成新列的列),两个变量之间用 ~ ,左边标识标识变量,右边标识可变变量
- value.var:数据列名,即该数值会被填充到新列
以下列gcookbook包中的一个名为plum的数据框为例
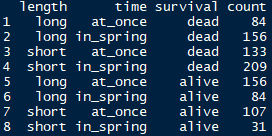
library(reshape2)
dcast(plum,length + time ~ survival,value.var = "count")
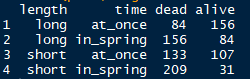
长变宽之spread
spread(plum,survival,count)

长变宽之unstack
这个函数貌似比较麻烦一点,目前看到的都是只有值和可变变量,只有两个,其它数据会被清除。。
unstack(x, form, ...)
- x:待转换的对象
- form:一个两边的公式,公式左边是值,右边是可变变量
unstack(plum,count ~ length)
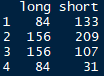
然后其它变量被自动清除,试过以下都不行
unstack(plum)
unstack(plum,count ~ survival + length +time)
宽变长之melt
melt(data, id.vars, measure.vars,
variable.name = "variable", ..., na.rm = FALSE, value.name = "value",
factorsAsStrings = TRUE)
- data:待转变的数据框
- id.vars:标识变量,表明哪些值要汇集到一起。可以是整数(变量列的下标)或者是字符串(变量的名称)
- measure.vars:度量变量,默认是除标识变量以外的所有变量,这些度量变量的名称会被放到一个叫variable.name列,而它们对应的取值则放到一个名为value.name的列中
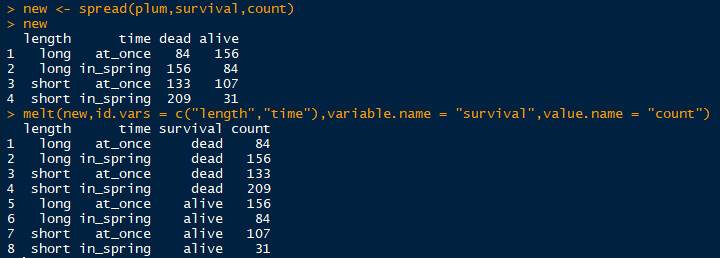
宽变长gather
gather(data, key = "key", value = "value", ..., na.rm = FALSE,
convert = FALSE, factor_key = FALSE)
- data:要变化的数据框
- key:相当于melt函数中的variable.name
- value:相当于melt函数中的value.name
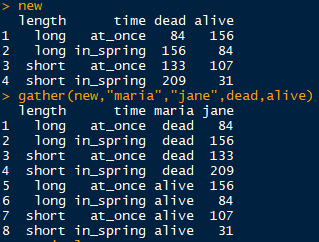
数据框合并
cbind和rbind
注意的点:cbind函数合并的数据框的变量长度(行数)要一致,因为是按列绑定。rbind函数合并的数据框变量数(列数)要一致并且列的名称要相同。
cbind(..., deparse.level = 1)
rbind(..., deparse.level = 1)
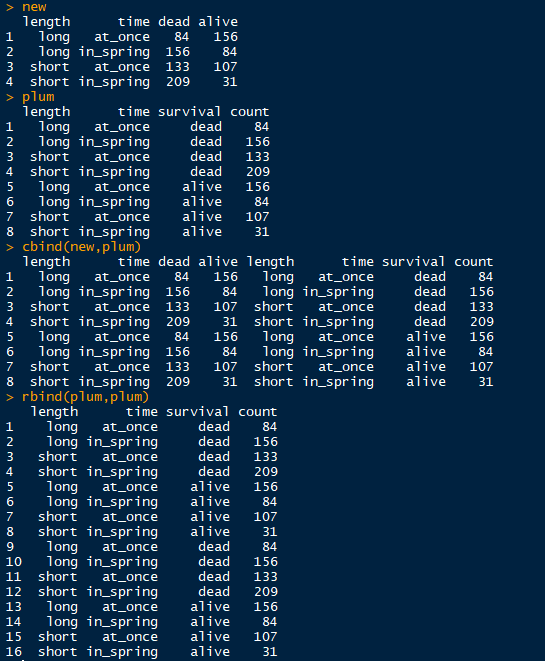
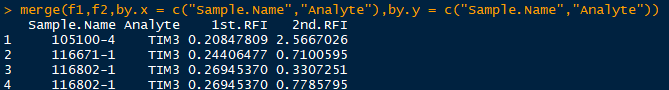
merge
merge(x, y, by = intersect(names(x), names(y)),
by.x = by, by.y = by, all = FALSE, all.x = all, all.y = all,
sort = TRUE, suffixes = c(".x",".y"), no.dups = TRUE,
incomparables = NULL, ...)
- x:数据框x
- y:数据框y
- by.x:x中的变量
- by.y:y中的变量
以例子说明,现在有两批样本的实验数据。
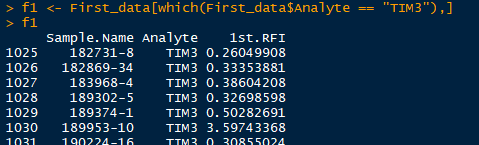
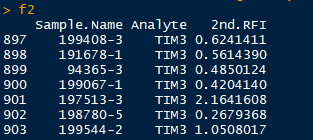
截图中都是只显示一小部分,这两批实验数据中,样本有些相同有些不同,两批实验所用抗体相同。现在我们来匹配合并两个数据框中样本、抗体相同的数据
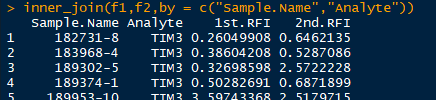
dplyr包
dplyr包中的inner_join()、left_join()、right_join()、full_join()。他们的用法完全一样,只是返回的值稍微有点不同
- inner_join:返回两数据框中匹配的值,如果不匹配则不返回。如x有60行,y有60行,按匹配条件,x和y有50行匹配,则显示这50行匹配合并的数据
- left_join:返回x数据框中的所有行,以及数据框x和y中的所有列。如果x中的行在y中未匹配,则在新列中该数据为NA
- right_join:返回y数据框中的所有行,以及数据框x和y中的所有列。如果y中的行在x中未匹配,则在新列中该数据为NA
- full_join:返回x和y的所有行和列。不匹配的各自为NA
inner_join(x, y, by = NULL, copy = FALSE, suffix = c(".x", ".y"),
...)
数据类型转化
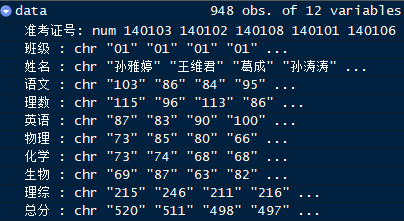
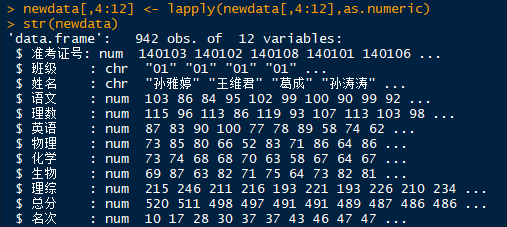
这些分数为字符型,现在我们将它转化为数值型,以便后面我们进行统计
如果我们直接对整个数据框套用as.numeric,将报错:(列表)对象不能被强制转换为双精度类型。

看看as.numeric()函数API
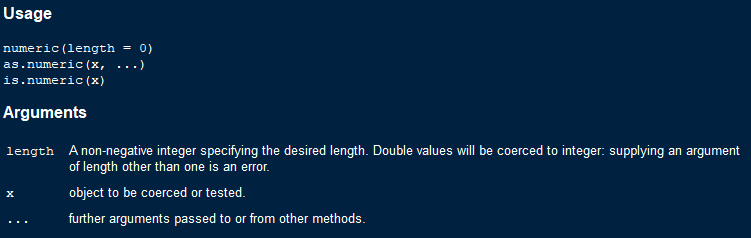
可以看到其中的length参数,意思是:该参数为非负整数,双精度值将被强制转换为整型,提交的长度参数大于1时将报错。这个报错的意思是,只能针对一维向量(矩阵和数组是特殊的向量)强制将元素转换为数值型。
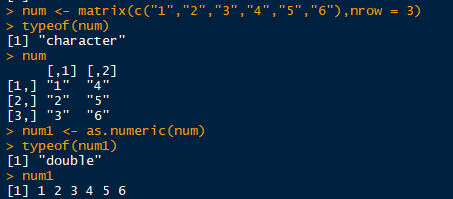
数据框的本质是多个向量组合构成,即是多维。。因此,直接对整个数据框应用该函数将报错,而对其中单列(单个向量)是不会报错的。
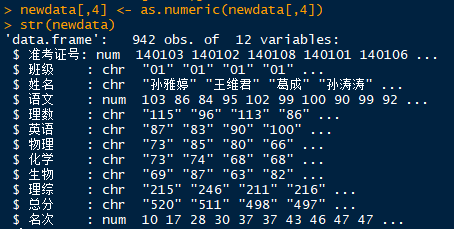
这个时候我们就可以运用一些函数,将as.numeric()运用到数据框中单列,比如lapply()

它的意思是lapply()函数返回一个与X(我们要处理的对象)长度相等的列表,列表中的每个元素是X中对应元素运用FUN(函数)的结果。