R语言版本的bedtools--bedtoolsr

bedtools是一款非常强大的用于处理bed,vcf,gff等格式数据的工具,该软件由犹他大学的Quinlan实验室开发。但是目前bedtools主要提供的是在linux,unxi等操作系统环境下的“命令行”运行方式,然而,对于想要使用R语言进行bedtools命令操作的科研人员来说就显得比较麻烦。今天,我就给大家介绍一款由北卡罗来纳大学教堂山分校开发的R语言版本的bedtools--bedtoolsr。话不多说,直接上干货。
- 安装bedtoolsr
install.packages("devtools")
devtools::install_github("PhanstielLab/bedtoolsr") # 首次使用需要安装
- 加载bedtoolsr
library(bedtoolsr)
- 生成测试数据
A.bed <- data.frame(chr = c("chr1", "chr1", "chr3"), start = c(10, 100, 50), end = c(40, 150, 110))
B.bed <- data.frame(chr = c("chr1", "chr5"), start = c(20, 60), end = c(35, 80))
print(A.bed)
# chr start end
#1 chr1 10 40
#2 chr1 100 150
#3 chr3 50 110
print(B.bed)
# chr start end
#1 chr1 20 35
#2 chr5 60 80
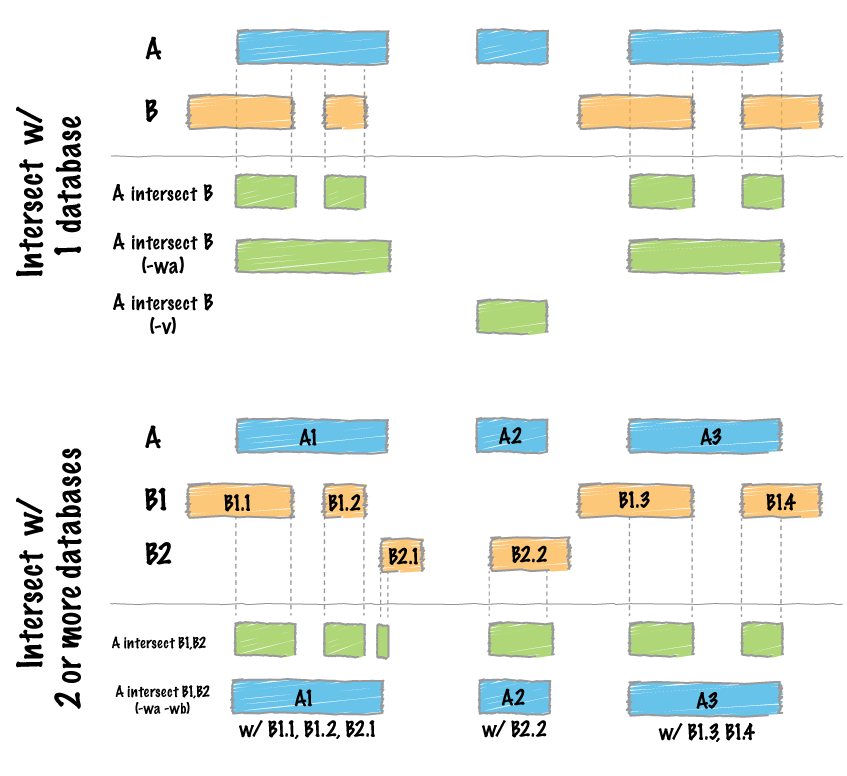
- 调用方法,这里主要介绍两种。bedtoolsr的函数跟bedtools的方法几乎是一一对应的。甚至函数名,参数名几乎都一样。因此,可以即便是新手,也可以快速入手bedtoolsr。下面以intersect方法为例,讲解调用方法。
#调用方法一
bt.intersect(a = A.bed, b = B.bed)
#output
# V1 V2 V3
#1 chr1 20 35
#调用方法二
bedtoolsr::bt.intersect(a = A.bed, b = B.bed)
#output
# V1 V2 V3
#1 chr1 20 35
#intersect所有参数
# a,
# b,
# wa = NULL,
# wb = NULL,
# loj = NULL,
# wo = NULL,
# wao = NULL,
# u = NULL,
# c = NULL,
# C = NULL,
# v = NULL,
# ubam = NULL,
# s = NULL,
# S = NULL,
# f = NULL,
# F = NULL,
# r = NULL,
# e = NULL,
# split = NULL,
# g = NULL,
# nonamecheck = NULL,
# sorted = NULL,
# names = NULL,
# filenames = NULL,
# sortout = NULL,
# bed = NULL,
# header = NULL,
# nobuf = NULL,
# iobuf = NULL,
# output = NULL
- bedtoolsr中所有的函数
| 函数名 | 函数名 | 函数名 |
|---|---|---|
| bt.annotate | bt.bamtobed | bt.bamtofastq |
| bt.bed12tobed6 | bt.bedpetobam | bt.bedtobam |
| bt.closest | bt.cluster | bt.closest |
| bt.cluster | bt.complement | bt.coverage |
| bt.expand | bt.fisher | bt.flank |
| bt.genomecov | bt.getfasta | bt.groupby |
| bt.igv | bt.intersect | bt.jaccard |
| bt.links | bt.makewindows | bt.map |
| bt.maskfasta | bt.merge | bt.multicov |
| bt.multiinter | bt.nuc | bt.overlap |
| bt.pairtobed | bt.pairtopair | bt.random |
| bt.reldist | bt.sample | bt.shift |
| bt.shuffle | bt.slop | bt.sort |
| bt.spacing | bt.split | bt.subtract |
| bt.summary | bt.tag | bt.unionbedg |
| bt.window |

