机器学习sklearn(49): 特征工程(十六) 特征降维(七)降维算法PCA和SVD(二)PCA与SVD
0 PCA与SVD

1 降维究竟是怎样实现?
class sklearn.decomposition.PCA (n_components=None, copy=True, whiten=False, svd_solver=’auto’, tol=0.0,iterated_power=’auto’, random_state=None)
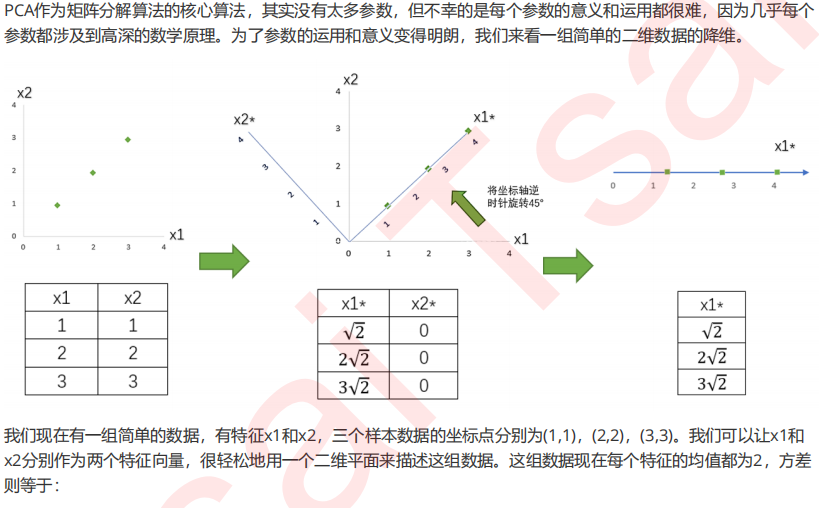







2 重要参数n_components

2.1 迷你案例:高维数据的可视化
1. 调用库和模块
import matplotlib.pyplot as plt from sklearn.datasets import load_iris from sklearn.decomposition import PCA
2. 提取数据集
iris = load_iris() y = iris.target X = iris.data #作为数组,X是几维? X.shape #作为数据表或特征矩阵,X是几维? import pandas as pd pd.DataFrame(X)
3. 建模
#调用PCA pca = PCA(n_components=2) #实例化 pca = pca.fit(X) #拟合模型 X_dr = pca.transform(X) #获取新矩阵 X_dr #也可以fit_transform一步到位 #X_dr = PCA(2).fit_transform(X)
4. 可视化
#要将三种鸢尾花的数据分布显示在二维平面坐标系中,对应的两个坐标(两个特征向量)应该是三种鸢尾花降维后的 x1和x2,怎样才能取出三种鸢尾花下不同的x1和x2呢? X_dr[y == 0, 0] #这里是布尔索引,看出来了么? #要展示三中分类的分布,需要对三种鸢尾花分别绘图 #可以写成三行代码,也可以写成for循环 """ plt.figure() plt.scatter(X_dr[y==0, 0], X_dr[y==0, 1], c="red", label=iris.target_names[0]) plt.scatter(X_dr[y==1, 0], X_dr[y==1, 1], c="black", label=iris.target_names[1]) plt.scatter(X_dr[y==2, 0], X_dr[y==2, 1], c="orange", label=iris.target_names[2]) plt.legend() plt.title('PCA of IRIS dataset') plt.show() """ colors = ['red', 'black', 'orange'] iris.target_names plt.figure() for i in [0, 1, 2]: plt.scatter(X_dr[y == i, 0] ,X_dr[y == i, 1] ,alpha=.7 ,c=colors[i] ,label=iris.target_names[i] ) plt.legend() plt.title('PCA of IRIS dataset') plt.show()
鸢尾花的分布被展现在我们眼前了,明显这是一个分簇的分布,并且每个簇之间的分布相对比较明显,也许versicolor和virginia这两种花之间会有一些分类错误,但setosa肯定不会被分错。这样的数据很容易分类,可以遇见,KNN,随机森林,神经网络,朴素贝叶斯,Adaboost这些分类器在鸢尾花数据集上,未调整的时候都可以有95%上下的准确率。
6. 探索降维后的数据
#属性explained_variance_,查看降维后每个新特征向量上所带的信息量大小(可解释性方差的大小) pca.explained_variance_ #属性explained_variance_ratio,查看降维后每个新特征向量所占的信息量占原始数据总信息量的百分比 #又叫做可解释方差贡献率 pca.explained_variance_ratio_ #大部分信息都被有效地集中在了第一个特征上 pca.explained_variance_ratio_.sum()
7. 选择最好的n_components:累积可解释方差贡献率曲线
当参数n_components中不填写任何值,则默认返回min(X.shape)个特征,一般来说,样本量都会大于特征数目,所以什么都不填就相当于转换了新特征空间,但没有减少特征的个数。一般来说,不会使用这种输入方式。但我们却可以使用这种输入方式来画出累计可解释方差贡献率曲线,以此选择最好的n_components的整数取值。
累积可解释方差贡献率曲线是一条以降维后保留的特征个数为横坐标,降维后新特征矩阵捕捉到的可解释方差贡献率为纵坐标的曲线,能够帮助我们决定n_components最好的取值。
import numpy as np pca_line = PCA().fit(X) plt.plot([1,2,3,4],np.cumsum(pca_line.explained_variance_ratio_)) plt.xticks([1,2,3,4]) #这是为了限制坐标轴显示为整数 plt.xlabel("number of components after dimension reduction") plt.ylabel("cumulative explained variance ratio") plt.show()
2.2 最大似然估计自选超参数
除了输入整数,n_components还有哪些选择呢?之前我们提到过,矩阵分解的理论发展在业界独树一帜,勤奋智慧的数学大神Minka, T.P.在麻省理工学院媒体实验室做研究时找出了让PCA用最大似然估计(maximum likelihood estimation)自选超参数的方法,输入“mle”作为n_components的参数输入,就可以调用这种方法
pca_mle = PCA(n_components="mle") pca_mle = pca_mle.fit(X) X_mle = pca_mle.transform(X) X_mle #可以发现,mle为我们自动选择了3个特征 pca_mle.explained_variance_ratio_.sum() #得到了比设定2个特征时更高的信息含量,对于鸢尾花这个很小的数据集来说,3个特征对应这么高的信息含量,并不 需要去纠结于只保留2个特征,毕竟三个特征也可以可视化
2.3 按信息量占比选超参数
输入[0,1]之间的浮点数,并且让参数svd_solver =='full',表示希望降维后的总解释性方差占比大于n_components指定的百分比,即是说,希望保留百分之多少的信息量。比如说,如果我们希望保留97%的信息量,就可以输入n_components = 0.97,PCA会自动选出能够让保留的信息量超过97%的特征数量。
pca_f = PCA(n_components=0.97,svd_solver="full") pca_f = pca_f.fit(X) X_f = pca_f.transform(X) pca_f.explained_variance_ratio_
本文来自博客园,作者:秋华,转载请注明原文链接:https://www.cnblogs.com/qiu-hua/p/14932959.html




【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】凌霞软件回馈社区,博客园 & 1Panel & Halo 联合会员上线
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】博客园社区专享云产品让利特惠,阿里云新客6.5折上折
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步