使用nnUNet跑BraTS脑肿瘤分割预测TC和ET非常低的原因。
使用nnUNet跑BraTS脑肿瘤分割预测TC和ET非常低,原来是预测的时候,使用了预处理后的标签。原本标签是:2:WT, 1:TC, 4:ET。但是预处理之后变为:1:WT, 2:TC, 3:ET。所以我们对于最后预测完需要变回原来的标签值。nnUNet需要改动代码位置:nnunet/inference/segmentation_export.py。代码只写了我如何修改的。
import torch
import numpy as np
one = torch.ones(9).reshape(1,3,3)
two = torch.ones(9).reshape(1,3,3) + 1
three = torch.ones(9).reshape(1,3,3) + 2
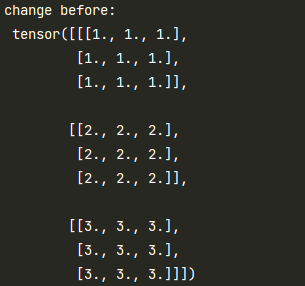
seg_old_size_postprocessed = torch.cat((one, two, three), dim=0)
#-----------------------需要添加的代码--------------------------------
h, w, t = seg_old_size_postprocessed.shape
pre = np.zeros((3, h, w, t), dtype=bool)
pre[0] = seg_old_size_postprocessed == 1
pre[1] = seg_old_size_postprocessed == 2
pre[2] = seg_old_size_postprocessed == 3
et = pre[2] # ET
net = np.logical_and(pre[1], np.logical_not(et)) # TC
ed = np.logical_and(pre[0], np.logical_not(pre[1])) # WT
labmap = np.zeros(seg_old_size_postprocessed.shape)
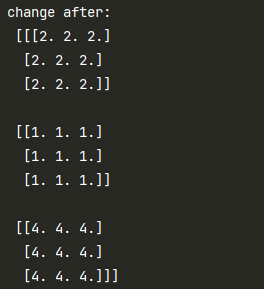
labmap[et] = 4
labmap[net] = 1
labmap[ed] = 2
#-----------------------需要添加的代码--------------------------------
print("change before:\n", seg_old_size_postprocessed)
print("change after:\n", labmap)
结果: