主成分析法-PCA
PCA
解决过拟合的三个方法:
- 提升数据量
- 正则化
- 降维
- 直接降维(直接选择某些重要的特征)
- 线性降维(PCA, MDS)
- 非线性降维(流形,Isomap,LLE)
数据维度过多会导致数据稀疏性增大,形成维度灾难
思想
数据从原来的坐标系转换到新的坐标系,新坐标系的选择是由数据本身决定的。第一个新坐标轴选择的是原始数据中方差最大的方向,第二个新坐标轴选择和第一个坐标轴正交且具有最大方差的方向。该过程一直重复,重复次数为原始数据中特征的数目。我们会发现,大部分方差都包含在最前面的几个新坐标轴中。因此,我们可以忽略余下的坐标轴,即对数据进行降维处理。
- 一个中心:原始特征空间的重构
找到一组正交基,将一组线性相关特征变换到线性无关
-
两个基本点
-
最大投影方差
使得往某个方向投影后的点尽可能分散,也就是让投影后的点的方差尽可能的大。
\[假设数据集:data = \{x_1, x_2, ... , x_n\}, x_i \in R^p\\ 数据的中心:\bar{x}_{p×1} = \frac{1}{N}\sum_{i = 1}^N x_i = \frac{1}{N}X^T1_N \\ 1_N = (1, 1, ... , 1)^T_{N×1}\\ 数据的方差:S_{p×p} = \frac{1}{N} \sum_{i = 1}^{N}(x_i - \bar{x})(x_i - \bar{x})^T = \frac{1}{N}X^THX\\ H = I_N-\frac{1}{N}1_N1_N^T \]那么投影后的点:
 \[s_{projection} = \cos \theta*x_i\\ \because u_1 * x_i = |u_1||x_i|\cos \theta \\ 设\quad |u_1| = 1\\ \therefore u_1 * x_i = |x_i|\cos \theta \\ \therefore s_{projection}= u_1 * x_i \\ \]
\[s_{projection} = \cos \theta*x_i\\ \because u_1 * x_i = |u_1||x_i|\cos \theta \\ 设\quad |u_1| = 1\\ \therefore u_1 * x_i = |x_i|\cos \theta \\ \therefore s_{projection}= u_1 * x_i \\ \]由于要求的是最大投影方差
\[中心化后的数据: x_i - \bar{x} \\ 投影后的点:(x_i - \bar{x})u_1 \\ 投影方差:((x_i - \bar{x})u_1)*((x_i - \bar{x})u_1)^T =((x_i - \bar{x})u_1)^2 \\ 设: J(u_1) = \frac{1}{N}\sum_{i = 1}^{N}((x_i - \bar{x})u_1)*((x_i - \bar{x})u_1)^T\quad s.t. u_1^Tu_1 = 1 \\ J(u_1) = \frac{1}{N}u_1^T(\sum_{i = 1}^{N}(x_i - \bar{x})(x_i - \bar{x})^T)u_1\\ J(u_1) = u_1^T(\frac{1}{N}\sum_{i = 1}^{N}(x_i - \bar{x})(x_i - \bar{x})^T)u_1 \\ J(u_1) = u_1^TS u_1 \]那么该问题变成了带有约束的优化问题:
\[\begin{cases} \hat{u_1} = argmax_{u_1}(u_1^TS u_1)\\ s.t. \quad u_1^Tu_1 = 1 \end{cases} \]用拉格朗日乘子法进行求解:
\[L(u_1,\lambda) = {u_1}(u_1^TS u_1) + \lambda(1 - u_1^Tu_1) \\ 对该函数进行求导:\\ \frac{\partial L}{\partial u_1} = 2Su_1 - 2 \lambda u_1 \\ 令该函数等于 0 \\ 2Su_1 = 2 \lambda u_1 \\ Su_1 = \lambda u_1 \]此时,\(S\) 就为方差矩阵,\(u_1\) 为特征值, \(\lambda\) 为特征向量
\[\hat{u_1} = argmax_{u_1} u_1^TSu_1 = argmax_{u_1} u_1^T\lambda u_1 argmax_{u_1} \lambda u_1^T u_1 = argmax_{u_1} \lambda \]找出投影方差最大的 \(q\) 个维度。
实际上 PCA 就是找出特征向量中最大的 \(q\) 个维度进行降维
-
最小重构代价
使新的坐标与原坐标之间的差值尽可能小。
\[中心化:\widetilde{x} = x_i - \bar{x} \\ 令 ||u_i|| = 1 \quad i = 1,2,...,p \\ 重构向量 x_i' = (\widetilde{x}^Tu_1)u_1 + (\widetilde{x}^Tu_2)u_2 \\ 标量:(\widetilde{x}^Tu_1) \quad 方向向量: u_1 \\ \widetilde{x} \in R^p \quad x_i' = \sum_{k = 1}^P(\widetilde{x}u_k)u_k \\ 降维后(丢掉一些信息):\hat x_i \in R^q(q < p) \quad \hat{x} = \sum_{k = 1}^q(\widetilde{x}u_k)u_k \\ 重构距离: \\ J = \frac{1}{N} \sum_{i = 1}^{N} ||x_i' - \hat{x}||^2\\ =\frac{1}{N} \sum_{i = 1}^{N} \sum_{i = q + 1}^P (\widetilde{x}^Tu_k) ^2\\ = \frac{1}{N} \sum_{i = 1}^{N} \sum_{i = q + 1}^P ((x_i - \bar{x})^Tu_k )^2 \\ \because \sum_{i = 1} ^ N \frac{1}{N} (({x_i - \bar{x}})u_k)^2 = u_k^TSu_k\\ \sum_{k = q + 1}^pu_k^TSu_k (s.t. \quad u_k^Tu_k = 1) \]此时又变成了一个带有约束的问题:
\[\begin{cases} u_k = argmin_{u_k}\sum_{k = q + 1}^pu_k^T Su_k \\ s.t. \quad u_k^Tu_k = 1 \end{cases} \]利用拉格朗日乘子法同样可得:
\[Su_k = \lambda u_k \\ u_k = argmin_{u_k}\sum_{k= q + 1}^{p} u_k^TSu_k = argmin_{u_k}\sum_{k= q + 1}^{p} \lambda_k \]找出重构距离影响最小的(p-q) 个维度舍弃。
-
PCA 算法流程
- 对数据进行去中心化(归一化处理)
- 计算归一化后的数据集的协方差矩阵
- 计算协方差矩阵的特征值和特征向量
- 根据特征值保留最重要的 \(k\) 个特征向量
- 将 \(m*n\) 的数据集乘以 \(k\) 个 \(n\) 维的特征向量 \((n*k)\),得到最后的降维数据 \(m * k\)
CODE
import numpy as np
from sklearn.datasets import load_iris
import matplotlib.pyplot as plt
def pca(data, n_dim):
# |data| = m * n
# 归一化(中心化、去平均值) data - mean(data)
data = data - np.mean(data, axis = 0, keepdims = True)
"""
去中心化后,那么每个维度均值就是为0,投影 方差就是 data^2
这里数据维度为 4
那么协方差 cov(x,y,q,p) = [
[cov(x, x), cov(x, y), cov(x, q), cov(x, p)]
[cov(y, x), cov(y, y), cov(y, q), cov(y, p)]
[cov(q, x), cov(q, y), cov(q, q), cov(q, p)]
[cov(p, x), cov(p, y), cov(p, q), cov(p, p)]
]
"""
cov = np.dot(data.T, data)
# 求方阵解特征值 1 * n 和特征向量 n * n
eig_values, eig_vector = np.linalg.eig(cov)
# 按照特征值的大小进行排序,此处按照从小到大的值进行排序,并且返回前 n_dim 个数据的下标 |index| = n_dim
indexs_ = np.argsort(-eig_values)[:n_dim]
# 提取 index_ 列中的特征向量 n * |index| = n * n_dim
picked_eig_vector = eig_vector[:, indexs_]
# data_dim = m * n x n * n_dim = m * n_dim,就是数据降维
data_ndim = np.dot(data, picked_eig_vector)
return data_ndim
def highdim_pca(data, n_dim):
'''
when n_features(D) >> n_samples(N), highdim_pca is O(N^3)
:param data: (n_samples, n_features)
:param n_dim: target dimensions
:return: (n_samples, n_dim)
'''
N = data.shape[0]
data = data - np.mean(data, axis = 0, keepdims = True)
Ncov = np.dot(data, data.T)
Neig_values, Neig_vector = np.linalg.eig(Ncov)
indexs_ = np.argsort(-Neig_values)[:n_dim]
Npicked_eig_values = Neig_values[indexs_]
# print(Npicked_eig_values)
Npicked_eig_vector = Neig_vector[:, indexs_]
# print(Npicked_eig_vector.shape)
picked_eig_vector = np.dot(data.T, Npicked_eig_vector)
picked_eig_vector = picked_eig_vector/(N*Npicked_eig_values.reshape(-1, n_dim))**0.5
# print(picked_eig_vector.shape)
data_ndim = np.dot(data, picked_eig_vector)
return data_ndim
if __name__ == "__main__":
data = load_iris()
X = data.data
y = data.target
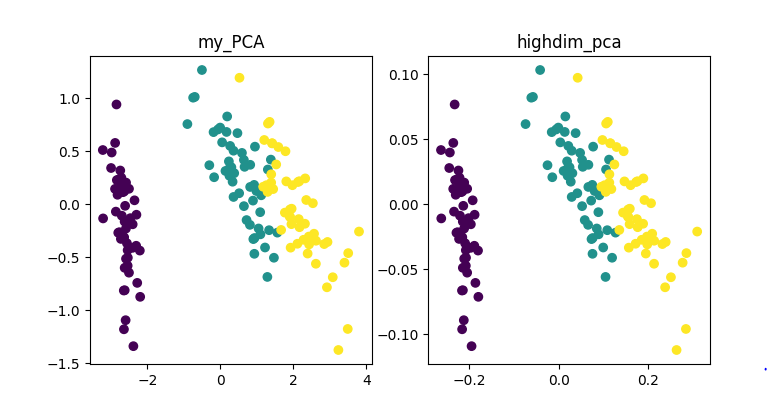
data_2d1 = pca(X, 2)
plt.figure(figsize=(8,4))
plt.subplot(121)
plt.title("my_PCA")
plt.scatter(data_2d1[:, 0], data_2d1[:, 1], c = y)
data_2d2 = data_2d1 = highdim_pca(X, 2)
plt.subplot(122)
plt.title("highdim_pca")
plt.scatter(data_2d2[:, 0], data_2d2[:, 1], c = y)
plt.show()
结果