ML聚类:k均值、学习向量量化LVQ、EM/高斯混合GMM、DBSCAN密度聚类、AGNES层次聚类、BIRCH、谱聚类原理推导及实现
目录
3.6 BIRCH层次方法的平衡迭代规约和聚类(Balanced Iterative Reducing and Clustering Using Hierarchies)
一下内容是《机器学习》周志华书籍第九章的个人理解
1.聚类概念
无监标记分类,数据集D={x1,x2,...xm},xi=(xi1,xi2,...,xin):如下图:相似据数聚成类

关键:如何描述数据(离散、连续)之间的差异?距离(差异大小)?多大范围内的距离可被认定成一个类?细分多少个类?
2.聚类结果的“好坏”评价指标
通常有2类指标:
1外部指标:参考外部的一些信息、数据;
2内部指标:仅仅利用自身数据得出结论;
2.1外部指标
需要引入外部评价模型,假设这个模型是无条件对的,现在拿这个模型来评价训练出的模型,以评估其好坏。

定义每个簇的伪标签:
![]()
从数据集D中任意取一对个(xi,xj)(i<j...(x3,x5)和(x5,x3)等价只取一个)做比较,有如下统计量关系:
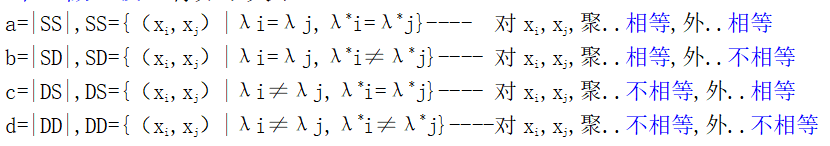
既,对于同一对数据(x3,x5),外部评价判为(1类,1类),内部评价均认为是(a类,a类),也就是说内外指标把x3,x5都归结到同一个类别,则记一个SS。这里1和a无关,两个评价体系有自己独立的类标签,只要在他们内部把这一对判为同一类就记为SS。
类似的,外部评价是同一类,内部评价为非同一类,就记一个SD。同样的DS,DD。
最后把这些标记的数量统计起来,a为所有记为SS的对的数量,......b,c,d。
共有m个数据,a+b+c+d=m(m-1)/2对评价。有如下常用的外部衡量指标关系:
Jaccard JC系数:
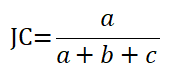
FM指数:
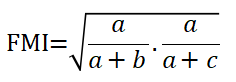
Rand指数:

所有这些计量数∈[0,1],越大越好。(因为假设外部评价是无条件对的,越大说明内部评价(训练出的模型)越接近外部评价)
2.2内部指标
定义:

有如下衡量指数:
DB指数:
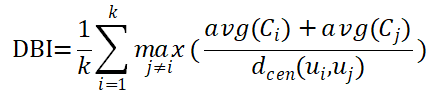
DI指数:
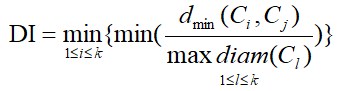
DBI越小越好,DI越大越好。
其它评估标准:
https://blog.csdn.net/sinat_26917383/article/details/70577710
不像监督学习的分类问题和回归问题,无监督聚类没有样本输出,也就没有比较直接的聚类评估方法。但是可以从簇内的稠密程度和簇间的离散程度来评估聚类的效果。常见的有:轮廓系数Silhouette Coefficient和Calinski-Harabasz Index。
1)Calinski-Harabasz分数值s越大则聚类效果越好:
其中m为训练集样本数,k为类别数。Bk为类别之间的协方差矩阵,Wk为类别内部数据的协方差矩阵。tr为矩阵的迹。也就是说,类别内部数据的协方差越小越好,类别之间的协方差越大越好,这样的Calinski-Harabasz分数会高。在scikit-learn中, Calinski-Harabasz Index对应的方法是metrics.calinski_harabaz_score.
2)轮廓系数silhouette(-1,1之间,值越大,聚类效果越好),聚类的质量也相对较好:
对于其中的一个点 i 来说:计算 a(i) = average(i向量到所有它属于的簇中其它点的距离)、计算 b(i) = min (i向量到各个非本身所在簇的所有点的平均距离),那么 i 向量轮廓系数就为:

2.3距离的计算
上面很多地方用到了距离的计算,样本属性中通常包含离散和连续的,距离计算分为连续有序、离散无序值的计算,这样就是计算样本之间的距离了。
连续、有序的属性:闵可夫斯基距离:
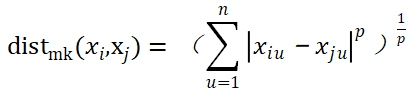 ,p=2欧氏距离,p=1曼哈顿距离;
,p=2欧氏距离,p=1曼哈顿距离;
离散、无序的属性:VDM(Value Difference Metric)
mu,a:u属性上取值为a的数量;mu,a,i:第i个簇中u属性上取值为a的数量;
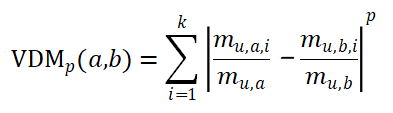
nc个连续与n-nc个离散属性的混合距离:
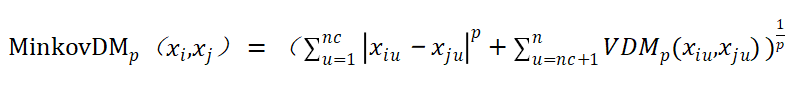
加权距离:
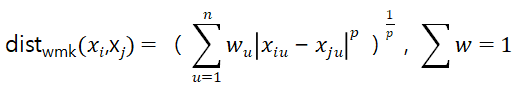
3聚类类算法
这里开始介绍聚类相关算法
3.1 k均值算法
数据集D={x1,x2,...xm};期望划分后的簇误差最小:
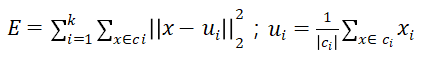


高级扩展:k均值仅在凸形簇结构上表现较好、核k均值、拉普拉斯映射降维后执行k均值
elkan K-Means:利用了两边之和大于等于第三边,以及两边之差小于第三边的三角形性质,来减少KMeans的距离的计算。
K-Means++:把原来初始时随机的选k个点作为初始簇中心做了优化。先随机取一个点作为簇中心,此时只有一个簇,然后计算所有点与簇中心点的距离,距离越大的概率越大,这个概率用来选择第二个簇的中心点,此时有两个簇;然后计算所有点与两个簇中心的距离,取2个距离中小的那个作为样本的计算距离,样本的计算距离越大的概率越大,用于选第3个簇中心,得到3个簇;然后计算所有样本点与3个簇中心的距离,取3个距离中小的那个作为样本的计算距离,......直到得到k个簇中心,作为原始kmeans的随机初始化的替换。
Mini Batch K-Means:在统的K-Means算法中,要计算所有的样本点到所有的质心的距离。如果样本量非常大,比如达到10万以上,特征有100以上,此时用传统的K-Means算法非常的耗时;Mini Batch,用样本集中的一部分的样本来做传统的K-Means,这样可以避免样本量太大时的计算难题,算法收敛速度大大加快。当然此时的代价就是我们的聚类的精确度也会有一些降低。一般来说这个降低的幅度在可以接受的范围之内。
Mini Batch K-Means中,我们会选择一个合适的批样本大小batch size,来做K-Means聚类。一般是通过无放回的随机采样得到的。
3.1.2 实现
scikit-learn中,包括两个K-Means的算法:传统的KMeans、基于采样的MiniBatchKMeans。
KMeans:
KMeans类的主要参数有:
1) n_clusters: 即我们的聚类数量k。
2)max_iter: 最大的迭代次数。
3)n_init:用不同的质心初始化算法的次数。由于K-Means是结果受初始值影响的局部最优的迭代算法,因此需要多跑几次,默认是10。如果k值较大,可适当增大。
4)init: 即初始值选择的方式,完全随机选择'random',优化过的'k-means++'或者自己指定初始化的k个质心。一般建议使用默认的'k-means++'。
5)algorithm:有“auto”, “full” or “elkan”三种选择。"full"是传统的K-Means算法, “elkan”是elkan K-Means算法。默认的"auto"会根据数据值是否是稀疏的,来决定如何选择"full"和“elkan”。一般数据是稠密的,那么就是 “elkan”,否则就是"full"。一般来说建议直接用默认的"auto"import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets.samples_generator import make_blobs
from sklearn.cluster import KMeans
from sklearn import metrics
# X为样本特征,Y为样本簇类别, 共1000个样本,每个样本5个特征,共4个簇(类),
# 簇中心在[-1,-1], [0,0],[1,1], [2,2], 簇方差分别为[0.4, 0.2, 0.2,0.2]
X, y = make_blobs(n_samples=1000, n_features=2, centers=[[-1, -1], [0, 0], [1, 1], [2, 2]],
cluster_std=[0.4, 0.2, 0.2, 0.2],
random_state=9)
plt.scatter(X[:, 0], X[:, 1], marker='o')
y_pred = KMeans(n_clusters=4, random_state=9).fit_predict(X)
print('Calinski-Harabasz分数:', metrics.calinski_harabaz_score(X, y_pred))
print('silhouette_score轮廓系数:', metrics.silhouette_score(X, y_pred))
plt.scatter(X[:, 0], X[:, 1], c=y_pred)
plt.show()
#
Calinski-Harabasz分数: 5924.050613480169
silhouette_score轮廓系数: 0.6634549555891298
基于采样的MiniBatchKMeans
MiniBatchKMeans类主要参数
MiniBatchKMeans类的主要参数比KMeans类稍多,主要有:
1) n_clusters:一样。
2)max_iter:一样。
3)n_init:用不同的初始化质心运行算法的次数,和KMeans类意义稍有不同的是,MiniBatchKMeans每次用不同的采样数据集来跑不同的初始化质心运行算法。
4)batch_size:算法采样集的大小,默认是100.如果数据集的类别较多或者噪音点较多,需要增加这个值。
5)init: 一样。
6)init_size: 做质心初始值候选的样本个数,默认是batch_size的3倍,一般用默认值就可以了。
7)reassignment_ratio: 某个类别质心被重新赋值的最大次数比例,这个和max_iter一样是为了控制算法运行时间的。这个比例是占样本总数的比例,乘以样本总数就得到了每个类别质心可以重新赋值的次数。如果取值较高的话算法收敛时间可能会增加,尤其是那些暂时拥有样本数较少的质心。默认是0.01。如果数据量不是超大的话,比如1w以下,建议使用默认值。如果数据量超过1w,类别又比较多,可能需要适当减少这个比例值。具体要根据训练集来决定。
8)max_no_improvement:即连续多少个Mini Batch没有改善聚类效果的话,就停止算法, 和reassignment_ratio, max_iter一样是为了控制算法运行时间的。默认是10.一般用默认值就足够了。import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets.samples_generator import make_blobs
from sklearn.cluster import MiniBatchKMeans
from sklearn import metrics
# X为样本特征,Y为样本簇类别, 共1000个样本,每个样本5个特征,共4个簇(类),
# 簇中心在[-1,-1], [0,0],[1,1], [2,2], 簇方差分别为[0.4, 0.2, 0.2,0.2]
X, y = make_blobs(n_samples=1000, n_features=2, centers=[[-1, -1], [0, 0], [1, 1], [2, 2]],
cluster_std=[0.4, 0.2, 0.2, 0.2],
random_state=9)
plt.scatter(X[:, 0], X[:, 1], marker='o')
y_pred = MiniBatchKMeans(n_clusters=4, batch_size=200, random_state=9).fit_predict(X)
print('Calinski-Harabasz分数:', metrics.calinski_harabaz_score(X, y_pred))
print('silhouette_score轮廓系数:', metrics.silhouette_score(X, y_pred))
plt.scatter(X[:, 0], X[:, 1], c=y_pred)
plt.show()
#
Calinski-Harabasz分数: 2827.388413921534
silhouette_score轮廓系数: 0.5349102143157655
3.2 LVQ学习向量量化算法
前提:假设样本带有标记类型(也可事先通过聚类等算法学到类型):D={(x1,y1),(x2,y2),...(xm,ym)}

学好之后的模型向量{p1,p2,...,pk}后,可定义一个样本空间X:pi对应其中一个区域Ri,这个区域内的点离pi最近
![]()

扩展:提高收敛速度的改进LVQ2、LVQ3
这个算法没有库,需要自己手动实现,感觉理论性不是很强,
3.3 高斯混合GMM
1)与其他聚类算法k-means、LVQ不同的是,以往的聚类分类都是具体的,要么必须是a类,要么必须是b类…;
2)高斯混合聚类GMM给出的是一个概率:样本xj有0.2的概率是男,0.8的概率是女;……..。
3)假设:n维度向量x,假设服从高斯分布,是这个样子:
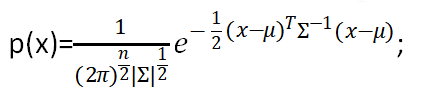
4)问题:GMM的问题是,假如现在我们有一堆数据,知道这一堆是男性,这一堆是女性了,显然我们就可以轻松的计算方差、均值得出高斯分布模型了。但是现在并不知道类别标签。
5)EM算法给出解法。具体GMM的求解在EM之后。也可以直接跳过EM。
3.3.1EM算法
1)EM算法解决这个的思路是先全部假设出来。想象一下平均分米,先随便分,然后多的给少的划入一些,直到大家都感觉差不多了,那其实真就差不了多少了,不信去称一下,重量几乎差不多的。EM就是这么个思路。
2)每个样本都不知道是从哪个分布抽取的,这个时候,对于每一个样本,就有两个东西需要猜测或者估计:
每一个样本属于哪个分布。(是男还是女)、每一个分布对应的参数。(男女分布的均值、方差)
3)基本流程

例:
(a)E初始化参数:先初始化男簇身高的正态分布的参数:如均值=1.7,方差=0.1,女性同样。
(b)E:(有了(a)的参数,就能)计算每一样本更可能属于男生分布或者女生分布了(如男:0.55,女:0.45,于是标记为男);
(c)M:标为男的有n个样本,现在用这n个样本的数据来重新估计男生身高分布的参数(最大似然估计),女生分布也按照相同的方式估计出来,更新分布。
(d)(c)后,两个分布的参数可能变了,那每个样本的概率又可以从新算了(原来男:0.55,女:0.45更可能属于男的,结果女:0.65,男:0.35更可能是女了),然后重复步骤(1)至(3),直到参数不发生变化收敛为止。
(e)然后我们就神奇的认为,我们学到了这个数据的分布,每个样本的类别标记,并且能拿来预测新的未知样本属于男、女的概率。
4)具体的计算推导
基本流程知道后,核心就是怎么更新参数了,这个在任何一个介绍EM算法里面只会给出一个抽象的概念,因为分布有千万种,更新分布的参数只能抽象来讲。具体来讲会放大下一节高斯混合模型HMM来讲,因为此时分布就是高斯分布,要更新的就是均值和方差。
a)对于m个样本的数据集,如果我们知道m个样本的类型是
,显然,在假设出分布后(如服从高斯分布),概率论中常见的(对数)极大似然法,就能得到我们分布的参数了
然而,如果类型z是未知的,这里面的参数用极大似然是求不出的,因此EM出来解决这个问题,转为可以极大似然求解的迭代形式。
b)额外知识,先看这个Jensen原理:https://blog.csdn.net/jiang425776024/article/details/87992127
Jensen不等式的表述是,如果f(x)是凸函数,x是随机变量,则下面不等式成立:

如果是凹函数:

E是数学期望,对于离散型随机变量,数学期望是求和,对连续型随机变量则为求定积分。如果f(x)是一个严格凹/凸函数,当且仅当x是常数时不等式取等号:

c)假设样本xi为类型zi(隐变量)的概率分布为Qi(什么意思?就是概率的概率分布,xi属于类别zi的概率的概率分布),显然根据分布,必须有这些条件成立:
![]()
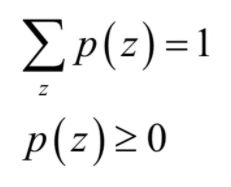
d)利用b)的知识,和c)的概率的概率分布假设,就能对a)中的极大似然目标做转换了,记:
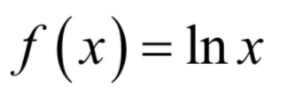
根据b),因为f(x)=lnx为凹函数,所以:

有(注意,f(x)=lnx中的x,被强行看做是p/Qi的期望,Qi为p/Qi的概率;因为累加Qi*(p/Qi)很像是求期望的形式):


所以最终变为:

显然,这个下界函数更容易求极值,因为对数函数里面已经没有求和项,对参数求导并令导数为0时一般可以得到公式解。去掉上式中为常数的部分,我们要做到,就是不断提高这个下界(极大似然):

e)EM算法流程
首先初始化参数θ的值(当然不是随机的,要满足概率和为1),接下来循环迭代直至收敛,每次迭代时分为两步:
E步,基于当前的参数(第一次就是基于随机的参数,后面的就是基于计算出来的参数了)估计x的隐变量z的条件概率:(有了θ就能估计属于男、女类的概率)
![]()
M步,基于E步参数极大化下界函数:(有了E计算的概率,取概率最大的为样本的类标记,于是就确定了所有类标记,就能用常规的极大似然法了。比如一堆数据男女类别都标好了,假设服从高斯分布,还算不出这个模型的参数了?现在当然能算了,这个M步argmax过程就是这么个极大似然过程。显然很抽象,结合到GMM的时候看到会详细点)
3.3.2 GMM中参数的求解
1)N(均值,方差)为高斯分布,假设有k个类型,wj为第j个分布的权重(j=1,..,k;∑wj=1),则GMM中的概率的概率分布Qi可求(上EM步骤那样,先随机假设出参数。如,分布权重:多少概率属于男,属于女;分布参数:方差、均值)
记Qi = qij,表示样本 xi 属于类 zj 的混合概率(由男女混合分布加成得到,这个概率作为最终类别概率):

例:

2)极大似然。通过1)我们已经可以知道每个样本的类别标记了(如上例,男:女=0.5625:0.4375,当前样本标记设为男。)
![]()
所以可以执行EM的M步极大似然了。展开原式子,替换成高斯分布的形式,方便极大似然的求导:

3)对均值uj,j=1,...,k求导,得到u的更新形式:

以下推导需要两个知识:
矩阵求导:https://blog.csdn.net/jiang425776024/article/details/87388015
多元高斯模型求导:https://blog.csdn.net/SZU_Hadooper/article/details/78090348
4)同理,∑的更新形式可求:

5)最后是wj,j=1,..,k的更新:
根据约束:
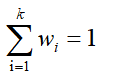
可以构造拉格朗日形式,因为L式子中其它两个不含有w,所以两个∑∑里面直接写成了:
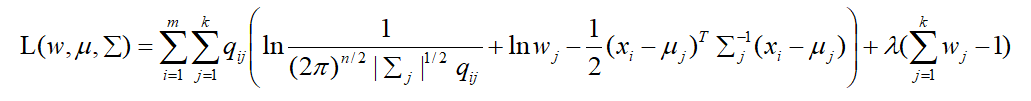
求导:

(纯原创,公式推得很辛苦,麻烦转载刷刷流量,就这点需求了)
6)最终,高斯混合模型的更新方式已全部得出,可以直接按照结论进行更新(第一次随机假设出的那些)参数了。
1.首先初始化μ,∑,w
2.E步,根据模型参数的当前估计值,计算第i个样本来自第j个高斯分布的概率:

3.M步,用极大似然推导公式进行μ,∑,w参数更新:

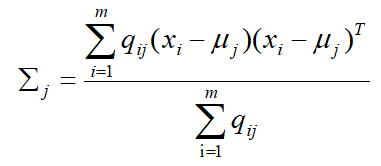

4.参数μ,∑,w几乎不再变化时,样本属于各个类的概率可求,其中最终标记可设为概率最大对应的类。
![]()
sklearn Gaussian mixture models API:https://scikit-learn.org/stable/modules/mixture.html
import numpy as np
import matplotlib.pyplot as plt
from matplotlib.colors import LogNorm
from sklearn import mixture
n_samples = 300
n_features = 2
# generate random sample, two components
np.random.seed(0)
# 数据1:generate spherical球形 data centered on (20, 20)
shifted_gaussian = np.random.randn(n_samples, n_features) + np.array([20, 20])
# 数据2:生成零中心拉伸高斯数据,C为矩阵变换用于拉伸数据,中心(0,0)
C = np.array([[0., -0.7], [3.5, .7]])
stretched_gaussian = np.dot(np.random.randn(n_samples, n_features), C)
# 合并数据,竖直
X_train = np.vstack([shifted_gaussian, stretched_gaussian])
'''
fit a Gaussian Mixture Model with two components
covariance_type={‘full’ (default), ‘tied’, ‘diag’, ‘spherical’}控制这每个簇的形状自由度。
默认设置是convariance_type=’diag’,意思是簇在每个维度的尺寸都可以单独设置,但椭圆边界的主轴要与坐标轴平行。
covariance_type=’spherical’时模型通过约束簇的形状,让所有维度相等。这样得到的聚类结果和k-means聚类的特征是相似的,虽然两者并不完全相同。
covariance_type=’full’时,该模型允许每个簇在任意方向上用椭圆建模。
'''
clf = mixture.GaussianMixture(n_components=2, covariance_type='full')
clf.fit(X_train)
plt.scatter(X_train[:, 0], X_train[:, 1], 0.5)
x = np.linspace(-20., 30.)
y = np.linspace(-20., 30.)
X, Y = np.meshgrid(x, y)
XX = np.array([X.ravel(), Y.ravel()]).T
# 预测簇类别
pz = clf.predict(XX).reshape(X.shape)
# 簇概率
prbz = clf.predict_proba(XX).round(5)
print('簇的中点', clf.means_)
print('簇的协方差', clf.covariances_)
# 计算每个样本的加权对数概率。
Z = clf.score_samples(XX)
Z = -Z.reshape(X.shape)
CS = plt.contour(X, Y, Z, norm=LogNorm(vmin=1.0, vmax=1000.0),
levels=np.logspace(0, 3, 10))
CB = plt.colorbar(CS, shrink=0.8, extend='both')
plt.title('Negative log-likelihood predicted by a GMM')
plt.axis('tight')
plt.show()
#
簇的中点 [[19.91453549 19.97556345]
[-0.13607006 -0.07059606]]
3.4 DBSCAN 密度聚类
DBSCAN是一种基于密度的聚类算法,此类算法假设聚类结构能通过样本分布的紧密深度确定,从样本的密度角度来考量样本之间的可连接性,基于可连接样本不断扩张聚类簇以获得最终聚类结果。
1)定义

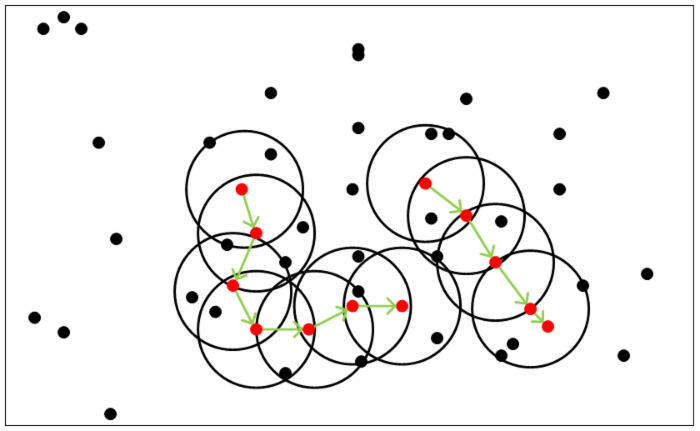
2)DBSCAN算法流程


在scikit-learn中,DBSCAN算法类为sklearn.cluster.DBSCAN
sklearn API:https://scikit-learn.org/stable/modules/generated/sklearn.cluster.DBSCAN.html#sklearn.cluster.DBSCAN
参考中文参数介绍:https://www.cnblogs.com/pinard/p/6217852.html
eps:ϵ-邻域的距离阈值,认值是0.5。
min_samples: 核心对象的ϵ-邻域的样本数阈值。默认值是5。min_samples过大,则核心对象会过少,此时簇内部分本来是一类的样本可能会被标为噪音点,类别数也会变多。反之min_samples过小的话,则会产生大量的核心对象,可能会导致类别数过少。
metric:距离度量参数:欧式距离 “euclidean”、曼哈顿距离 “manhattan”、切比雪夫距离“chebyshev”、闵可夫斯基距离 “minkowski”、带权重闵可夫斯基距离 “wminkowski”、标准化欧式距离 “seuclidean”: 即对于各特征维度做了归一化以后的欧式距离。此时各样本特征维度的均值为0,方差为1、马氏距离“mahalanobis”。
algorithm:最近邻搜索算法参数,‘brute’对应第一种蛮力实现,‘kd_tree’对应第二种KD树实现,‘ball_tree’对应第三种的球树实现, ‘auto’则会在上面三种算法中做权衡,选择一个拟合最好的最优算法
leaf_size:最近邻搜索算法参数,为使用KD树或者球树时, 停止建子树的叶子节点数量的阈值。这个值越小,则生成的KD树或者球树就越大,层数越深,建树时间越长,反之,则生成的KD树或者球树会小,层数较浅,建树时间较短。默认是30. 因为这个值一般只影响算法的运行速度和使用内存大小,因此一般情况下可以不管它。
p: 最近邻距离度量参数。只用于闵可夫斯基距离和带权重闵可夫斯基距离中p值的选择,p=1为曼哈顿距离, p=2为欧式距离。如果使用默认的欧式距离不需要管这个参数。
import numpy as np
from sklearn.cluster import DBSCAN
from sklearn import metrics
from sklearn.datasets.samples_generator import make_blobs
from sklearn.preprocessing import StandardScaler
# #############################################################################
# Generate sample data
centers = [[1, 1], [-1, -1], [1, -1]]
X, labels_true = make_blobs(n_samples=750, centers=centers, cluster_std=0.4,
random_state=0)
# 标准化
X = StandardScaler().fit_transform(X)
# #############################################################################
# 0.3邻域、10核心对象阈值
db = DBSCAN(eps=0.3, min_samples=10).fit(X)
# 全false
core_samples_mask = np.zeros_like(db.labels_, dtype=bool)
# 核心样本位置为True
core_samples_mask[db.core_sample_indices_] = True
labels = db.labels_
# 计算簇标签, 忽略噪点(label=-1)
n_clusters_ = np.unique(labels).shape[0] - (1 if -1 in labels else 0)
# 计算噪点数量
n_noise_ = labels[labels == -1].shape[0]
print('簇数量: %d' % n_clusters_)
print('噪点数量: %d' % n_noise_)
# #########################衡量指标####################################################
# reference:https://blog.csdn.net/sinat_26917383/article/details/70577710
# homogeneity_score(labels_true, labels_pred)集群标签的同质性度量,得分在0.0到1.0之间。1.0代表完全均匀的标签。
print("Homogeneity: %0.3f" % metrics.homogeneity_score(labels_true, labels))
# 集群标签的完整性度量。如果所有数据点都是同一簇的元素,则聚类结果满足完整性。得分在0.0到1.0之间。1.0代表完美的标签
print("Completeness: %0.3f" % metrics.completeness_score(labels_true, labels))
# 同质性和完整性的调和平均
print("V-measure: %0.3f" % metrics.v_measure_score(labels_true, labels))
# 兰德系数,[-1,1]越大意味着与真实情况越吻合
print("Adjusted Rand Index: %0.3f"
% metrics.adjusted_rand_score(labels_true, labels))
# 互信息,也是衡量吻合度,[-1,1]
print("Adjusted Mutual Information: %0.3f"
% metrics.adjusted_mutual_info_score(labels_true, labels))
# 轮廓系数,适用于实际类型未知情况,[-1,1]同类别样本越近,不同类样本距离越远分数越高
print("Silhouette Coefficient: %0.3f"
% metrics.silhouette_score(X, labels))
# ################################绘图#############################################
# Plot result
import matplotlib.pyplot as plt
# Black removed and is used for noise instead.
unique_labels = set(labels)
colors = [plt.cm.Spectral(each)
for each in np.linspace(0, 1, len(unique_labels))]
# 遍历类别、颜色列表,给不同的簇类画上不同的颜色
for k, col in zip(unique_labels, colors):
# 噪点用黑色
if k == -1:
col = [0, 0, 0, 1]
# 类别为k的bool矩阵
class_member_mask = (labels == k)
# 取类别为k,且core_samples_mask中为True(核心样本位置为True)的位置的样本
# 既,核心样本markersize=14,比非核心样本大
xy = X[class_member_mask & core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),
markeredgecolor='k', markersize=14)
# ~ 取反:把1变为0,把0变为1
# 取类别为k,且core_samples_mask中为False(非核心样本位置为False)的位置的样本
# 既,非核心样本markersize=6,比核心样本小
xy = X[class_member_mask & ~core_samples_mask]
plt.plot(xy[:, 0], xy[:, 1], 'o', markerfacecolor=tuple(col),
markeredgecolor='k', markersize=6)
plt.title('Estimated number of clusters: %d' % n_clusters_)
plt.show()
output:
簇数量: 3
噪点数量: 18
Homogeneity: 0.953
Completeness: 0.883
V-measure: 0.917
Adjusted Rand Index: 0.952
Adjusted Mutual Information: 0.883
Silhouette Coefficient: 0.626

3.5 AGNES层次聚类
试图从不同层次对数据集进行划分,从而形成树形的聚类结构。自底向上的聚会策略,先将每个样本看做一个聚类簇(m个样本就是m个簇),然后每次运行找出距离最近的两个簇进行合并,不断重复,直到达到设定的k值。注意的是,簇的距离计算的定义:
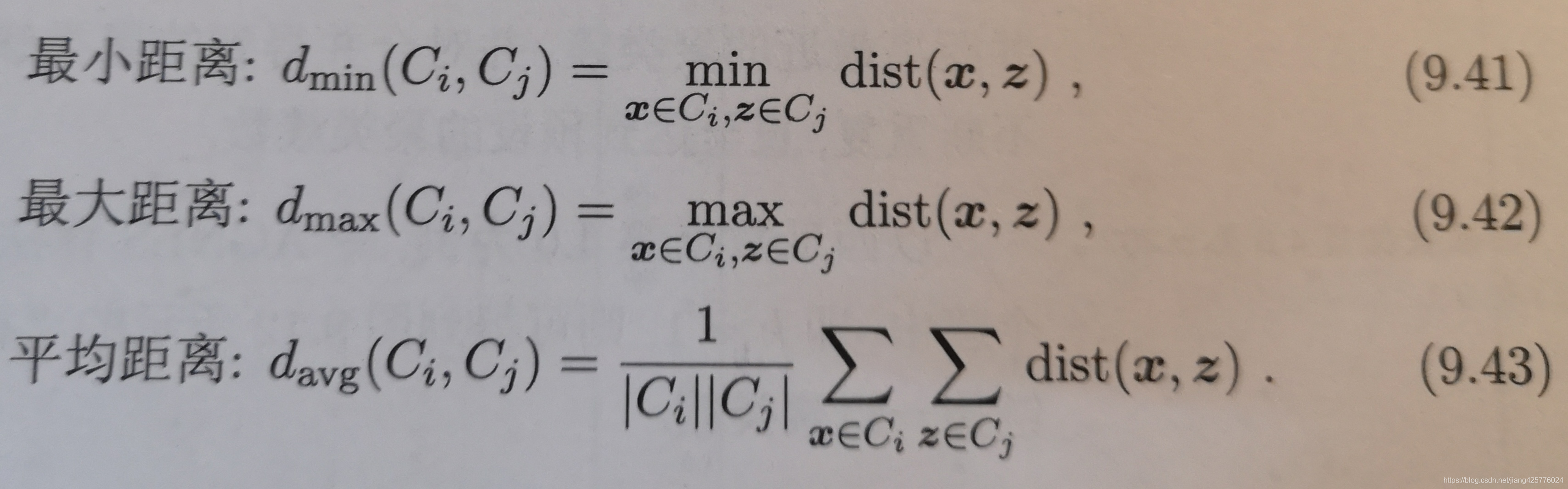
按照这3种min、max、avg计算距离,AGNES算法被称为“单链接”、“全链接”、“均链接”
算法流程(《机器学习》周志华,p215):
6,7行计算每个样本间相互距离;13-15合并 i* 和 j* 两号的簇并入 i* 内,j* 以后的簇全部更新编号;
17行,删除所有与 j* 号簇有关的距离信息;
19-20行,(只需要)更新第 i* 号簇与其他簇的距离;

图例:

sklearn中没有实现DBSCAN层次聚类的算法,不过这个算法实际操作简单,而且下面有一个更实用的层次聚类算法在sklearn中有实现,因此这个可以看看作为思想,通常更实用的还是下面的Brich算法。
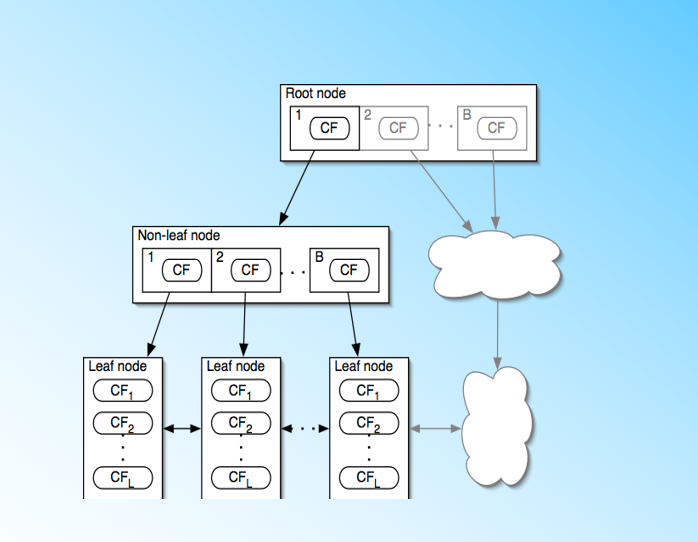
3.6 BIRCH层次方法的平衡迭代规约和聚类(Balanced Iterative Reducing and Clustering Using Hierarchies)
sklearn API:https://scikit-learn.org/stable/modules/generated/sklearn.cluster.Birch.html#sklearn.cluster.Birch
原理介绍:https://www.cnblogs.com/pinard/p/6179132.html
使用介绍:https://www.cnblogs.com/pinard/p/6200579.html
BIRCH算法利用了一个树结构来帮助我们快速的聚类,这个数结构类似于平衡B+树。
优点:速度快、比较适合于数据量大,类别数K也比较多的情况。
3.7 谱聚类(spectral clustering)
原理介绍:https://www.cnblogs.com/pinard/p/6221564.html
使用:https://www.cnblogs.com/pinard/p/6235920.html
谱聚类是从图论中演化出来的算法,后来在聚类中得到了广泛的应用。它的主要思想是把所有的数据看做空间中的点,这些点之间可以用边连接起来。距离较远的两个点之间的边权重值较低,而距离较近的两个点之间的边权重值较高,通过对所有数据点组成的图进行切图,让切图后不同的子图间边权重和尽可能的低,而子图内的边权重和尽可能的高,从而达到聚类的目的。
优缺点:谱聚类只需要数据之间的相似度矩阵,因此对于处理稀疏数据的聚类很有效。由于使用了降维,因此在处理高维数据聚类时的复杂度比传统聚类算法好。聚类的维度非常高,则可能由于降维的幅度不够,谱聚类的运行速度和最后的聚类效果均不好。



 浙公网安备 33010602011771号
浙公网安备 33010602011771号