MICCAI2018-BraTSfirst-3D MRI brain tumor segmentation using autoencoder regularization
前言
这是MICCAI2018发表的论文,并且在BraTS2018脑肿瘤分割比赛中取得第一名。
Abstract
- based on encoder-decoder architecture
- a variational auto-encoder branch is added to reconstruct the input image
1 Introduction
- BraTS 2018 training dataset:285 cases(210 HGG and 75LGG)
HGG
HGG :高级别胶质瘤(WHO3~4级)为低分化胶质瘤;这类肿瘤为恶性肿瘤,患者预后较差。LGG :低级别胶质瘤(WHO1~2级)为分化良好的胶质瘤;虽然这类肿瘤在生物上并不属于良性肿瘤,但是患者的预后相对较好。 - 每一个cases都有四种 3D MRI 模态(T1,T1c,T2,and FLAIR)
在医学界把像 T1、T1c、T2、flair这样的称为序列,一个病例的可以有多个序列,每个序列由许多切片组成, 此外,获得每种序列的方式不同,例如T1、T2是由于测量电磁波的物理量不同而产生的两种不同的序列,再例如T1c序列要在做MRI之前往血液打造影剂.FLAIR序列是核磁共振(MR)的一种常用的序列,全称是液体衰减反转回复序列,也称为水抑制成像技术. 通俗地说,它是压水像。在该序列上,脑脊液呈现低信号(暗一些),实质性病灶和含有结合水的病灶显示为明显的高信号(亮一些) - The input image size is 240x240x155.
- The data were collected from 19 institutions, using various MRI scanners.
- 标注包括三个区域:whole tumor (WT), tumor core (TC) and enhancing tumor(ET)
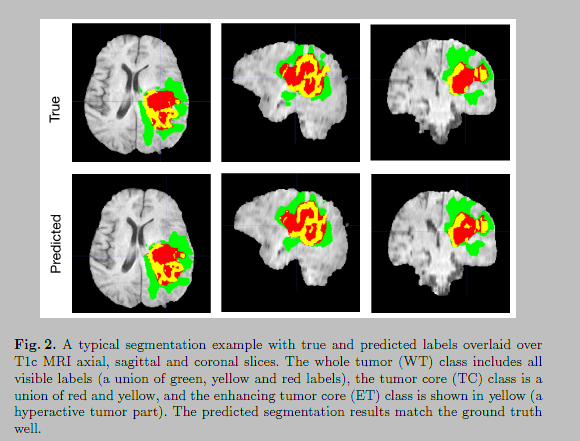
- 还有两个额外数据集:验证(66 cases) 和 测试(191 cases),但是没有标注
- 验证数据集可以一直测试提交代码,测试数据集只测试一次提交代码作为最终成绩
- 网络结构: 遵循CNN的编码解码结构,可以直接输出三个区域而不是使用几个网络,
在训练过程中,增加一个额外的分支去正则化共享编码器
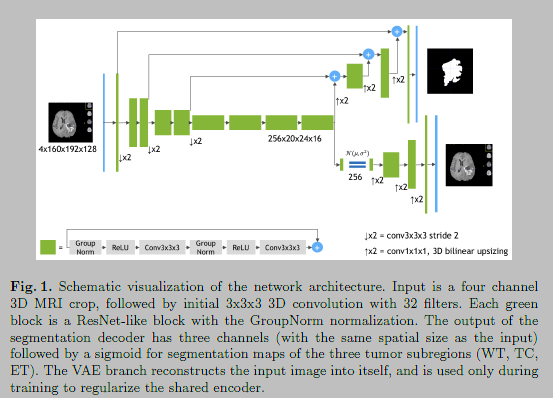
2 Related work
- BraTS2017first EMMA 结合了DeepMedic、FCN、U-net
- BraTS2018Second Isensee U-net进行小的优化可以实现很不错的效果,可以使用自己机构的额外数据集进行训练


