使用并行计算
正如我们在上一节提到的,R 是被设计成单线程的,但它仍然可以使用多线程并行计
算,即运行多个 R 会话进行计算。该技术是由一个随 R 一起发布的并行库来支持的。
假设我们需要做一次模拟:生成一个遵循特定随机过程的随机路径,看看在任何一点
是否有值超出了起点的一个固定邻域。
以下代码是一种实现:
set.seed(1)
sim_data <- 100 * cumprod(1 + rnorm(500, 0, 0.006))
plot(sim_data, type = "s", ylim = c(85, 115),
main = "A simulated random path")
abline(h = 100, lty = 2, col = "blue")
448 第 13 章 高性能计算
abline(h = 100 * (1 + 0.1 * c(1, -1)), lty = 3, col = "red")
生成的结果如图 13-6 所示。
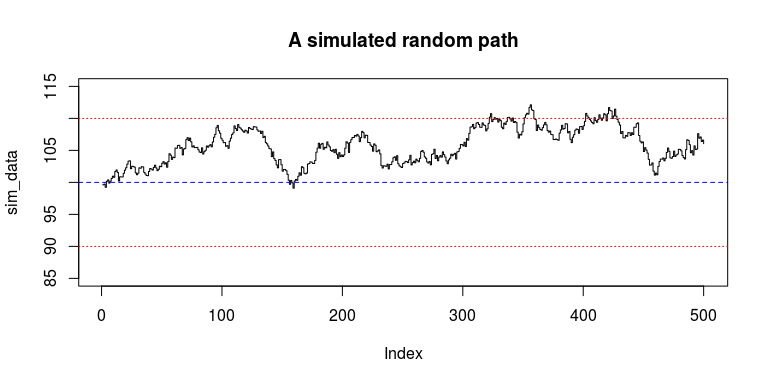
图 13-6
图 13-6 显示了随机路径以及 10%的边界。很明显,在 300~500 之间有很多值超出了
上界。
这只是一条路径,一个有效的模拟要求我们根据需要运行尽可能多的次数,以得到具
有统计意义的结果。以下函数对随机路径生成器进行参数化,并返回我们感兴趣的统计指
标。注意,signal 表示路径上是否存在超出边界的点:
simulate <- function(i, p = 100, n = 10000,
r = 0, sigma = 0.0005, margin = 0.1) {
ps <- p * cumprod(1 + rnorm(n, r, sigma))
list(id = i,
first = ps[[1]],
high = max(ps),
low = min(ps),
last = ps[[n]],
signal = any(ps > p * (1 + margin) | ps < p * (1 - margin)))
}
接着可以运行一次,查看模拟结果:
simulate(1)
$id
[1] 1
$first
[1] 100.0039
$high
[1] 101.4578
$low
[1] 94.15108
$last
[1] 96.13973
$signal
[1] FALSE
要进行模拟,就需要运行该函数很多次。在实际运用中,我们可能需要运行至少数百
万次,这可能需要耗费大量的时间。接下来,测试一下运行一万次迭代的模拟需要耗费多
长时间:
system.time(res <- lapply(1:10000, simulate))
## user system elapsed
## 8.768 0.000 8.768
模拟结束后,我们将所有结果存入一个数据表中:
library(data.table)
res_table <- rbindlist(res)
head(res_table)
## id first high low last signal
## 1: 1 100.03526 100.7157 93.80330 100.55324 FALSE
## 2: 2 100.03014 104.7150 98.85049 101.97831 FALSE
## 3: 3 99.99356 104.9834 95.28500 95.59243 FALSE
## 4: 4 99.93058 103.4315 96.10691 97.22223 FALSE
## 5: 5 99.99785 100.6041 94.12958 95.97975 FALSE
## 6: 6 100.03235 102.1770 94.65729 96.49873 FALSE
还可以计算 signal == TRUE 的实现概率:
res_table[, sum(signal) /.N]
## [1] 0.0881
更进一步地,如果实际问题需要运行数百万次呢?在这种情况下,一些研究人员可能
就会选择具有更高性能的 C 和 C++ 等灵活高效的编程语言来实现。它们都是实现算法的强
大工具,但是需要更多的精力来处理编译器、链接器和数据的输入输出。
我们注意到,在之前的模拟中,每一次迭代都是相互独立的,所以使用并行计算进行
实现会更好。
由于不同的操作系统有不同的进程和线程模型的实现,某些功能在 Linux 和 MacOS 操
作系统下可用,而在 Windows 操作系统下不可用。因此,我们会更详细地介绍如何
在 Windows 操作系统下进行并行计算。
1.在 Windows 上使用并行计算
在 Windows 操作系统下进行并行计算,需要创建一个包含多个 R 会话的本地集群:
library(parallel)
cl <- makeCluster(detectCores( ) )
detectCores( ) 函数返回你的计算机配备的核数目。创建超过该数量的节点集群
是可行的,但这并没有什么好处,因为你的计算机无法同时执行这么多任务。
然后,调用 parLapply( ) 函数,即并行版本的 lapply( ) 函数:
system.time(res <- parLapply(cl, 1:10000, simulate))
## user system elapsed
## 0.024 0.008 3.772
我们注意到,耗时比原来减少了一半以上。当不再需要这个集群时,可以调用 stop
Cluster( ) 终止刚才创建的 R 会话:
stopCluster(cl)
当调用 parLapply( ) 时,它会自动为每个集群节点安排任务。更具体地说,所有
的集群节点各自同时运行输入为 1:10000 的 simulate( ) 函数,所有的计算并行完成。
最后,收集所有结果,我们便可以得到一个像 lapply( ) 函数返回结果那样的列表:
length(res)
## [1] 10000
res_table <- rbindlist(res)
res_table[, sum(signal) /.N]
## [1] 0.0889
并行代码看起来很简单,因为 simulate( ) 函数是自给自足的,不依赖用户定义的外部
变量或数据集。如果我们并行运行一个函数,该函数引用了主会话(创建了集群的当前会
话)中的变量,那么程序就会报错找不到该变量:
cl <- makeCluster(detectCores( ) )
n <- 1
parLapply(cl, 1:3, function(x) x + n)
## Error in checkForRemoteErrors(val): 3 nodes produced errors; first
error: object 'n' not found
stopCluster(cl)
所有的节点都出现了错误,因为它们都启动了新的 R 会话,环境里没有用户定义的变
量。为了使集群节点获得所需的变量值,我们需要将其导入所有节点。
下面的例子演示了如何完成这一步。假设我们有一个包含一些数值的数据框,然后进
行随机抽样:
n <- 100
data <- data.frame(id = 1:n, x = rnorm(n), y = rnorm(n))
take_sample <- function(n) {
data[sample(seq_ _len(nrow(data)),
size = n, replace = FALSE), ]
}
如果想并行抽样,就要求所有节点必须共享数据框和函数。为此,我们可以使用 cluster
EvalQ( ) 函数在每一个集群节点上计算表达式。首先,创建一个集群:
cl <- makeCluster(detectCores( ) )
Sys.getpid( ) 函数返回当前 R 会话的进程 ID。由于集群中有 4 个节点,每个节点
都是具有唯一进程 ID 的 R 会话。我们可以调用函数 clusterEvalQ( ),并将函
数 Sys.getpid( ) 作为一个参数,查看每个节点的进程 ID:
clusterEvalQ(cl, Sys.getpid( ) )
## [[1]]
## [1] 20714
##
## [[2]]
## [1] 20723
##
## [[3]]
## [1] 20732
##
## [[4]]
## [1] 20741
如果想要查看每个节点在全局环境中的变量,可以调用 ls( ),就像在自己的工作环
境中调用一样:
clusterEvalQ(cl, ls( ) )
## [[1]]
## character(0)
##
## [[2]]
## character(0)
##
## [[3]]
## character(0)
##
## [[4]]
## character(0)
正如前面提到的,默认情况下,所有集群节点在初始化时,全局环境均为空。要将
data 和 take_sample( ) 导入每个节点,可以调用 clusterExport( ):
clusterExport(cl, c("data", "take_sample"))
clusterEvalQ(cl, ls( ) )
## [[1]]
## [1] "data" "take_sample"
##
## [[2]]
## [1] "data" "take_sample"
##
## [[3]]
## [1] "data" "take_sample"
##
## [[4]]
## [1] "data" "take_sample"
此时,如你所见,所有的节点都包含 data 和 take_sample。现在就可以对每个节
点调用 take_sample( ) :
clusterEvalQ(cl, take_ _sample(2))
## [[1]]
## id x y
## 88 88 0.6519981 1.43142886
## 80 80 0.7985715 -0.04409101
##
## [[2]]
## id x y
## 65 65 -0.4705287 -1.0859630
## 35 35 0.6240227 -0.3634574
##
## [[3]]
## id x y
## 75 75 0.3994768 -0.1489621
## 8 8 1.4234844 1.8903637
##
## [[4]]
## id x y
## 77 77 0.4458477 1.420187
## 9 9 0.3943990 -0.196291
或者,我们可以使用 clusterCall( ) 和 <<- 在每个节点中创建全局变量,而 <-
只能在函数中创建局部变量:
invisible(clusterCall(cl, function() {
local_var <- 10
global_var <<- 100
}))
clusterEvalQ(cl, ls())
## [[1]]
## [1] "data" "global_var" "take_sample"
##
## [[2]]
## [1] "data" "global_var" "take_sample"
##
## [[3]]
## [1] "data" "global_var" "take_sample"
##
## [[4]]
## [1] "data" "global_var" "take_sample"
注意到,clusterCall( ) 给出了每个节点的返回值。在上面这段代码中,我们调
用函数 invisible( ) 隐藏节点的返回值。
由于每个集群节点启动时都是原始的状态,因此,只加载了基本的扩展包。在每个节点
处加载特定的扩展包,也可以调用函数 clusterEvalQ( ) 进行加载。以下代码在每个节
点加载了 data.table 扩展包,以便 parLapply( ) 在每个节点都可以运行包里的函数:
clusterExport(cl, "simulate")
invisible(clusterEvalQ(cl, {
library(data.table)
}))
res <- parLapply(cl, 1:3, function(i) {
res_table <- rbindlist(lapply(1:1000, simulate))
res_table[, id := NULL]
summary(res_table)
})
结果返回一个数据汇总列表:
res
## [[1]]
## first high low
## Min. : 99.86 Min. : 99.95 Min. : 84.39
## 1st Qu.: 99.97 1st Qu.:101.44 1st Qu.: 94.20
## Median :100.00 Median :103.32 Median : 96.60
## Mean :100.00 Mean :103.95 Mean : 96.04
## 3rd Qu.:100.03 3rd Qu.:105.63 3rd Qu.: 98.40
## Max. :100.17 Max. :121.00 Max. :100.06
## last signal
## Min. : 84.99 Mode :logical
## 1st Qu.: 96.53 FALSE:911
## Median : 99.99 TRUE :89
## Mean : 99.92 NA's :0
## 3rd Qu.:103.11
## Max. :119.66
##
## [[2]]
## first high low
## Min. : 99.81 Min. : 99.86 Min. : 83.67
## 1st Qu.: 99.96 1st Qu.:101.48 1st Qu.: 94.32
## Median :100.00 Median :103.14 Median : 96.42
## Mean :100.00 Mean :103.91 Mean : 96.05
## 3rd Qu.:100.04 3rd Qu.:105.76 3rd Qu.: 98.48
## Max. :100.16 Max. :119.80 Max. :100.12
## last signal
## Min. : 85.81 Mode :logical
## 1st Qu.: 96.34 FALSE:914
## Median : 99.69 TRUE :86
## Mean : 99.87 NA's :0
## 3rd Qu.:103.31
## Max. :119.39
##
## [[3]]
## first high low
## Min. : 99.84 Min. : 99.88 Min. : 85.88
## 1st Qu.: 99.97 1st Qu.:101.61 1st Qu.: 94.26
## Median :100.00 Median :103.42 Median : 96.72
## Mean :100.00 Mean :104.05 Mean : 96.12
## 3rd Qu.:100.03 3rd Qu.:105.89 3rd Qu.: 98.35
## Max. :100.15 Max. :117.60 Max. :100.03
## last signal
## Min. : 86.05 Mode :logical
## 1st Qu.: 96.70 FALSE:920
## Median :100.16 TRUE :80
## Mean :100.04 NA's :0
## 3rd Qu.:103.24
## Max. :114.80
当不需要集群时,可以运行以下代码来移除:
stopCluster(cl)
2.在 Linux 和 MacOS 上使用并行计算
在 Linux 和 MacOS 操作系统下使用并行计算,比在 Windows 操作系统下容易很多。
我们无需手动地创建一个基于套接字的集群,mclapply( )直接将当前的 R 会话分配到
多个 R 会话中,保留所有内容,并为每个子会话安排任务,并行运行:
system.time(res <- mclapply(1:10000, simulate,
mc.cores = detectCores( ) ))
## user system elapsed
## 9.732 0.060 3.415
因此,我们无需导出变量,因为在每个分配的进程中,它们都是可以直接使用的:
mclapply(1:3, take_sample, mc.cores = detectCores( ) )
## [[1]]
## id x y
## 62 62 0.1679572 -0.5948647
##
## [[2]]
## id x y
## 56 56 1.5678983 0.08655707
## 39 39 0.1015022 -1.98006684
##
## [[3]]
## id x y
## 98 98 0.13892696 -0.1672610
## 4 4 0.07533799 -0.6346651
## 76 76 -0.57345242 -0.5234832
此外,还可以用非常灵活的方式创建并行作业。例如,创建一个生成 10 个随机数的作业:
job1 <- mcparallel(rnorm(10), "job1")
创建了一项作业之后,我们就可以调用 mccollect( ) 函数收集作业结果。这样,
该函数在作业完成之前不会返回任何结果:
mccollect(job1)
## $`20772`
## [1] 1.1295953 -0.6173255 1.2859549 -0.9442054 0.1482608
## [6] 0.4242623 0.9463755 0.6662561 0.4313663 0.6231939
还可以通过编程方式创建并运行多项作业。例如,我们创建 8 项作业,每项作业都随
机休眠一段时间,mccollect( ) 在所有作业完成休眠之前不会返回任何结果。作业是并
行运行的,因而 mccollect( ) 耗费的时间不会太长:
jobs <- lapply(1:8, function(i) {
mcparallel({
t <- rbinom(1, 5, 0.6)
Sys.sleep(t)
t
}, paste0("job", i))
})
system.time(res <- mccollect(jobs))
## user system elapsed
## 0.012 0.040 4.852
这就允许我们自定义任务调度机制。



【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】凌霞软件回馈社区,携手博客园推出1Panel与Halo联合会员
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步