11.分类与监督学习,朴素贝叶斯分类算法
1.理解分类与监督学习、聚类与无监督学习。
简述分类与聚类的联系与区别。
简述什么是监督学习与无监督学习。
答:
简述分类与聚类的联系与区别:
聚类:在没有训练的条件下把样本划分为若干类。
分类:已知存在哪些类,即对于目标数据库中存在哪些类是知道的,要做的就是将每一条记录分别属于哪一类标记出来。
区别:两者区别就是条件中有没有已知类别。
简述什么是监督学习与无监督学习:
监督学习:从大量的先前知识中来判断是什么类型。
无监督学习:把已有的数据进行分类,然后判断是什么类型。
2.朴素贝叶斯分类算法 实例
利用关于心脏病患者的临床历史数据集,建立朴素贝叶斯心脏病分类模型。
有六个分类变量(分类因子):性别,年龄、KILLP评分、饮酒、吸烟、住院天数
目标分类变量疾病:
–心梗
–不稳定性心绞痛
新的实例:–(性别=‘男’,年龄<70, KILLP=‘I',饮酒=‘是’,吸烟≈‘是”,住院天数<7)
最可能是哪个疾病?
上传手工演算过程。
|
|
性别 |
年龄 |
KILLP |
饮酒 |
吸烟 |
住院天数 |
疾病 |
|
1 |
男 |
>80 |
1 |
是 |
是 |
7-14 |
心梗 |
|
2 |
女 |
70-80 |
2 |
否 |
是 |
<7 |
心梗 |
|
3 |
女 |
70-81 |
1 |
否 |
否 |
<7 |
不稳定性心绞痛 |
|
4 |
女 |
<70 |
1 |
否 |
是 |
>14 |
心梗 |
|
5 |
男 |
70-80 |
2 |
是 |
是 |
7-14 |
心梗 |
|
6 |
女 |
>80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
7 |
男 |
70-80 |
1 |
否 |
否 |
7-14 |
心梗 |
|
8 |
女 |
70-80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
9 |
女 |
70-80 |
1 |
否 |
否 |
<7 |
心梗 |
|
10 |
男 |
<70 |
1 |
否 |
否 |
7-14 |
心梗 |
|
11 |
女 |
>80 |
3 |
否 |
是 |
<7 |
心梗 |
|
12 |
女 |
70-80 |
1 |
否 |
是 |
7-14 |
心梗 |
|
13 |
女 |
>80 |
3 |
否 |
是 |
7-14 |
不稳定性心绞痛 |
|
14 |
男 |
70-80 |
3 |
是 |
是 |
>14 |
不稳定性心绞痛 |
|
15 |
女 |
<70 |
3 |
否 |
否 |
<7 |
心梗 |
|
16 |
男 |
70-80 |
1 |
否 |
否 |
>14 |
心梗 |
|
17 |
男 |
<70 |
1 |
是 |
是 |
7-14 |
心梗 |
|
18 |
女 |
70-80 |
1 |
否 |
否 |
>14 |
心梗 |
|
19 |
男 |
70-80 |
2 |
否 |
否 |
7-14 |
心梗 |
|
20 |
女 |
<70 |
3 |
否 |
否 |
<7 |
不稳定性心绞痛
|

3.使用朴素贝叶斯模型对iris数据集进行花分类。
尝试使用3种不同类型的朴素贝叶斯:
- 高斯分布型
- 多项式型
- 伯努利型
并使用sklearn.model_selection.cross_val_score(),对各模型进行交叉验证。
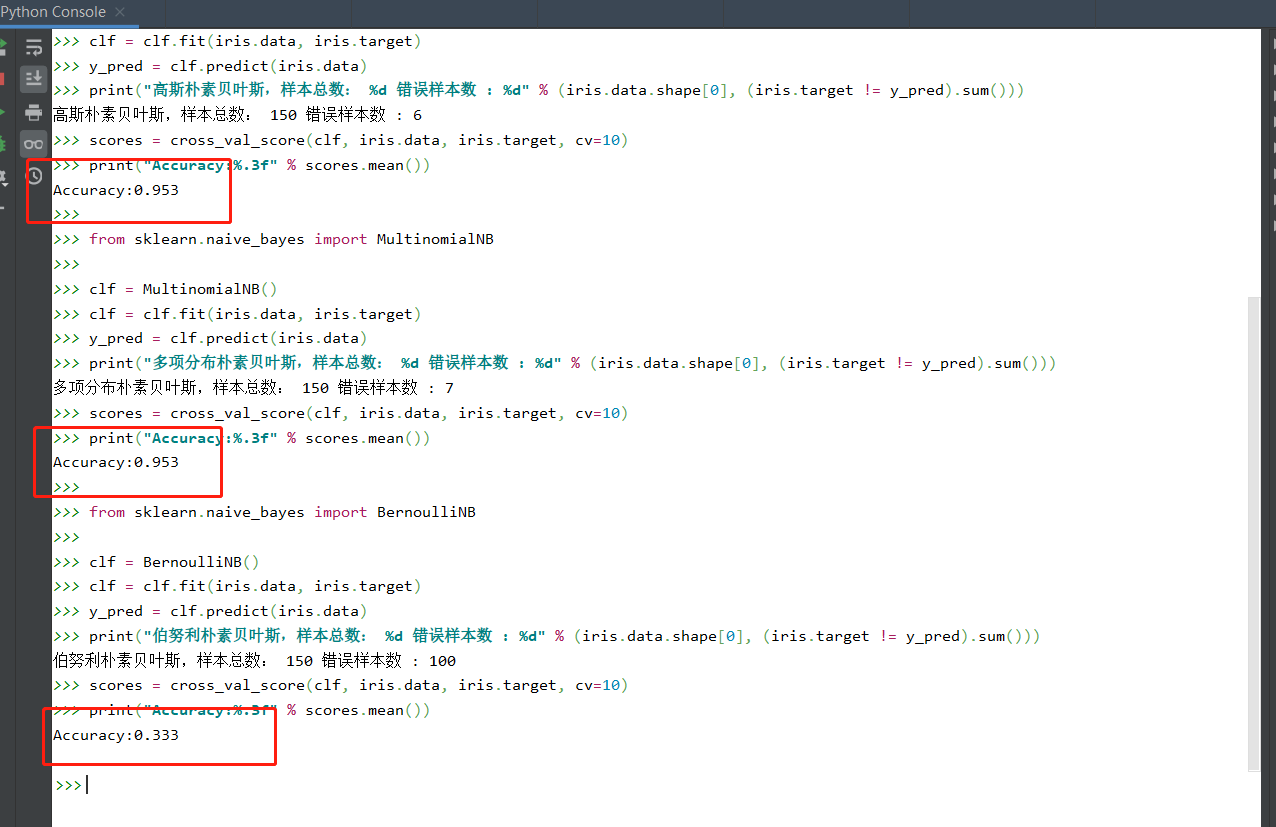
from sklearn.datasets import load_iris from sklearn.model_selection import cross_val_score iris=load_iris() from sklearn.naive_bayes import GaussianNB clf = GaussianNB() clf = clf.fit(iris.data, iris.target) y_pred=clf.predict(iris.data) print("高斯朴素贝叶斯,样本总数: %d 错误样本数 : %d" % (iris.data.shape[0],(iris.target != y_pred).sum())) scores=cross_val_score(clf,iris.data,iris.target,cv=10) print("Accuracy:%.3f"%scores.mean()) from sklearn.naive_bayes import MultinomialNB clf = MultinomialNB() clf = clf.fit(iris.data, iris.target) y_pred=clf.predict(iris.data) print("多项分布朴素贝叶斯,样本总数: %d 错误样本数 : %d" % (iris.data.shape[0],(iris.target != y_pred).sum())) scores=cross_val_score(clf,iris.data,iris.target,cv=10) print("Accuracy:%.3f"%scores.mean()) from sklearn.naive_bayes import BernoulliNB clf = BernoulliNB() clf = clf.fit(iris.data, iris.target) y_pred=clf.predict(iris.data) print("伯努利朴素贝叶斯,样本总数: %d 错误样本数 : %d" % (iris.data.shape[0],(iris.target != y_pred).sum())) scores=cross_val_score(clf,iris.data,iris.target,cv=10) print("Accuracy:%.3f"%scores.mean())