硒蛋白的生物信息学
硒蛋白的生物信息学
李思思 孟琪 金伟萍
摘要:生物信息学为硒的研究领域带来了重要的启示。过去二十年来,随着基因组资源的不断增长,计算工具的发展取得了进展,为研究硒蛋白提供了新的机会。本文综述了硒蛋白基因发现和其他硒生物学研究的生物信息学方法。最近的进展完整的硒蛋白组的可用性可以评估在整个生命树中硒半胱氨酸的使用的全球分布,以及研究硒蛋白的进化及其生物合成途径。除了基因鉴定和特性描述之外,人类硒蛋白基因的遗传变异还被用来检查不同人群对硒水平的适应能力,并估计针对基因缺失的选择性约束。在人类中,硒蛋白基因的一些突变与罕见的先天性疾病有关。然而,哺乳动物细胞中硒半胱氨酸的插入和硒蛋白合成的调控机制尚不完全清楚。组学技术为研究硒蛋白和硒半胱氨酸在细胞、组织和生物中的结合机制提供了新的可能性。
1 前言
继1973年,硒被鉴定为某些蛋白质的主要成分后,一系列的研究阐明了新发现的硒半胱氨酸合成的分子和遗传基础。已经确定硒以Sec的形式存在于硒蛋白中,而Sec残基由一个框内TGA密码子(mRNA中的UGA)编码。在大肠杆菌和动物中,发现了一个Sec‐插入tRNA (tRNA‐Sec)作为SelC基因。硒蛋白通常被标准的基因发现程序错误预测,并在基因组注释项目和蛋白质数据库中被错误注释。
2 硒蛋白基因注释
在非模式生物中硒蛋白通常被错误预测,因为标准基因注释程序只将UGA视为停止信号,而Sec - UGA密码子的正确识别需要额外的管理步骤。典型的误注包括Sec‐UGA被认为是停止的,包含Sec‐UGA的外显子被跳过,或者编码序列(CDS)开始于Sec‐UGA的下游。硒蛋白注释中的错误会随着数据库中的蛋白质序列被用来注释新的基因组而传播。支持硒蛋白候选基因正确识别和注释的主要特征是:1)存在一个正确定位的SECIS;2)硒蛋白同源物的鉴定(Sec/Sec比对);3)鉴定含有半胱氨酸(Cys)的同源物(Sec/Cys比对);4) UGA 5'和3'的蛋白编码电位序列特征。在基因组序列中寻找硒蛋白可以分为两个概念上不同的问题:寻找已知的硒蛋白和寻找新的硒蛋白。
3 寻找已知硒蛋白的基因
硒蛋白基因可以通过其与先前硒蛋白序列的同源性来识别。简单地说,一个“查询”包含秒的氨基酸序列被用来扫描“目标”DNA或RNA序列组合(如基因组、转录组或宏基因组),使用Tblastn(2)或类似的方法。Tblastn将核苷酸序列转换为所有6帧,并发现查询和目标之间氨基酸序列相似性的高得分区域。将查询中的Sec残基与靶基因中的帧内TGA匹配的结果是硒蛋白基因的潜在指标。同源性搜索不仅可以识别硒蛋白,还可以识别同一蛋白家族中的其他标准基因。这些同源物携带Cys取代活性位点上的Sec,所以我们称它们为Cys‐homolog。
4 新型硒蛋白的基因发现
用于鉴定新硒蛋白(即那些不含Sec的同源物的硒蛋白)的方法依赖于SECIS元素的鉴定。这些顺式作用的元素在不同位置的生命域之间存在差异(真核生物、古菌的3'UTR;(图1),以及RNA结构和同一性(图2),因此每个域都需要对一般方法进行专门的调整。简单地说,用专门的方法扫描DNA或RNA序列组合以找到SECIS候选序列(稍后详细介绍)。分析每个SECIS对应的区域是否存在含有TGA的开放阅读框(ORFs),然后根据它们编码硒蛋白的可能性进行评估。在基因预测领域,评估一个序列的“编码潜能”有两个主要原则:(i)从头开始寻找基因,(ii)基于同源性的匹配。在同源性匹配中,邻近SECIS元素的核苷酸序列被翻译成6个框架,并与蛋白质数据库对齐。这种方法类似于上述已知硒蛋白的检测,其中TGA与注释蛋白的Sec残基排列。然而,对于新的硒蛋白,需要考虑TGA密码子和注释蛋白的Cys残基之间的匹配。假设在硒蛋白的蛋白质数据库中已经注释了Cys‐同源物,而这些蛋白质还没有被发现。由于几乎所有的硒蛋白都有Cys‐同源物,这种方法可以发现新的硒蛋白。

图1 真核生物、古菌的3'UTR
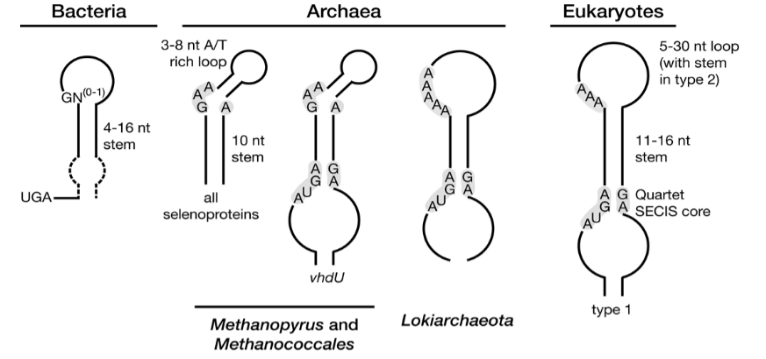
图2 真核生物、古菌的RNA结构和同一性
5 Sec机械因素
Sec的生物合成和协同翻译插入需要一个专门的遗传机制的作用。虽然对人类和其他脊椎动物必不可少,Sec不是无处不在的生命。Sec的生物合成发生在其同源tRNA上,其反密码子UCA与UGA互补。tRNA‐Sec最初被seryl‐tRNA合成酶(SerRS)与丝氨酸(Ser)氨基酰化,SerRS是Sec合成的主链。在细菌中,磷酸吡哆醛(PLP)依赖的蛋白Sec合酶(SELA)将Ser转化为Sec。在真核生物和古菌中,发生两步反应,其中Ser首先被激酶PSTK磷酸化,产生磷酸丝氨酸,然后由Sec合酶SEPSECS转化为Sec(100)(系统发育上有别于细菌的SELA)。硒的活性供体磷酸硒是由硒化物经磷酸硒合成酶SEPHS2(原核生物中的SELD)合成的[1]。SECIS二级结构促进了SECIS的协同翻译整合,真核生物需要SECIS特异性延伸因子EEFSEC,原核生物需要SECIS特异性延伸因子SELB。
6 硒基因组学的兴起
6.1真核生物硒基因组学
基因组中硒蛋白基因的研究是由序列中SECIS元素检测程序的开发启动的。第一个真核SECIS发现的计算工具是SECISearch[2]。该程序用于预测转录序列中的SECIS元素,然后对其进行分析,找到相应的含有TGA‐硒蛋白编码潜能的orf。这两项研究通过计算方法鉴定了前三种新的硒蛋白,并通过用放射性硒标记细胞证实了Sec与这些蛋白的结合。这三种蛋白质目前被称为MSRB1、SELENOT和SELENON[3]。另一项研究采用了类似的方法,重点是预测SECIS元素,然后鉴定硒蛋白ORFs[4]。由此鉴定出三种硒蛋白,其中两种是新发现的。
6.2原核生物硒基因组学
自然,类似的计算方法被应用于细菌和古菌,以解释特定区域SECIS元素的差异(图1)。细菌的SECIS (bSECIS)位于UGA编码序列的下游,其功能受茎环结构和到UGA密码子的距离的限制[5]。该程序扫描一个核苷酸序列,并检查每个UGA三联体下游潜在bSECIS的发生。基于与含cy蛋白同源性匹配的独立于SECIS的方法也已应用于细菌基因组和环境样本,产生了许多新的硒蛋白。
6.3硒蛋白基因预测的计算工具
在过去的十年中,基因组测序的快速发展使得手工鉴定硒蛋白变得不切实际。得益于以下专门用于硒基因组学的计算工具,如今可以在很少或不需要人工干预的情况下分析大量基因组:
Selenoprofiles是一个基于同源性的管道,用于预测基因组序列中已知的硒蛋白。该程序也适用于寻找含有Cys‐的同源物,以及Sec机制的蛋白质因子。该程序提供了一组默认的手动管理的配置文件,用于所有已知的硒蛋白和机械蛋白因子,并可以扩展到注释标准基因。SECISearch3是目前鉴定真核生物SECIS元素最有效、最广泛使用的方法。SECISearch3是对原始SECISearch的改进,通过引入协方差模型,提高了RNA motif查找的速度和准确性。该程序也是Seblastian的组成部分,Seblastian是一个依赖于SECIS的管道,用于鉴定真核硒蛋白。Seblastian分析SECIS元素上游区域,通过同源性找到硒蛋白基因。Seblastian既可以用于识别已知的硒蛋白(将orf与含有Sec注释的同源物匹配),也可以用于预测新的硒蛋白(将Cys‐同源物匹配)。然后,可以使用专用工具SECISaln对真核生物SECIS进行结构分析[6]。
为了找到细菌SECIS元素,使用bSECISearch方法。这个程序可以在http://genomics.unl.edu/bSECISearch/网站上找到,它扫描核苷酸序列并返回潜在的bSECIS元素及其宿主ORF。bSECIS元素是通过匹配预定义的结构模式和几个序列约束来识别的。SelGenAmic是一种专为硒蛋白基因开发的从头开始基因预测器。所以Secmarker可以用来快速扫描数千个基因组,并预测哪些基因组可能编码硒蛋白。
6.4硒蛋白数据库
由于硒研究中广泛使用的生物信息学资源历史上的不可靠性,许多专门的硒蛋白数据库应运而生。它的第二次发布扩展了Selenoprofiles的自动注释数据库,以包括更多的基因组,主要是脊椎动物(81个)。然后,数据库dbTEU收集了与微量元素相关的原核生物和真核生物的蛋白质,包括硒蛋白。接下来,recode是一个通过编码事件进行非标准翻译的基因数据库,如硒半胱氨酸和其他类型的终止密码子重新定义。最后,我们在NCBI参考序列数据库(RefSeq)中对原核生物和脊椎动物中的硒蛋白进行重新注释。
6.5硒蛋白在生命树中的分布
测序技术的兴起带来了巨大的生物多样性,科学家可以使用核苷酸序列的形式。许多研究人员分析了整个生命树的序列,以描绘硒蛋白的分类分布和其他形式的硒利用。在真核生物中硒蛋白的分布概述了一个高度动态的进化史(图3)。许多硒蛋白家族在单细胞真核生物和脊椎动物之间共享,特别是硒磷酸酶合成酶(SPS)、谷胱甘肽过氧化物酶(GPX)、硫氧还蛋白还原酶(TR)和甲硫氨酸- R -亚氧基还原酶(MSRB)等,表明大多数真核硒蛋白的起源较早[7]。
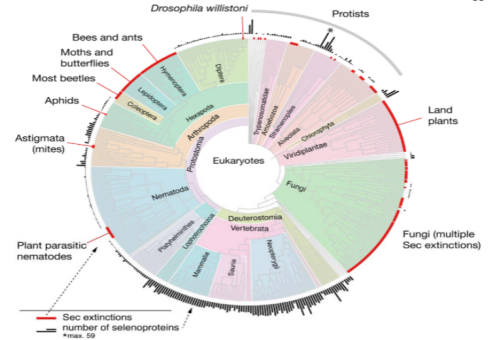
图3 真核生物中硒蛋白的分布
硒蛋白广泛存在于后生动物中。重建的原始脊椎动物硒蛋白组由28个基因组成,其进化历史被详细描述。这25种人类硒蛋白在哺乳动物中是严格保守的(小鼠有24种,GPX6中含有Cys而不是Sec)。鱼类基因组编码多达38个硒蛋白基因,是多谱系特异性基因复制的产物。三个硒蛋白家族是脊椎动物特有的:SELENOI (EPT1或SelI), SELENOV (SelV)和SELENOE (Fep15,只在鱼类中发现)。可以作为不同进化尺度上硒利用的定量标记:它在血浆中的浓度反映了小鼠和人类硒的可利用性,其编码的Sec残基数量与脊椎动物和其他动物的硒蛋白组大小相关。值得注意的是,与坚硬骨鱼相比,SELENOP在哺乳动物中的Sec残基更少。再次支持了水环境在塑造硒蛋白进化中的作用。
对全序列基因组的细菌分析表明,硒蛋白被20 - 25%的细菌使用,在不同的谱系中广泛分散分布。目前已在细菌中鉴定出约50个硒蛋白家族(103个),其中只有少数家族存在于真核生物中:硒磷酸酶合成酶(SelD或SPS2)、谷胱甘肽过氧化物酶(GPX)、类去碘酶(DI)、甲硫氨酸- S -亚砜还原酶(MsrA)、烷基氢过氧化物还原酶C (AhpC)和自由基SAM结构域蛋白(RSAM)。尽管大多数细菌(~75%)不使用Sec,但大多数分支含有含有硒蛋白的成员。富含硒蛋白的细菌被鉴定为3个门:Deltaproteobacteria, Clostridia和Synergistetes,迄今为止最大的硒蛋白组被鉴定为Syntrophobacter fumaroxidans (Deltaproteobacteria),含有39个硒蛋白。然而,大多数细菌硒蛋白是通过生物信息学分析鉴定出来的,其功能只能通过序列同源性来推断。进一步的研究将是必要的,以更好地了解硒在原核生物中的生物学。此外,很有可能许多硒蛋白还没有被鉴定出来,需要更有效和准确的工具来鉴定分析大量正在测序的基因组和宏基因组。
核糖体图谱(Ribo‐seq)已成为研究硒蛋白合成机制的重要工具。这种相对较新的技术是基于高通量测序核糖体保护片段(RPF),大约30个核苷酸长序列大致对应于单个核糖体覆盖的序列。对RPFs的分析揭示了转录组中哪些区域处于主动翻译状态。定量的RPFs提供了一个测量核糖体丰度与密码子分辨率。Ribo‐seq实验对于研究翻译的许多方面都很有用。特别是,对于硒蛋白,它允许量化UGA读通,作为Sec掺入效率的代理。UGA编码的一个特别有趣的例子是SelenoP基因,它包含两个SECIS元素和多个UGA密码子。Ribo‐seq被用于研究两种SECIS在小鼠中的作用,最近用于研究太平洋牡蛎中含有46个UGAs‐SelenoP的翻译。
人群遗传学研究人群中硒蛋白基因的遗传变异是研究人类适应、选择和疾病的有力工具。将这些基因与小鼠的敲除表型进行比较。虽然在人类和小鼠之间发现了良好的对应关系,但在一些基因中发现了显著的差异,如GPx4的耐量差异,其中小鼠会更敏感。令人惊讶的是,碘甲状腺原氨酸脱碘酶似乎不耐受人类的LoF变异,而在小鼠中删除它们只产生了轻微的表型。
硒蛋白是生物信息学的早期应用,在人类基因组发表之前。核苷酸序列中硒蛋白的预测仍然具有挑战性,这主要是由于可用的基因组和转录组的数量不断增加。自动化程序缓解了这一问题,现在可以进行大规模的调查,提供了生命之树中Sec使用情况的详细地图。硒蛋白的研究也受益于新的测序技术,对人类基因组和外显子的测序,现在已经涵盖了数十万个个体,以及对疾病患者的测序,有望为硒蛋白的分子机制以及硒在健康和疾病中的作用提供新的见解。
参考文献
[1].Gladyshev VN. Eukaryotic Selenoproteomes. In: Selenium. Cham, Springer International Publishing, 2016, pp. 127–139.
[2]. Kryukov G V., Kryukov VM, and Gladyshev VN. New mammalian selenocysteine‐containing proteins identified with an algorithm that searches for selenocysteine insertion sequence elements. J Biol Chem 274: 33888–33897, 1999.
[3]. Gladyshev VN, Arnér ES, Berry MJ, Brigelius‐Flohé R, Bruford EA, Burk RF, Carlson BA, Castellano S, Chavatte L, Conrad M, Copeland PR, Diamond AM, Driscoll DM, Ferreiro A, Flohé L, Green FR, Guigó R, Handy DE, Hatfield DL, Hesketh J, Hoffmann PR, Holmgren A, Hondal RJ, Howard MT, Huang K, Kim H‐Y, Kim IY, Köhrle J, Krol A, Kryukov G V, Lee BJ,li BC, Lei XG, Liu Q, Lescure A, Lobanov A V, Loscalzo J, Maiorino M, Mariotti M, Sandeep Prabhu K, Rayman MP, Rozovsky S, Salinas G, Schmidt EE, Schomburg L, Schweizer U, SimonovićJ Biol Chem 291: 24036-24040, 2016。
[4]. Martin‐Romero FJ, Kryukov G V., Lobanov A V., Carlson BA, Lee BJ, Gladyshev VN, and Hatfield DL. Selenium metabolism in Drosophila. Selenoproteins, selenoprotein mRNA expression, fertility, and mortality. J Biol Chem 276: 29798–804, 2001.
[5]. Chen GFT, Fang L, and Inouye M. Effect of the relative position of the UGA codon to
the unique secondary structure in the fdhF mRNA on its decoding by selenocysteinyl tRNA in Escherichia coli. J Biol Chem 268: 23128–31, 1993.
[6]. Chapple CE, Guigó R, and Krol A. SECISaln, a web‐based tool for the creation of structure‐based alignments of eukaryotic SECIS elements. Bioinformatics 25: 674–5,2009.
[7]. Gladyshev VN. Eukaryotic Selenoproteomes. In: Selenium. Cham, Springer International Publishing, 2016, pp. 127–139.
内容分配:
李思思:框架构思、文本整理撰写
孟琪:搜集整理资料、文本格式排版
金伟萍:搜集整理资料、文本格式排版





【推荐】国内首个AI IDE,深度理解中文开发场景,立即下载体验Trae
【推荐】编程新体验,更懂你的AI,立即体验豆包MarsCode编程助手
【推荐】抖音旗下AI助手豆包,你的智能百科全书,全免费不限次数
【推荐】轻量又高性能的 SSH 工具 IShell:AI 加持,快人一步
· winform 绘制太阳,地球,月球 运作规律
· AI与.NET技术实操系列(五):向量存储与相似性搜索在 .NET 中的实现
· 超详细:普通电脑也行Windows部署deepseek R1训练数据并当服务器共享给他人
· 【硬核科普】Trae如何「偷看」你的代码?零基础破解AI编程运行原理
· 上周热点回顾(3.3-3.9)